आदर्श फाइलोजेनी: Difference between revisions
mNo edit summary |
mNo edit summary |
||
| Line 1: | Line 1: | ||
उत्तम जैविक विकास एक शब्द है जिसका उपयोग [[कम्प्यूटेशनल फाइलोजेनेटिक्स|गणनीय वंशावली]] में एक [[ वंशावली वृक्ष |वंशावली वृक्ष]] को निरूपित करने के लिए किया जाता है जिसमें सभी वर्ण [[ होमप्लासी |समाकृति या समान बनावट]] के बिना वृक्ष के नीचे विकसित होते हैं और उनकी आंतरिक ग्रंथि को वर्गीकरण किया जा सकता है। इसलिए विशिष्ट गुण अभिसारी विकास पर निर्भर नहीं करते है, और उनकी [[सादृश्य (जीव विज्ञान)|समान (जीव विज्ञान)]] संरचना भी नहीं है। सांख्यिकीय रूप से, इसे | उत्तम जैविक विकास एक शब्द है जिसका उपयोग [[कम्प्यूटेशनल फाइलोजेनेटिक्स|गणनीय वंशावली]] में एक [[ वंशावली वृक्ष |वंशावली वृक्ष]] को निरूपित करने के लिए किया जाता है जिसमें सभी वर्ण [[ होमप्लासी |समाकृति या समान बनावट]] के बिना वृक्ष के नीचे विकसित होते हैं और उनकी आंतरिक ग्रंथि को वर्गीकरण किया जा सकता है। इसलिए विशिष्ट गुण अभिसारी विकास पर निर्भर नहीं करते है, और उनकी [[सादृश्य (जीव विज्ञान)|समान (जीव विज्ञान)]] संरचना भी नहीं है। सांख्यिकीय रूप से, इसे प्रजनक के रूप में प्रदर्शित किया जा सकता है जिसमें सभी विशिष्ट गुणों की स्थिति 0 होती है जहां 0 उस विशिष्ट गुण की कमी का वर्णन करता है। इनमें से प्रत्येक विशिष्ट गुण 0 से 1 में ठीक एक बार बदलती है और कभी भी 0 की स्थिति पर नहीं लौटती है। यह दुर्लभ है कि वास्तविक डेटा उत्तम जैविक विकास की अवधारणा का पालन करता है।<ref>{{cite web |last=Fernandez-Baca |first=David | name-list-style = vanc |title= बिल्कुल सही फाइलोजेनी समस्या|url=http://www.cs.iastate.edu/~fernande/PAPERS/survey.pdf|publisher=Kluwer Academic Publishers|access-date=30 September 2012}}</ref><ref>{{cite web| vauthors = Nakhleh L, Ringe D, Warnow T |title=Perfect Phylogenetic Networks: A New Methodology for Reconstructing the Evolutionary History of Natural Languages|url=http://www.cs.rice.edu/~nakhleh/Papers/81.2nakhleh.pdf|access-date=1 October 2012 }}</ref> | ||
Revision as of 09:39, 4 April 2023
उत्तम जैविक विकास एक शब्द है जिसका उपयोग गणनीय वंशावली में एक वंशावली वृक्ष को निरूपित करने के लिए किया जाता है जिसमें सभी वर्ण समाकृति या समान बनावट के बिना वृक्ष के नीचे विकसित होते हैं और उनकी आंतरिक ग्रंथि को वर्गीकरण किया जा सकता है। इसलिए विशिष्ट गुण अभिसारी विकास पर निर्भर नहीं करते है, और उनकी समान (जीव विज्ञान) संरचना भी नहीं है। सांख्यिकीय रूप से, इसे प्रजनक के रूप में प्रदर्शित किया जा सकता है जिसमें सभी विशिष्ट गुणों की स्थिति 0 होती है जहां 0 उस विशिष्ट गुण की कमी का वर्णन करता है। इनमें से प्रत्येक विशिष्ट गुण 0 से 1 में ठीक एक बार बदलती है और कभी भी 0 की स्थिति पर नहीं लौटती है। यह दुर्लभ है कि वास्तविक डेटा उत्तम जैविक विकास की अवधारणा का पालन करता है।[1][2]
बिल्डिंग
सामान्य तौर पर दो अलग-अलग डेटा प्रकार होते हैं जिनका उपयोग वंशावली वृक्ष के निर्माण में किया जाता है। दूरी-आधारित संगणनाओं में प्रजातियों के बीच की दूरी और संबंधित वृक्ष के किनारों की लंबाई के बीच संबंधों का विश्लेषण करके एक वंशावली वृक्ष बनाया जाता है। एक स्वाभाविक आधारित दृष्टिकोण का उपयोग करते हुए प्रजातियों में स्वाभाविक स्थितियों को एक निविष्ट के रूप में नियोजित किया जाता है ताकि सबसे सही वंशावली वृक्ष को खोजने का प्रयास किया जा सके।[3][4]
एक संपूर्ण वंशावली वृक्ष के सांख्यिकीय अंशो को सबसे अच्छा इस प्रकार वर्णित किया जा सकता है:[3]
n x m कैरेक्टर स्टेट मैट्रिक्सM के लिए एक आदर्श फाइलोजेनी एक जड़ वाला वृक्ष T है जिसमें n पत्तियां संतोषजनक हैं:
मैं। M की प्रत्येक पंक्ति T के बिल्कुल एक पत्ते को वर्गीकरण करती है
द्वितीय। M का प्रत्येक स्तंभ T के ठीक एक किनारे को वर्गीकरण करता है
तृतीय। T के प्रत्येक आंतरिक किनारे को M के कम से कम एक स्तंभ द्वारा वर्गीकरण किया गया है
iv. रूट से लीफ v तक अद्वितीय पथ के साथ किनारों से जुड़े वर्ण v के वर्ण वेक्टर को सटीक रूप से निर्दिष्ट करते हैं, अर्थात वर्ण वेक्टर में पथ किनारों से संबंधित वर्णों के अनुरूप सभी स्तंभों में 1 प्रविष्टि होती है और अन्यथा 0 प्रविष्टि होती है।</ ब्लॉककोट> यह ध्यान देने योग्य है कि वास्तविक वंशावली डेटा मिलना बहुत दुर्लभ है जो यहां विस्तृत अवधारणाओं और सीमाओं का पालन करता है। इसलिए, अक्सर ऐसा होता है कि शोधकर्ताओं को वृक्ष ों को विकसित करके समझौता करने के लिए मजबूर किया जाता है जो केवल होमोप्लासी को कम करने की कोशिश करते हैं, संगत वर्णों का एक अधिकतम-कार्डिनैलिटी सेट ढूंढते हैं, या फ़िलेजनी का निर्माण करते हैं जो पात्रों द्वारा निहित विभाजनों से यथासंभव निकटता से मेल खाते हैं।उदाहरण
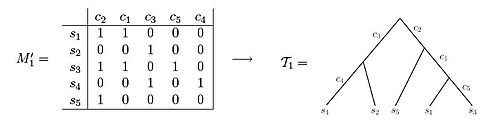
ये दोनों डेटा सेट कैरेक्टर स्टेट मैट्रिक्स (गणित) के उदाहरण दिखाते हैं। मैट्रिक्स एम' का उपयोग करना1 कोई यह देखने में सक्षम है कि परिणामी वंशावली वृक्ष को इस तरह बनाया जा सकता है कि प्रत्येक वर्ण वृक्ष के ठीक एक किनारे को वर्गीकरण करता है। इसके विपरीत, मैट्रिक्स M' का प्रेक्षण करते समय2, कोई भी देख सकता है कि वंशावली वृक्ष को स्थापित करने का कोई तरीका नहीं है जैसे कि प्रत्येक वर्ण केवल एक किनारे की लंबाई को वर्गीकरण करता है।[3]यदि नमूने अध्ययन के तहत कोशिकाओं की आबादी के वैरिएंट एलील फ़्रीक्वेंसी (VAF) डेटा से आते हैं, तो वर्ण मैट्रिक्स में प्रविष्टियाँ म्यूटेशन की आवृत्तियाँ होती हैं, और 0 और 1 के बीच का मान लेती हैं। अर्थात्, यदि जीनोम में एक स्थिति का प्रतिनिधित्व करता है, फिर संबंधित प्रविष्टि और नमूना नमूने में जीनोम की आवृत्तियों को धारण करेगा स्थिति में उत्परिवर्तन के साथ .[5][6][7][8][9]
- Character state matrices
उपयोग
उत्तम जैविक विकास एक सैद्धांतिक ढांचा है जिसका उपयोग अधिक व्यावहारिक तरीकों में भी किया जा सकता है। ऐसा ही एक उदाहरण है इंकंप्लीट डायरेक्टेड उत्तम जैविक विकास । इस अवधारणा में वास्तविक, और इसलिए अधूरे और अपूर्ण, डेटासेट के साथ सही फाइलोजेनी का उपयोग करना शामिल है। इस तरह की विधि विकासवादी समानता निर्धारित करने के लिए रेट्रोट्रांसपोज़न का उपयोग करती है। ये लघु अंतर्विभाजित तत्व कई जीनोम में मौजूद हैं और उनके फ़्लैंकिंग अनुक्रमों द्वारा पहचाने जा सकते हैं। साइन विभिन्न प्रजातियों में कुछ लक्षणों की आनुवंशिकता के बारे में जानकारी प्रदान करते हैं। दुर्भाग्य से, यदि कोई SINE गुम है, तो यह जानना मुश्किल है कि क्या वे SINE विलोपन से पहले मौजूद थे। सही फाइलोजेनी डेटा से प्राप्त एल्गोरिदम का उपयोग करके हम इन सीमाओं के बावजूद एक वंशावली पेड़ के पुनर्निर्माण का प्रयास करने में सक्षम हैं।[10]
इंटरनेशनल हाप मैप प्रोजेक्ट प्रोजेक्ट के निर्माण में उत्तम जैविक विकास का भी उपयोग किया जाता है। सही फाइलोजेनी में वर्णित अवधारणाओं और एल्गोरिदम का उपयोग करके लापता और अनुपलब्ध हैप्लोटाइप डेटा के बारे में जानकारी निर्धारित कर सकते हैं।[11] यह मानते हुए कि जीनोटाइप मैपिंग से उत्पन्न होने वाले हैप्लोटाइप्स का सेट मेल खाता है और सही फाइलोजेनी की अवधारणा का पालन करता है (साथ ही साथ अन्य धारणाएं जैसे कि सही मेंडेलियन इनहेरिटेंस और तथ्य यह है कि प्रति एसएनपी केवल एक उत्परिवर्तन है), एक अनुमान लगाने में सक्षम है लापता हैप्लोटाइप डेटा।[12][13][14] [15]
पीपीएम के तहत शोर वाले वीएएफ डेटा से फाइलोजेनी का उल्लेख करना एक कठिन समस्या है।[5]अधिकांश अनुमान उपकरणों में अनुमान लगाने के लिए कम्प्यूटेशनल रूप से ट्रैक्टेबल बनाने के लिए कुछ अनुमानी कदम शामिल हैं। उपकरण के उदाहरण जो शोर वाले VAF डेटा से फाइलोजेनी का अनुमान लगाते हैं, उनमें AncesTree, Canopy, CITUP, EXACT और PhyloWGS शामिल हैं।[5][6][7][8][9]विशेष रूप से, EXACT छोटे आकार की समस्याओं के लिए सभी संभावित वृक्ष ों पर पश्च संभाव्यता की गणना करने के लिए GPU का उपयोग करके सटीक अनुमान लगाता है। संलग्न उपकरणों के साथ पीपीएम का विस्तार किया गया है।[16][17] उदाहरण के लिए, MEDICC, TuMult, और FISHtrees जैसे उपकरण किसी दिए गए आनुवंशिक तत्व, या प्लोइडी की प्रतियों की संख्या को बढ़ाने या घटाने दोनों की अनुमति देते हैं, इस प्रकार प्रभावी रूप से उत्परिवर्तन को हटाने की अनुमति देते हैं।[18][19][20]
बाहरी संबंध
- List of phylogenetics software
- One of several programs available for analysis and creation of phylogenetic trees
- Another such program for phylogenetic tree analysis
- Additional program for tree analysis
- A paper detailing an example of how perfect phylogeny can be utilized outside of the field of genetics, as in language association
- Github for "Algorithm for clonal tree reconstruction from multi-sample cancer sequencing data" (AncesTree)
- Github for "Accessing Intra-Tumor Heterogeneity and Tracking Longitudinal and Spatial Clonal Evolutionary History by Next-Generation Sequencing" (Canopy)
- Github for "Clonality Inference in Tumors Using Phylogeny" (CITUP)
- Github for "Exact inference under the perfect phylogeny model" (EXACT)
- Github for "Reconstructing subclonal composition and evolution from whole-genome sequencing of tumors" (PhyloWGS)
संदर्भ
- ↑ Fernandez-Baca D. "बिल्कुल सही फाइलोजेनी समस्या" (PDF). Kluwer Academic Publishers. Retrieved 30 September 2012.
- ↑ Nakhleh L, Ringe D, Warnow T. "Perfect Phylogenetic Networks: A New Methodology for Reconstructing the Evolutionary History of Natural Languages" (PDF). Retrieved 1 October 2012.
- ↑ 3.0 3.1 3.2 Uhler C. "एक संपूर्ण फाइलोजेनी ढूँढना" (PDF). Archived from the original (PDF) on 4 March 2016. Retrieved 29 September 2012.
- ↑ Nikaido M, Rooney AP, Okada N (August 1999). "Phylogenetic relationships among cetartiodactyls based on insertions of short and long interpersed elements: hippopotamuses are the closest extant relatives of whales". Proceedings of the National Academy of Sciences of the United States of America. 96 (18): 10261–6. Bibcode:1999PNAS...9610261N. doi:10.1073/pnas.96.18.10261. PMC 17876. PMID 10468596.
- ↑ 5.0 5.1 5.2 El-Kebir M, Oesper L, Acheson-Field H, Raphael BJ (June 2015). "मल्टी-सैंपल सीक्वेंसिंग डेटा से क्लोनल ट्री और ट्यूमर कंपोजीशन का पुनर्निर्माण". Bioinformatics. 31 (12): i62-70. doi:10.1093/bioinformatics/btv261. PMC 4542783. PMID 26072510.
- ↑ 6.0 6.1 Satas G, Raphael BJ (July 2017). "वृक्ष-विवश महत्व नमूनाकरण का उपयोग करते हुए ट्यूमर फाइलोजेनी अनुमान". Bioinformatics. 33 (14): i152–i160. doi:10.1093/bioinformatics/btx270. PMC 5870673. PMID 28882002.
- ↑ 7.0 7.1 Malikic S, McPherson AW, Donmez N, Sahinalp CS (May 2015). "फाइलोजेनी का उपयोग करके कई ट्यूमर नमूनों में क्लोनलिटी का अनुमान". Bioinformatics. 31 (9): 1349–56. doi:10.1093/bioinformatics/btv003. PMID 25568283.
- ↑ 8.0 8.1 Ray S, Jia B, Safavi S, van Opijnen T, Isberg R, Rosch J, Bento J (2019-08-22). "सही फाइलोजेनी मॉडल के तहत सटीक अनुमान" (in English). arXiv:1908.08623 [q-bio.QM].
- ↑ 9.0 9.1 Deshwar AG, Vembu S, Yung CK, Jang GH, Stein L, Morris Q (February 2015). "PhyloWGS: reconstructing subclonal composition and evolution from whole-genome sequencing of tumors". Genome Biology. 16 (1): 35. doi:10.1186/s13059-015-0602-8. PMC 4359439. PMID 25786235.
- ↑ Pe'er I, Pupko T, Shamir R, Sharan R. "अधूरा निर्देशित बिल्कुल सही फाइलोजेनी". Tel-Aviv University. Archived from the original on 20 October 2013. Retrieved 30 October 2012.
{{cite web}}: CS1 maint: bot: original URL status unknown (link)- ↑ Eskin E, Halperin E, Karp RM (April 2003). "सही फाइलोजेनी के माध्यम से हैप्लोटाइप संरचना का कुशल पुनर्निर्माण" (PDF). Journal of Bioinformatics and Computational Biology. University of California, Berkeley. 1 (1): 1–20. doi:10.1142/S0219720003000174. PMID 15290779. Retrieved 30 October 2012.
- ↑ Gusfield D. "हाप्लोटाइप अनुमान के लिए कम्प्यूटेशनल विधियों का अवलोकन" (PDF). University of California, Davis. Retrieved 18 November 2012.
- ↑ Ding Z, Filkov V, Gusfield D. "बिल्कुल सही फाइलोजेनी हैप्लोटाइपिंग समस्या के लिए एक रेखीय समय एल्गोरिथम". University of California, Davis. Retrieved 18 November 2012.
- ↑ Bafna V, Gusfield D, Lancia G, Yooseph S (2003). "Haplotyping as perfect phylogeny: a direct approach". Journal of Computational Biology. 10 (3–4): 323–40. doi:10.1089/10665270360688048. PMID 12935331.
- ↑ Seyalioglu H. "परफेक्ट फाइलोजेनी के रूप में हाप्लोटाइपिंग" (PDF). Archived from the original (PDF) on 30 September 2011. Retrieved 30 October 2012.
- ↑ Bonizzoni P, Carrieri AP, Della Vedova G, Trucco G (October 2014). "विवश निरंतर परिपूर्ण फाइलोजेनी के माध्यम से विकास की व्याख्या करना". BMC Genomics. 15 Suppl 6 (S6): S10. doi:10.1186/1471-2164-15-S6-S10. PMC 4240218. PMID 25572381.
- ↑ Hajirasouliha I, Raphael BJ (2014), Brown D, Morgenstern B (eds.), "Reconstructing Mutational History in Multiply Sampled Tumors Using Perfect Phylogeny Mixtures", Algorithms in Bioinformatics, Springer Berlin Heidelberg, vol. 8701, pp. 354–367, doi:10.1007/978-3-662-44753-6_27, ISBN 9783662447529
- ↑ Schwarz RF, Trinh A, Sipos B, Brenton JD, Goldman N, Markowetz F (April 2014). Beerenwinkel N (ed.). "इंट्रा-ट्यूमर विषमता का फाइलोजेनेटिक मात्रा का ठहराव". PLOS Computational Biology. 10 (4): e1003535. arXiv:1306.1685. Bibcode:2014PLSCB..10E3535S. doi:10.1371/journal.pcbi.1003535. PMC 3990475. PMID 24743184.
- ↑ Letouzé E, Allory Y, Bollet MA, Radvanyi F, Guyon F (2010). "एक ही मरीज के कई ट्यूमर नमूनों की कॉपी नंबर प्रोफाइल के विश्लेषण से ट्यूमरजेनिसिस में क्रमिक चरणों का पता चलता है". Genome Biology. 11 (7): R76. doi:10.1186/gb-2010-11-7-r76. PMC 2926787. PMID 20649963.
- ↑ Gertz EM, Chowdhury SA, Lee WJ, Wangsa D, Heselmeyer-Haddad K, Ried T, et al. (2016-06-30). "FISHtrees 3.0: Tumor Phylogenetics Using a Ploidy Probe". PLOS ONE. 11 (6): e0158569. Bibcode:2016PLoSO..1158569G. doi:10.1371/journal.pone.0158569. PMC 4928784. PMID 27362268.