आनुवंशिक दूरी: Difference between revisions
(text) |
No edit summary |
||
| (2 intermediate revisions by 2 users not shown) | |||
| Line 171: | Line 171: | ||
{{DEFAULTSORT:Genetic Distance}} | {{DEFAULTSORT:Genetic Distance}} | ||
[[Category:Articles with hatnote templates targeting a nonexistent page|Genetic Distance]] | |||
[[Category:CS1 maint]] | |||
[[Category: | [[Category:Created On 10/06/2023|Genetic Distance]] | ||
[[Category:Created On 10/06/2023]] | [[Category:Lua-based templates|Genetic Distance]] | ||
[[Category:Machine Translated Page|Genetic Distance]] | |||
[[Category:Pages with script errors|Genetic Distance]] | |||
[[Category:Templates Translated in Hindi|Genetic Distance]] | |||
[[Category:Templates Vigyan Ready|Genetic Distance]] | |||
[[Category:Templates that add a tracking category|Genetic Distance]] | |||
[[Category:Templates that generate short descriptions|Genetic Distance]] | |||
[[Category:Templates using TemplateData|Genetic Distance]] | |||
[[Category:Webarchive template wayback links|Genetic Distance]] | |||
[[Category:आनुवंशिकी की अवधारणाएँ|Genetic Distance]] | |||
[[Category:फाइलोजेनेटिक्स|Genetic Distance]] | |||
Latest revision as of 16:46, 19 June 2023
| Part of a series on |
| Genetics |
|---|
 |

आनुवंशिक दूरी प्रजातियों के बीच या एक प्रजाति के भीतर आबादी के बीच आनुवंशिकी विचलन का एक उपाय है, चाहे दूरी विकल्पी या विभेदीकरण की घात से समय को मापती है। [2] कई समान युग्मविकल्पी वाली आबादी में छोटी आनुवंशिक दूरी होती है। यह इंगित करता है कि वे निकट से संबंधित हैं और हाल ही में एक सामान्य पूर्वज हैं।
आबादी के इतिहास के पुनर्निर्माण के लिए आनुवंशिक दूरी उपयोगी है, जैसे कि अफ्रीका सिद्धांत के बाहर कई मानव विस्तार है। [3] इसका उपयोग जैव विविधता की उत्पत्ति को समझने के लिए भी किया जाता है। उदाहरण के लिए, पालतू जानवरों की विभिन्न जाति के बीच आनुवंशिक दूरी की प्रायः जांच की जाती है ताकि यह निर्धारित किया जा सके कि आनुवंशिक विविधता को बनाए रखने के लिए किन जाति को संरक्षित किया जाना चाहिए। [4]
जैविक नींव
एक जीव के सजीव में, प्रत्येक जीन एक विशिष्ट स्थान पर स्थित होता है जिसे उस जीन के लिए संस्थिति (आनुवांशिकी) कहा जाता है। इन लोकी में युग्मक भिन्नता प्रजातियों के भीतर दृश्यप्ररूपी भिन्नता का कारण बनती है (जैसे बालों का रंग, आंखों का रंग)। हालांकि, अधिकांश युग्म विकल्पी का लक्षणसमष्टि पर कोई प्रभाव नहीं पड़ता है। आबादी के भीतर उत्परिवर्तन द्वारा उत्पन्न नए युग्म विकल्पी या तो मर जाते हैं या पूरी आबादी में विस्तारित हो जाते हैं। जब एक आबादी को अलग-अलग आबादी में विभाजित किया जाता है (भौगोलिक या पारिस्थितिक कारकों द्वारा), तो विभाजन के बाद होने वाले उत्परिवर्तन केवल पृथक आबादी में उपस्थित होंगे। युग्मविकल्पी आवृति का यादृच्छिक उतार-चढ़ाव भी आबादी के बीच आनुवंशिक भेदभाव उतपन्न करता है। इस प्रक्रिया को आनुवंशिक बहाव के रूप में जाना जाता है। आबादी और कंप्यूटिंग आनुवंशिक दूरी के बीच युग्मविकल्पी आवृत्तियों के बीच अंतर की जांच करके, हम आकलन लगा सकते हैं कि कितनी देर पहले दो आबादी अलग हो गई थी। [5]
उपाय
यद्यपि आनुवंशिक दूरी को आनुवंशिक विचलन के माप के रूप में परिभाषित करना सरल है, फिर भी कई अलग-अलग सांख्यिकीय उपाय प्रस्तावित किए गए हैं। ऐसा इसलिए हुआ है क्योंकि अलग-अलग लेखकों ने अलग-अलग विकासवादी प्रतिरूप पर विचार किया। सबसे अधिक इस्तेमाल किया जाने वाला नी, [5] कवेली-स्फोर्ज़ा और एडवर्ड्स माप,[6] और रेनॉल्ड्स, वीर और कॉकरहम आनुवंशिक दूरी हैं। [7]
इस खंड के सभी सूत्रों में, और जिसके लिए दो अलग-अलग आबादी का प्रतिनिधित्व करने के लिए लोकी का अध्ययन किया गया है। मान लीजिये वें स्थान पर युग्मविकल्पी आवृत्ति का प्रतिनिधित्व करते हैं।
नेई की मानक आनुवंशिक दूरी
1972 में, मसातोशी नेई ने प्रकाशित किया जिसे नेई की मानक आनुवंशिक दूरी के रूप में जाना जाने लगा। इस दूरी का यह अच्छा गुण है कि यदि प्रति वर्ष या पीढ़ी में आनुवंशिक परिवर्तन (अमीनो अम्ल प्रतिस्थापन) की दर स्थिर रहती है तो नेई की मानक आनुवंशिक दूरी (D) विचलन समय के अनुपात में बढ़ जाती है। यह उपाय मानता है कि आनुवंशिक अंतर उत्परिवर्तन और आनुवंशिक बहाव के कारण होता है।[5]
यह दूरी जीन पहचान के अंकगणितीय माध्य के संदर्भ में भी व्यक्त की जा सकती है। मान लीजिये जनसंख्या के दो सदस्यों के लिए संभावना हो एक विशेष स्थान पर एक ही युग्मविकल्पी हो और जनसंख्या में संगत संभावना हो। इसके अतिरिक्त, मान लीजिये के एक सदस्य के लिए संभावना हो और का एक सदस्य एक ही युग्मविकल्पी है। अब मान लीजिये , और के अंकगणितीय माध्य का प्रतिनिधित्व , और क्रमशः सभी लोकी पर करते हैं । दूसरे शब्दों में,
जहाँ लोकी की जांच की कुल संख्या है। [8]
नेई की मानक दूरी को तब इस प्रकार लिखा जा सकता है [5]
कवेली-स्फोर्ज़ा तार दूरी
1967 में लुइगी लुका कवेली-स्फोर्ज़ा और ए.डब्ल्यू.एफ. एडवर्ड्स ने इस उपाय को प्रकाशित किया। यह मानता है कि अनुवांशिक मतभेद केवल अनुवांशिक बहाव के कारण उत्पन्न होते हैं। इस उपाय का एक प्रमुख लाभ यह है कि आबादी को अति क्षेत्र में दर्शाया जाता है, जिसका मापक्रम प्रति जीन प्रतिस्थापन एक इकाई है। हाइपरडिमेंशनल वृत्त में जीवा की दूरी किसके द्वारा दी जाती है [2][6]
कुछ लेखक विशेषता को खोने की कीमत पर सूत्र को सरल बनाने के लिए कारक छोड़ देते हैं।
रेनॉल्ड्स, वीर, और कॉकरहम की आनुवंशिक दूरी
1983 में, इस माप को जॉन रेनॉल्ड्स, ब्रूस वीर और सी. क्लार्क कॉकरहैम द्वारा प्रकाशित किया गया था।
यह उपाय मानता है कि अनुवांशिक भिन्नता उत्परिवर्तन के बिना अनुवांशिक बहाव से ही होती है। यह मैलेकोट की सहवंश की विधि का आकलन लगाता है जो अनुवांशिक विचलन का एक उपाय प्रदान करता है: [7]
अन्य उपाय
अलग-अलग सफलता के साथ आनुवंशिक दूरी के कई अन्य उपाय प्रस्तावित किए गए हैं।
नया DA दूरी 1983
यह दूरी मानती है कि उत्परिवर्तन और आनुवंशिक बहाव के कारण आनुवंशिक अंतर उत्पन्न होते हैं, लेकिन यह दूरी माप विशेष रूप से माइक्रोसेटेलाइट डीएनए डेटा के लिए अन्य दूरियों की तुलना में अधिक विश्वसनीय जनसंख्या वृक्ष देने के लिए जाना जाता है। [9][10]
यूक्लिडियन दूरी
गोल्डस्टीन दूरी 1995
यह विशेष रूप से माइक्रोसैटेलाइट चिह्नक के लिए विकसित किया गया था और यह चरणगत-परिवर्तन प्रतिरूप (SMM) पर आधारित है। और जनसंख्या X और Y में युग्मविकल्पी आकार के साधन हैं। [12]
नेई की न्यूनतम अनुवांशिक दूरी 1973
यह उपाय मानता है कि उत्परिवर्तन और अनुवांशिक बहाव के कारण अनुवांशिक मतभेद उत्पन्न होते हैं। [13]
रोजर की दूरी 1972
यौगिकीकरण तालिका
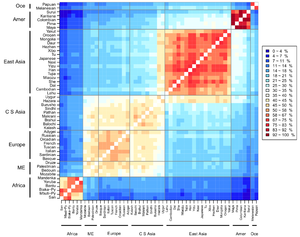
अनुवांशिक दूरी का सामान्यतः इस्तेमाल किया जाने वाला माप निर्धारण सूचकांक (FST) जो 0 और 1 के बीच भिन्न होता है। 0 का मान इंगित करता है कि दो आबादी आनुवंशिक रूप से समान हैं (दो आबादी के बीच न्यूनतम या कोई आनुवंशिक विविधता नहीं है) जबकि 1 का मान इंगित करता है कि दो आबादी आनुवंशिक रूप से भिन्न हैं (दो आबादी के बीच अधिकतम आनुवंशिक विविधता)। कोई उत्परिवर्तन नहीं माना जाता है। बड़ी आबादी, जिनके बीच बहुत अधिक प्रवासन होता है, उदाहरण के लिए, थोड़ा अलग होने की प्रवृत्ति होती है, जबकि छोटी आबादी जिनके बीच बहुत कम प्रवास होता है, उनमें बहुत अंतर होता है। FST इस भेदभाव का एक सुविधाजनक उपाय है, और परिणामस्वरूप FST और संबंधित आँकड़े जनसंख्या और विकासवादी आनुवंशिकी में सबसे व्यापक रूप से उपयोग किए जाने वाले वर्णनात्मक आंकड़ों में से हैं। लेकिन FST एक वर्णनात्मक आंकड़े और अनुवांशिक भेदभाव के उपाय से अधिक है। FST आबादी के बीच युग्मविकल्पी आवृत्ति में भिन्नता से सीधे संबंधित है और इसके विपरीत आबादी के भीतर व्यक्तियों के बीच समानता की घात है। अगर FST छोटा है, इसका मतलब है कि प्रत्येक आबादी के भीतर युग्मविकल्पी आवृत्तियां बहुत समान हैं; यदि यह बड़ा है, तो इसका अर्थ है कि युग्मविकल्पी आवृति बहुत अलग हैं।
सॉफ्टवेयर
- पीएचवाईएलआईपी जेंडरिस्ट उपयोग करता है
- नेई की मानक आनुवंशिक दूरी 1972
- कवेली-स्फोर्ज़ा और एडवर्ड्स 1967
- रेनॉल्ड्स, वीर, और कॉकरहैम 1983
- टीएफपीजीए
- नेई की मानक आनुवंशिक दूरी (मूल और निष्पक्ष)
- नेई की न्यूनतम आनुवंशिक दूरी (मूल और निष्पक्ष)
- राइट्स (1978) रोजर्स (1972) की दूरी का संशोधन
- रेनॉल्ड्स, वीर, और कॉकरहैम 1983
- जीडीए
- पॉपजीन
- पीओपीटीआरई2 तकज़ाकि, नेई, और तमूरा (2010, 2014)
- सामान्यतः इस्तेमाल की जाने वाली आनुवंशिक दूरी और जीन विविधता विश्लेषण
- डीआईएसपीएएन
- नेई की मानक आनुवंशिक दूरी 1972
- नेई का DA आबादी के बीच की दूरी 1983
यह भी देखें
संदर्भ
- ↑ Cavalli-Sforza, L.L., Menozzi, P. & Piazza, A. (1994). The History and Geography of Human Genes. New Jersey: Princeton University Press.
- ↑ 2.0 2.1 2.2 Nei, M. (1987). "Chapter 9". आणविक विकासवादी आनुवंशिकी. New York: Columbia University Press.
- ↑ Ramachandran S, Deshpande O, Roseman CC, Rosenberg NA, Feldman MW, Cavalli-Sforza LL (November 2005). "अफ्रीका में उत्पन्न होने वाले क्रमिक संस्थापक प्रभाव के लिए मानव आबादी में आनुवंशिक और भौगोलिक दूरी के संबंध से समर्थन". Proc Natl Acad Sci U S A. 102 (44): 15942–7. Bibcode:2005PNAS..10215942R. doi:10.1073/pnas.0507611102. PMC 1276087. PMID 16243969.
- ↑ Ruane J (1999). "पशु आनुवंशिक संसाधनों के संरक्षण में आनुवंशिक दूरी के अध्ययन के मूल्य की एक महत्वपूर्ण समीक्षा". Journal of Animal Breeding and Genetics. 116 (5): 317–323. doi:10.1046/j.1439-0388.1999.00205.x.
- ↑ 5.0 5.1 5.2 5.3 Nei, M. (1972). "आबादी के बीच आनुवंशिक दूरी". Am. Nat. 106 (949): 283–292. doi:10.1086/282771. S2CID 55212907.
- ↑ 6.0 6.1 L.L. Cavalli-Sforza; A.W.F. Edwards (1967). "Phylogenetic Analysis – Models and Estimation Procedures". The American Journal of Human Genetics. 19 (3 Part I (May)): 233–257. PMC 1706274. PMID 6026583.
- ↑ 7.0 7.1 John Reynolds; B.S. Weir; C. Clark Cockerham (November 1983). "Estimation of the coancestry coefficient: Basis for a short-term genetic distance". Genetics. 105 (3): 767–779. doi:10.1093/genetics/105.3.767. PMC 1202185. PMID 17246175.
- ↑ Nei, M. (1987) Genetic distance and molecular phylogeny. In: Population Genetics and Fishery Management (N. Ryman and F. Utter, eds.), University of Washington Press, Seattle, WA, pp. 193–223.
- ↑ Nei M., Tajima F., Tateno Y. (1983). "आणविक डेटा से अनुमानित फ़ाइलोजेनेटिक पेड़ों की सटीकता। द्वितीय। जीन आवृत्ति डेटा". J. Mol. Evol. 19 (2): 153–170. doi:10.1007/bf02300753. PMID 6571220. S2CID 19567426.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Takezaki N. (1996). "माइक्रोसैटेलाइट डीएनए से जेनेटिक दूरियां और फाइलोजेनेटिक पेड़ों का पुनर्निर्माण". Genetics. 144 (1): 389–399. doi:10.1093/genetics/144.1.389. PMC 1207511. PMID 8878702.
- ↑ Magalhães TR, Casey JP, Conroy J, Regan R, Fitzpatrick DJ, Shah N; et al. (2012). "Ancestry Mapper द्वारा HGDP और HapMap विश्लेषण से स्थानीय और वैश्विक जनसंख्या संबंधों का पता चलता है।". PLOS ONE. 7 (11): e49438. Bibcode:2012PLoSO...749438M. doi:10.1371/journal.pone.0049438. PMC 3506643. PMID 23189146.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Gillian Cooper; William Amos; Richard Bellamy; Mahveen Ruby Siddiqui; Angela Frodsham; Adrian V. S. Hill; David C. Rubinsztein (1999). "An Empirical Exploration of the Genetic Distance for 213 Human Microsatellite Markers". The American Journal of Human Genetics. 65 (4): 1125–1133. doi:10.1086/302574. PMC 1288246. PMID 10486332.
- ↑ Nei M, Roychoudhury AK (February 1974). "विषमलैंगिकता और आनुवंशिक दूरी का नमूनाकरण संस्करण". Genetics. 76 (2): 379–90. doi:10.1093/genetics/76.2.379. PMC 1213072. PMID 4822472.
- ↑ Rogers, J. S. (1972). Measures of similarity and genetic distance. In Studies in Genetics VII. pp. 145−153. University of Texas Publication 7213. Austin, Texas.
बाहरी संबंध
- The Estimation of Genetic Distance and Population Substructure from Microsatellite allele frequency data., Brent W. Murray (May 1996), McMaster University website on genetic distance
- Computing distance by stepwise genetic distance model, web pages of Bruce Walsh at the Department of Ecology and Evolutionary Biology at the University of Arizona Archived 2006-12-10 at the Wayback Machine