आरएनए-बाध्यकारी प्रोटीन: Difference between revisions
(Created page with "{{short description|Type of protein}} {{Hatnote|Not to be confused with retinol binding proteins, also abbreviated as RBPs.}} {{Use dmy dates|date=December 2016}} आर...") |
No edit summary |
||
| (4 intermediate revisions by 3 users not shown) | |||
| Line 1: | Line 1: | ||
{{short description|Type of protein}} | {{short description|Type of protein}} | ||
{{Hatnote| | {{Hatnote|[[रेटिनोल बाइंडिंग प्रोटीन]] के साथ भ्रमित न हों, जिसे आरबीपी के रूप में भी संक्षिप्त किया जाता है।}} | ||
'''आरएनए बाध्यकारी [[प्रोटीन]]''' अधिकांशतः आरबीपी के रूप में ऐसे संक्षिप्त प्रोटीन होते हैं जो आपस में एक-दूसरे से फंसे हुए आरएनए से जुड़ते हैं,<ref>{{MeshName|RNA-Binding+Proteins}}</ref> इस प्रकार कोशिकाएं जीव विज्ञान में और [[ ribonucleoprotein |राइबो न्यूक्लियोप्रोटीन]] कॉम्प्लेक्स बनाने में भाग लेते हैं। | |||
आरबीपी में विभिन्न [[संरचनात्मक रूप|संरचनात्मक रूपांकन]] होते हैं, जैसे कि [[आरएनए मान्यता मूल भाव|आरएनए मान्यता वाले मूल भाव]] (आरआरएम), डीएसआरएनए बाध्यकारी डोमेन, [[ जस्ता उंगली |जिंक फिंगर]] और <ref name="ReferenceA">{{cite journal | vauthors = Lunde BM, Moore C, Varani G | title = RNA-binding proteins: modular design for efficient function | journal = Nature Reviews. Molecular Cell Biology | volume = 8 | issue = 6 | pages = 479–90 | date = June 2007 | pmid = 17473849 | pmc = 5507177 | doi = 10.1038/nrm2178 }}</ref><ref>{{cite journal |last1=Liu |first1=S |last2=Li |first2=B |last3=Liang |first3=Q |last4=Liu |first4=A |last5=Qu |first5=L |last6=Yang |first6=J |title=आरएनए-प्रोटीन इंटरैक्शन का वर्गीकरण और कार्य।|journal=Wiley Interdisciplinary Reviews. RNA |date=November 2020 |volume=11 |issue=6 |pages=e1601 |doi=10.1002/wrna.1601 |pmid=32488992|s2cid=219284021 }}</ref> इसमें अन्य [[ कोशिका द्रव्य |कोशिका द्रव्य]] और [[ कोशिका केंद्रक |कोशिका केंद्रक]] प्रोटीन हैं। चूंकि अधिकांश परिपक्व आरएनए नाभिक से अपेक्षाकृत तेज़ी से निर्यात किए जाते हैं, नाभिक में अधिकांश आरबीपी प्रोटीन और अग्रदूत [[एमआरएनए]] के क्षेत्रों के रूप में उपस्थित होते हैं। इस प्रकार पूर्व-एमआरएनए को [[विषम राइबोन्यूक्लियोप्रोटीन कण]] (एचएनआरएनपी) कहा जाता है। | |||
विभिन्न सेलुलर प्रक्रियाओं में आरबीपी की महत्वपूर्ण भूमिका होती है जैसे: सेलुलर फलन, परिवहन और स्थानीयकरण इत्यादि। ये मुख्यतः विशेष रूप से आरएनए के पोस्ट-ट्रांसक्रिप्शनल नियंत्रण में प्रमुख भूमिका निभाते हैं, जैसे: [[आरएनए स्पिलिंग]], [[पॉलीएडेनाइलेशन]], एमआरएनए स्थिरीकरण, एमआरएनए [[उपकोशिकीय स्थानीयकरण]] और [[अनुवाद (जीव विज्ञान)]] इत्यादि। इस प्रकार [[यूकेरियोट]] कोशिकाएं विविध आरबीपी को अद्वितीय आरएनए-बाध्यकारी गतिविधि और प्रोटीन-प्रोटीन इंटरैक्शन के साथ व्यक्त करती हैं। यूकेरियोटिक आरबीपी डाटाबेस (यूआरबीपीडीबी) के अनुसार, मनुष्यों में 2961 [[जीन]] एन्कोडिंग आरबीपी हैं। [[विकास]] के समय, [[इंट्रोन्स]] की संख्या में वृद्धि के साथ आरबीपी की विविधता बहुत बढ़ गई। इस कारण विभिन्न प्रकार की विविधताओं के कारण सक्षम यूकेरियोटिक कोशिकाएं विभिन्न व्यवस्थाओं में आरएनए एक्सॉन का उपयोग करने के लिए, प्रत्येक आरएनए के लिए अद्वितीय आरएनपी (राइबोन्यूक्लियोप्रोटीन) को जन्म देती हैं। यद्यपि इस प्रकार जीन अभिव्यक्ति में पोस्ट-ट्रांसक्रिप्शनल विनियमन में आरबीपी की महत्वपूर्ण भूमिका है, अपेक्षाकृत कुछ आरबीपी का व्यवस्थित रूप से अध्ययन किया गया है। अब यह स्पष्ट हो गया है कि आरएनए-आरबीपी इंटरैक्शन जीवों के बीच कई जैविक प्रक्रियाओं में महत्वपूर्ण भूमिका निभाते हैं।<ref>{{cite journal | vauthors = Hogan DJ, Riordan DP, Gerber AP, Herschlag D, Brown PO | title = विविध आरएनए-बाध्यकारी प्रोटीन आरएनए के कार्यात्मक रूप से संबंधित सेटों के साथ परस्पर क्रिया करते हैं, एक व्यापक नियामक प्रणाली का सुझाव देते हैं| journal = PLOS Biology | volume = 6 | issue = 10 | pages = e255 | date = October 2008 | pmid = 18959479 | pmc = 2573929 | doi = 10.1371/journal.pbio.0060255 }}</ref><ref name="Glisovic" /><ref>{{cite journal |last1=Liu |first1=S |last2=Li |first2=B |last3=Liang |first3=Q |last4=Liu |first4=A |last5=Qu |first5=L |last6=Yang |first6=J |title=आरएनए-प्रोटीन इंटरैक्शन का वर्गीकरण और कार्य।|journal=Wiley Interdisciplinary Reviews. RNA |date=November 2020 |volume=11 |issue=6 |pages=e1601 |doi=10.1002/wrna.1601 |pmid=32488992|s2cid=219284021 }}</ref> | |||
== संरचना == | == संरचना == | ||
कई आरबीपी में मॉड्यूलर संरचनाएं होती हैं और केवल कुछ विशिष्ट | कई आरबीपी में मॉड्यूलर संरचनाएं होती हैं और केवल कुछ विशिष्ट मौलिक डोमेन के कई दोहराव से बने होते हैं जिनमें अधिकांशतः सीमित अनुक्रम होते हैं। विविधता की आवश्यकता को पूरा करने के लिए इन अनुक्रमों को अलग-अलग संयोजनों में व्यवस्थित किया जाता है। इस प्रकार विशिष्ट आरएनए की विशिष्ट प्रोटीन की मान्यता इन कुछ मौलिक डोमेन के पुनर्व्यवस्था के माध्यम से विकसित हुई है। प्रत्येक मूल डोमेन आरएनए को पहचानता है, अपितु इनमें से कई प्रोटीनों को कार्य करने के लिए कई सामान्य डोमेन में से की कई प्रतियों की आवश्यकता होती है।<ref name="ReferenceA"/> | ||
== विविधता == | == विविधता == | ||
जैसे ही परमाणु आरएनए [[आरएनए पोलीमरेज़]] से निकलता है, आरएनए ट्रांसक्रिप्ट तुरंत आरएनए-बाध्यकारी प्रोटीन से आच्छादित हो जाते हैं जो आरएनए चयापचय के हर पहलू को नियंत्रित करते हैं और आरएनए जैवजनन, परिपक्वता, परिवहन, सेलुलर स्थानीयकरण और स्थिरता सहित कार्य करते हैं। सभी आरबीपी आरएनए को बांधते हैं, | जैसे ही परमाणु आरएनए [[आरएनए पोलीमरेज़]] से निकलता है, आरएनए ट्रांसक्रिप्ट तुरंत आरएनए-बाध्यकारी प्रोटीन से आच्छादित हो जाते हैं जो आरएनए चयापचय के हर पहलू को नियंत्रित करते हैं और आरएनए जैवजनन, परिपक्वता, परिवहन, सेलुलर स्थानीयकरण और स्थिरता सहित कार्य करते हैं। इस प्रकार सभी आरबीपी आरएनए को बांधते हैं, चूंकि वे विभिन्न आरएनए-अनुक्रम विशिष्टताओं और समानता के साथ ऐसा करते हैं, जो आरबीपी को उनके लक्ष्यों और कार्यों के रूप में विविध होने की अनुमति देता है।<ref name="Glisovic" /> इन लक्ष्यों में एमआरएनए सम्मिलित होते हैं, जो इस प्रकार प्रोटीन के लिए कोड करता है, साथ ही कई कार्यात्मक [[गैर-कोडिंग आरएनए]] भी सम्मिलित हैं। एनसीआरएनए लगभग हमेशा राइबोन्यूक्लियोप्रोटीन कॉम्प्लेक्स के रूप में कार्य करते हैं न कि नग्न आरएनए के रूप में कार्य करते हैं। इस प्रकार इन गैर-कोडिंग आरएनए में [[माइक्रोआरएनए]], छोटे हस्तक्षेप करने वाले आरएनए (siआरएनए), साथ ही स्प्लिसेसोमल [[छोटे परमाणु आरएनए]] (एसएनआरएनए) सम्मिलित किया जाता हैं।<ref name="Matera">{{cite journal | vauthors = Matera AG, Terns RM, Terns MP | title = Non-coding RNAs: lessons from the small nuclear and small nucleolar RNAs | journal = Nature Reviews. Molecular Cell Biology | volume = 8 | issue = 3 | pages = 209–20 | date = March 2007 | pmid = 17318225 | doi = 10.1038/nrm2124 | s2cid = 30268055 }}</ref> | ||
== फलन == | |||
== | |||
=== आरएनए प्रसंस्करण और संशोधन === | === आरएनए प्रसंस्करण और संशोधन === | ||
==== वैकल्पिक | ==== वैकल्पिक विभाजन ==== | ||
वैकल्पिक विभाजन | '''वैकल्पिक विभाजन''' वह तंत्र है जिसके द्वारा [[जीन]] से परिपक्व एमआरएनए संदेशवाहक आरएनए के विभिन्न रूप उत्पन्न होते हैं। यह नियामक तंत्र है जिसके द्वारा [[एक्सॉन]] को एमआरएनए में सम्मिलित करने में भिन्नता से अधिक संबंधित प्रोटीन के उत्पादन की ओर ले जाती है, इस प्रकार संभावित जीनोमिक आउटपुट का विस्तार होता है। आरबीपी इस प्रक्रिया के नियमन में व्यापक रूप से कार्य करते हैं। कुछ बाध्यकारी प्रोटीन जैसे कि न्यूरोनल विशिष्ट आरएनए-बाध्यकारी प्रोटीन, अर्थात् [[NOVA1|नोवा1]], आरएनए (वाईसीएवाई जहां वाई पाइरीमिडीन, यू या सी को इंगित करता है) में विशिष्ट अनुक्रम को पहचानने और बाध्य करके एचएनआरएनए के उपसमूह के वैकल्पिक विभाजन को नियंत्रित करता है।<ref name="Glisovic">{{cite journal | vauthors = Glisovic T, Bachorik JL, Yong J, Dreyfuss G | title = आरएनए-बाध्यकारी प्रोटीन और पोस्ट-ट्रांसक्रिप्शनल जीन विनियमन| journal = FEBS Letters | volume = 582 | issue = 14 | pages = 1977–86 | date = June 2008 | pmid = 18342629 | pmc = 2858862 | doi = 10.1016/j.febslet.2008.03.004 }}</ref> ये प्रोटीन फिर इस लक्ष्य स्थल पर स्प्लिसोमल प्रोटीन की भर्ती करते हैं। एसआर प्रोटीन एसएनआरएनपी की भर्ती के माध्यम से वैकल्पिक विभाजन में अपनी भूमिका के लिए भी जाने जाते हैं, जो कि यू1 एसएनआरएनपी और यू2एएफ एसएनआरएनपी नामक [[spliceosome|स्पलाइसोसम]] बनाते हैं। चूंकि, आरबीपीभी इसका ही भाग हैं। इस प्रकार स्प्लिसोम एसएनआरएनए [[एसआर प्रोटीन]] सबयूनिट्स का जटिल है और यांत्रिक एजेंट के रूप में कार्य करता है जो [[इंट्रॉन]] को हटाता है और फ्लैंकिंग एक्सॉन को लिगेट करता है।<ref name="Matera"/> इस प्रकार कोर स्प्लिससम कॉम्प्लेक्स के अतिरिक्त, आरबीपी सिस-एक्टिंग आरएनए तत्वों की साइटों से भी जुड़ते हैं जो एक्सॉन को सम्मिलित करने या विभाजन के समय बहिष्करण को प्रभावित करते हैं। इन साइटों को एक्सोनिक स्प्लिसिंग एन्हांसर (ईएसई), एक्सोनिक स्प्लिसिंग साइलेंसर (ईएसएस), इंट्रोनिक स्प्लिसिंग एनहांसर (आईएसई) और इंट्रोनिक स्प्लिसिंग साइलेंसर (आईएसएस) के रूप में संदर्भित किया जाता है और उनके बंधन के स्थान के आधार पर, आरबीपी स्प्लिसिंग साइलेंसर या एन्हांसर के रूप में कार्य करते हैं।<ref>{{cite journal | vauthors = Fu XD, Ares M | title = आरएनए-बाइंडिंग प्रोटीन द्वारा वैकल्पिक स्प्लिसिंग का संदर्भ-निर्भर नियंत्रण| journal = Nature Reviews. Genetics | volume = 15 | issue = 10 | pages = 689–701 | date = October 2014 | pmid = 25112293 | pmc = 4440546 | doi = 10.1038/nrg3778 }}</ref> | ||
====आरएनए संपादन ==== | |||
[[File:Protein ADAR PDB 1qbj.png|thumb|right|alt=ADAR Protein. : आरएनए संपादन घटनाओं में शामिल एक आरएनए बाध्यकारी प्रोटीन।]]'''आरएनए संपादन''' के सबसे व्यापक रूप से अध्ययन किए गए रूप में एडीएआर प्रोटीन सम्मिलित है। यह प्रोटीन आरएनए के [[न्यूक्लियोटाइड]] सामग्री को बदलकर एमआरएनए ट्रांसक्रिप्ट के [[पोस्ट-ट्रांसक्रिप्शनल संशोधन]] के माध्यम से कार्य करता है। इस प्रकार यह एडीएआर द्वारा उत्प्रेरित एंजाइमिक प्रतिक्रिया में [[एडेनोसाइन]] को [[ आइनोसीन |आइनोसीन]] में परिवर्तित करने के माध्यम से किया जाता है। यह प्रक्रिया [[जीनोम]] द्वारा एन्कोड किए गए आरएनए अनुक्रम को प्रभावी विधि से परिवर्तित हो जाती है और जीन उत्पादों की विविधता को बढ़ाती है। इस प्रकार अधिकांश आरएनए संपादन आरएनए के गैर-कोडिंग क्षेत्रों पर होता है; चूंकि, कुछ प्रोटीन-एन्कोडिंग आरएनए ट्रांसक्रिप्शंस को संपादन के अधीन दिखाया गया है, जिसके परिणामस्वरूप उनके प्रोटीन के अमीनो एसिड अनुक्रम में अंतर होता है। इसका उदाहरण ग्लूटामेट रिसेप्टर एमआरएनए है जहां ग्लूटामाइन को आर्गिनिन में परिवर्तित किया जाता है जिससे प्रोटीन की कार्यक्षमता में परिवर्तन होता है।<ref name="Glisovic" /> | |||
==== | |||
[[File:Protein ADAR PDB 1qbj.png|thumb|right|alt=ADAR Protein. : आरएनए संपादन घटनाओं में शामिल एक आरएनए बाध्यकारी प्रोटीन।]]आरएनए संपादन के सबसे व्यापक रूप से अध्ययन किए गए रूप में एडीएआर प्रोटीन | |||
==== पॉलीएडेनिलेशन ==== | ==== पॉलीएडेनिलेशन ==== | ||
{{Main| | {{Main|पॉलीएडेनाइलेशन}} | ||
'''पॉलीएडेनाइलेशन''' [[तीन प्रमुख अअनुवादित क्षेत्र]] के भीतर एएयूएएए अनुक्रम के डाउनस्ट्रीम लगभग 20 आधारों के आरएनए प्रतिलेख में एडिनाइलेट अवशेषों की एक टेल के अतिरिक्त प्राप्त होती है। इस प्रकार एमआरएनए के पॉलीएडेनाइलेशन का इसके [[परमाणु परिवहन]], अनुवाद दक्षता और स्थिरता पर गहरा प्रभाव पड़ता है। इन सभी के साथ-साथ पॉलीएडेनाइलेशन की प्रक्रिया विशिष्ट आरबीपी के बंधन पर निर्भर करती है। इस प्रकार कुछ अपवादों के साथ सभी यूकेरियोटिक एमआरएनए को लगभग 200 न्यूक्लियोटाइड्स की 3' पॉली (ए) टेल प्राप्त करने के लिए संसाधित किया जाता है। इस प्रकार इस प्रक्रिया में आवश्यक प्रोटीन क्षेत्रों में से सीपीएसएफ है। [[सीपीएसएफ]] 3' टेल (एएयूएएए) अनुक्रम से जुड़ता है और साथ में पॉली (ए)[[पॉली (ए) -बाइंडिंग प्रोटीन|पॉली (ए) -बाध्यकारी प्रोटीन]] नामक अन्य प्रोटीन के साथ, पॉली (ए) पोलीमरेज़ की गतिविधि को भरती और उत्तेजित करता है। पॉली (ए) पोलीमरेज़ अपने आप में निष्क्रिय है और इन अन्य प्रोटीनों को ठीक से कार्य करने के लिए बाध्य करने की आवश्यकता है।<ref name="Glisovic" /> | |||
=== निर्यात === | === निर्यात === | ||
प्रसंस्करण पूरा होने के | प्रसंस्करण पूरा होने के पश्चात एमआरएनए को सेल न्यूक्लियस से साइटोप्लाज्म तक ले जाने की आवश्यकता होती है। यह तीन-चरण की प्रक्रिया है जिसमें नाभिक में कार्गो-वाहक क्षेत्र की पीढ़ी सम्मिलित है, जिसके बाद [[परमाणु छिद्र परिसर|परमाणु छिद्र क्षेत्र]] के माध्यम से क्षेत्र का स्थानांतरण होता है और अंत में कार्गो को साइटोप्लाज्म में छोड़ दिया जाता है। इस प्रकार वाहक को बाद में पुनर्नवीनीकरण किया जाता है। टैप/एनएक्सएफ1:p15 हेटेरोडिमर को एमआरएनए निर्यात में प्रमुख खिलाड़ी माना जाता है। ज़ेनोपस लाविस मेंढकों में टैप की ओवर-एक्सप्रेशन उन प्रतिलेखों के निर्यात को बढ़ाती है जो अन्यथा अक्षम रूप से निर्यात किए जाते हैं। चूंकि टैप को एडेप्टर प्रोटीन की आवश्यकता होती है क्योंकि यह एमआरएनए के साथ सीधे संपर्क करने में असमर्थ है। इस कारण एली/आरईएफ प्रोटीन एमआरएनए भर्ती टीएपी से संपर्क करता है, और बांध कर रखता है।<ref name="Glisovic" /> | ||
=== एमआरएनए स्थानीयकरण === | === एमआरएनए स्थानीयकरण === | ||
स्थानिक रूप से विनियमित प्रोटीन उत्पादन की अनुमति देकर जीन अभिव्यक्ति के नियमन के लिए | स्थानिक रूप से विनियमित प्रोटीन उत्पादन की अनुमति देकर जीन अभिव्यक्ति के नियमन के लिए '''एमआरएनए स्थानीयकरण''' महत्वपूर्ण है। एमआरएनए स्थानीयकरण के माध्यम से प्रोटीन को सेल के अपने इच्छित लक्ष्य स्थल में अनुवादित किया जाता है। प्रारंभिक विकास के समय यह विशेष रूप से महत्वपूर्ण है जब तेजी से सेल दरार अलग-अलग कोशिकाओं को एमआरएनए के विभिन्न संयोजन देते हैं जो बाद में अलग-अलग सेल भाग्य को जन्म दे सकते हैं। आरबीपी इस एमआरएनए के स्थानीयकरण में महत्वपूर्ण हैं जो सुनिश्चित करता है कि प्रोटीन केवल उनके इच्छित क्षेत्रों में अनुवादित हैं। इस प्रकार इनमें से प्रोटीन [[ZBP1|जेडबीपी1]] है। जेडबीपी1 प्रतिलेखन के स्थल पर [[बीटा actin|बीटा एक्टिन]] एमआरएनए से जुड़ता है और एमआरएनए के साथ साइटोप्लाज्म में चला जाता है। इसके पश्चात यह इस एमआरएनए को कई असममित सेल प्रकारों के लैमेला (कोशिका जीव विज्ञान) क्षेत्र में स्थानांतरित करता है जहां इसका अनुवाद किया जा सकता है।<ref name="Glisovic" /> इस प्रकार 2008 में यह प्रस्तावित किया गया था कि [[FMR1|एफएमआर1]] सुसंस्कृत हिप्पोकैम्पस न्यूरॉन्स के न्यूरोनल डेंड्राइट्स में कई डेंड्राइटिक एमआरएनए के उत्तेजना-प्रेरित स्थानीयकरण में सम्मिलित था।<ref>{{cite journal | vauthors = Dictenberg JB, Swanger SA, Antar LN, Singer RH, Bassell GJ | title = गतिविधि-निर्भर डेंड्राइटिक एमआरएनए ट्रांसपोर्ट में एफएमआरपी के लिए एक सीधी भूमिका फ़िलाओपोडियल-स्पाइन मॉर्फोजेनेसिस को नाजुक एक्स सिंड्रोम से जोड़ती है| journal = Developmental Cell | volume = 14 | issue = 6 | pages = 926–39 | date = June 2008 | pmid = 18539120 | pmc = 2453222 | doi = 10.1016/j.devcel.2008.04.003 }}</ref> सीए1 हिप्पोकैम्पस न्यूरॉन्स के माइक्रोडिसेक्टेड डेंड्राइट्स में उपस्थित एफएमआरP-बाउंड आरएनए के अधिकांशतः वर्तमान समय के अध्ययनों से भिन्न प्रकार से इसके विरुद्ध एफएमआरपी-रिक्त माउस में स्थानीयकरण में कोई परिवर्तन नहीं हुआ हैं।<ref>{{Cite journal |last=Hale |first=Caryn R. |last2=Sawicka |first2=Kirsty |last3=Mora |first3=Kevin |last4=Fak |first4=John J. |last5=Kang |first5=Jin Joo |last6=Cutrim |first6=Paula |last7=Cialowicz |first7=Katarzyna |last8=Carroll |first8=Thomas S. |last9=Darnell |first9=Robert B. |date=2021-12-23 |title=FMRP सेल बॉडी और CA1 पिरामिड न्यूरॉन्स के डेंड्राइट्स में अलग-अलग कार्यों को एन्कोडिंग करने वाले mRNAs को नियंत्रित करता है|url=https://pubmed.ncbi.nlm.nih.gov/34939924/ |journal=eLife |volume=10 |pages=e71892 |doi=10.7554/eLife.71892 |issn=2050-084X |pmc=8820740 |pmid=34939924}}</ref> | ||
=== अनुवाद === | === अनुवाद === | ||
ट्रांसलेशनल विनियमन जीन अभिव्यक्ति को नियंत्रित करने के लिए | ट्रांसलेशनल विनियमन जीन अभिव्यक्ति को नियंत्रित करने के लिए तीव्र तंत्र प्रदान करता है। इस प्रकार ट्रांसक्रिप्शनल स्तर पर जीन अभिव्यक्ति को नियंत्रित करने के अतिरिक्त, एमआरएनए पहले से ही लिखित है अपितु राइबोसोम की भर्ती नियंत्रित है। जब सिग्नल ट्रांसलेशन को सक्रिय करता है तो यह तेजी से प्रोटीन उत्पन्न करता है। इस प्रकार जेडबीपी1 बी-एक्टिन एमआरएनए के स्थानीयकरण में अपनी भूमिका के अतिरिक्त अनुवाद दीक्षा को अवरुद्ध करके बीटा-एक्टिन एमआरएनए के अनुवाद संबंधी दमन में भी सम्मिलित है। राइबोसोम को ठीक से बाँधने और अनुवाद प्रारंभ करने की अनुमति देने के लिए जेडबीपी1 को एमआरएनए से हटाया जाना चाहिए।<ref name="Glisovic" /> | ||
== प्रोटीन-आरएनए इंटरैक्शन == | == प्रोटीन-आरएनए इंटरैक्शन == | ||
[[File:RNA binding proteins RNA binding sites.svg|thumb|right|आरएनए-बाध्यकारी प्रोटीन के विविध आरएनए संपर्क]]आरएनए-बाध्यकारी प्रोटीन अपने अनुक्रमों, संरचनाओं, रूपांकनों और आरएनए संशोधनों को पहचानकर अपने आरएनए लक्ष्यों की अत्यधिक विशिष्ट पहचान प्रदर्शित करते हैं।<ref>{{cite journal |last1=Liu |first1=S |last2=Li |first2=B |last3=Liang |first3=Q |last4=Liu |first4=A |last5=Qu |first5=L |last6=Yang |first6=J |title=आरएनए-प्रोटीन इंटरैक्शन का वर्गीकरण और कार्य।|journal=Wiley Interdisciplinary Reviews. RNA |date=November 2020 |volume=11 |issue=6 |pages=e1601 |doi=10.1002/wrna.1601 |pmid=32488992|s2cid=219284021 }}</ref> आरएनए-बाध्यकारी प्रोटीन के विशिष्ट बंधन उन्हें अपने लक्ष्यों को अलग करने और आरएनए प्रतिलेख की पीढ़ी, परिपक्वता और जीवन काल के नियंत्रण के माध्यम से विभिन्न प्रकार के सेलुलर कार्यों को विनियमित करने की अनुमति देते हैं। यह अंतःक्रिया ट्रांसक्रिप्शन के | [[File:RNA binding proteins RNA binding sites.svg|thumb|right|आरएनए-बाध्यकारी प्रोटीन के विविध आरएनए संपर्क]]आरएनए-बाध्यकारी प्रोटीन अपने अनुक्रमों, संरचनाओं, रूपांकनों और आरएनए संशोधनों को पहचानकर अपने आरएनए लक्ष्यों की अत्यधिक विशिष्ट पहचान प्रदर्शित करते हैं।<ref>{{cite journal |last1=Liu |first1=S |last2=Li |first2=B |last3=Liang |first3=Q |last4=Liu |first4=A |last5=Qu |first5=L |last6=Yang |first6=J |title=आरएनए-प्रोटीन इंटरैक्शन का वर्गीकरण और कार्य।|journal=Wiley Interdisciplinary Reviews. RNA |date=November 2020 |volume=11 |issue=6 |pages=e1601 |doi=10.1002/wrna.1601 |pmid=32488992|s2cid=219284021 }}</ref> इस प्रकार आरएनए-बाध्यकारी प्रोटीन के विशिष्ट बंधन उन्हें अपने लक्ष्यों को अलग करने और आरएनए प्रतिलेख की पीढ़ी, परिपक्वता और जीवन काल के नियंत्रण के माध्यम से विभिन्न प्रकार के सेलुलर कार्यों को विनियमित करने की अनुमति देते हैं। यह अंतःक्रिया ट्रांसक्रिप्शन के समय प्रारंभ होती है क्योंकि कुछ आरबीपी गिरावट तक आरएनए के लिए बाध्य रहते हैं, जबकि अन्य आरएनए स्पिलिंग, प्रसंस्करण, परिवहन और स्थानीयकरण को विनियमित करने के लिए केवल क्षणिक रूप से आरएनए से जुड़ते हैं।<ref name="Stefl">{{cite journal | vauthors = Stefl R, Skrisovska L, Allain FH | title = राइबोन्यूक्लियोप्रोटीन कण में प्रोटीन द्वारा आरएनए अनुक्रम- और आकार-निर्भर पहचान| journal = EMBO Reports | volume = 6 | issue = 1 | pages = 33–8 | date = January 2005 | pmid = 15643449 | pmc = 1299235 | doi = 10.1038/sj.embor.7400325 }}</ref> विभिन्न प्रकार के ऊतकों और जीवों में आरएनए-बाध्यकारी प्रोटीनों के प्रत्यक्ष आरएनए बाध्यकारी साइटों की पहचान करने के लिए [[क्रॉस-लिंकिंग इम्यूनोप्रिसिपेशन]] (सीएलआईपी) विधियों का उपयोग किया जाता है। इस खंड में, सबसे व्यापक रूप से अध्ययन किए गए आरएनए-बाध्यकारी डोमेन (आरएनए-रिकग्निशन मोटिफ, डबल-स्ट्रैंडेड आरएनए-बाध्यकारी मोटिफ, जिंक-फिंगर मोटिफ) के तीन वर्गों पर चर्चा की जाती हैं। | ||
=== आरएनए-रिकग्निशन मोटिफ (आरआरएम) === | === आरएनए-रिकग्निशन मोटिफ (आरआरएम) === | ||
आरएनए रिकग्निशन मोटिफ, जो सबसे | आरएनए रिकग्निशन मोटिफ, जो सबसे साधारण आरएनए-बाध्यकारी मोटिफ है, 75-85 [[ एमिनो एसिड |एमिनो एसिड]] का छोटा प्रोटीन डोमेन है जो दो α-हेलीकॉप्स के खिलाफ चार-फंसे हुए β-शीट बनाता है। यह मान्यता मूल भाव कई सेलुलर कार्यों में विशेष रूप से एमआरएनए / rआरएनए प्रसंस्करण, विभाजन, अनुवाद विनियमन, आरएनए निर्यात और आरएनए स्थिरता में अपनी भूमिका निभाता है। [[एनएमआर स्पेक्ट्रोस्कोपी]] और [[एक्स - रे क्रिस्टलोग्राफी]] के माध्यम से आरआरएम की दस संरचनाओं की पहचान की गई है। इस प्रकार ये संरचनाएं आरआरएम की प्रोटीन-आरएनए मान्यता की गहनता को दर्शाती हैं क्योंकि इसमें प्रोटीन-आरएनए इंटरैक्शन के अतिरिक्त आरएनए-आरएनए और प्रोटीन-प्रोटीन इंटरैक्शन सम्मिलित हैं। उनकी जटिलता के अतिरिक्त, सभी दस संरचनाओं में कुछ सामान्य विशेषताएं हैं। सभी आरआरएम की मुख्य प्रोटीन सतहों की चार-फंसे β-शीट को आरएनए के साथ बातचीत करने के लिए पाया गया, जो साधारणतौर पर विशिष्ट तरीके से दो या तीन न्यूक्लियोटाइड्स से संपर्क करता है। इसके अतिरिक्त, अंतर-डोमेन लिंकर और इस प्रकार आरएनए के बीच और यह स्वयं आरआरएम के बीच बातचीत के माध्यम से भिन्नता के प्रति मजबूत आरएनए बंधन संबंध और विशिष्टता प्राप्त की जाती है। आरआरएम की यह नमनीयता बताती है कि आरआरएम सबसे प्रचुर मात्रा में डोमेन क्यों है और यह विभिन्न जैविक कार्यों में महत्वपूर्ण भूमिका क्यों निभाता है।<ref name="Stefl" /> | ||
=== डबल-स्ट्रैंडेड आरएनए-बाध्यकारी मोटिफ === | |||
=== डबल-स्ट्रैंडेड आरएनए- | |||
{{Pfam box | {{Pfam box | ||
|image=PDB 2b7t EBI.jpg | |image=PDB 2b7t EBI.jpg | ||
| Line 66: | Line 48: | ||
|below=Use the Pfam clan for the homologous superfamily. | |below=Use the Pfam clan for the homologous superfamily. | ||
}} | }} | ||
डबल-स्ट्रैंडेड आरएनए- | '''डबल-स्ट्रैंडेड आरएनए-बाध्यकारी मोटिफ''' (डीएसआरएम, डीएसआरबीडी), 70-75 एमिनो-एसिड डोमेन, [[आरएनए प्रसंस्करण]], आरएनए सबसेलुलर लोकलाइजेशन, [[आरएनए हस्तक्षेप]], [[आरएनए संपादन]] और ट्रांसलेशनल दमन में महत्वपूर्ण भूमिका निभाता है। इस प्रकार 2005 तक हल की गई डोमेन की सभी तीन संरचनाओं में एकत्रित करने वाली विशेषताएँ हैं जो बताती हैं कि कैसे डीएसआरएमएस केवल डीएसडीएनए के अतिरिक्त डीएसआरएनए से जुड़ते हैं। डीएसआरएम आरएनए डुप्लेक्स के साथ α-हेलीकॉप्टर और β1-β2 लूप दोनों के माध्यम से बातचीत करने के लिए पाए गए हैं। इसके अतिरिक्त, सभी तीन डीएसआरबीएम संरचनाएं प्रमुख संरचना और छोटे संरचना के चीनी-फॉस्फेट रीढ़ के साथ संपर्क बनाती हैं, जो इस प्रकार [[अल्फा हेलिक्स]] 2 के ए[[ N- टर्मिनस | N- टर्मिनस]] क्षेत्र के साथ β1-β2 लूप द्वारा मध्यस्थ होता है। यह बातचीत है आरएनए डबल हेलिक्स के आकार के लिए अद्वितीय अनुकूलन क्योंकि इसमें 2'-हाइड्रॉक्सिल और फॉस्फेट ऑक्सीजन सम्मिलित है। इस प्रकार डीएसआरबीएम के बीच सामान्य संरचनात्मक विशेषताओं के अतिरिक्त, वे अलग-अलग रासायनिक ढांचे का प्रदर्शन करते हैं, जो आरएनए संरचनाओं के लिए विविधता के लिए विशिष्टता की अनुमति देता है जिसमें स्टेम-लूप, आंतरिक लूप, उभार या बेमेल सम्मिलित हैं।<ref name="Stefl" /> | ||
=== जिंक फिंगर === | |||
[[File:Zinc finger rendered.png|thumb|right|alt=Zinc finger.| जिंक फिंगर: प्रोटीन के जिंक-फिंगर मोटिफ का कार्टून प्रतिनिधित्व। जिंक आयन (हरा) दो हिस्टडीन और दो सिस्टीन अमीनो एसिड अवशेषों द्वारा समन्वित होता है।]]यूकेरियोटिक जीनोम के भीतर सीसीएचएच-टाइप [[ जस्ता उंगली |जिंक फिंगर]] डोमेन सबसे साधारण [[डीएनए-बाध्यकारी डोमेन]] हैं। डीएनए की उच्च अनुक्रम-विशिष्ट पहचान प्राप्त करने के लिए, मॉड्यूलर फैशन में कई जिंक फिंगर का उपयोग किया जाता है। जिंक फिंगर ββα प्रोटीन फोल्ड प्रदर्शित करती हैं जिसमें β-हेयरपिन और α-हेलिक्स के माध्यम से {{chem|Zn|2+}} आयन जुड़ जाते हैं। इसके अतिरिक्त, प्रमुख संरचना में डीएनए आधारों के साथ α-हेलिक्स की प्रोटीन साइड-चेन के बीच की बातचीत डीएनए-अनुक्रम-विशिष्ट पहचान के लिए अनुमति देती है। डीएनए की व्यापक मान्यता के अतिरिक्त, हाल ही में ऐसी खोजें हुई हैं कि ज़िंक फिंगर में आरएनए को पहचानने की क्षमता भी होती है। इस प्रकार सीसीएचएच जिंक फिंगर्स के अतिरिक्त, सीसीसीएच जिंक फिंगर्स को हाल ही में इंटरमॉलिक्युलर [[ हाइड्रोजन बंध |हाइड्रोजन बंध]] और आरएनए बेस के वाटसन-क्रिक किनारों के बीच बातचीत के माध्यम से एकल-फंसे हुए आरएनए की अनुक्रम-विशिष्ट पहचान को नियोजित करने के लिए खोजा गया था। इस प्रकार सीसीएचएच-टाइप जिंक फिंगर्स आरएनए बाध्यकारी के दो तरीके अपनाते हैं। सबसे पहले, जिंक फिंगर [[ दोहरी कुंडली |दोहरी कुंडली]] की रीढ़ के साथ गैर-विशिष्ट बातचीत करती हैं, जबकि दूसरी मोड जिंक फिंगर को विशेष रूप से अलग-अलग आधारों को पहचानने की अनुमति देती है जो बाहर निकलते हैं। सीसीएचएच-प्रकार से भिन्न, सीसीसीएच-प्रकार की जिंक फिंगर आरएनए बाध्यकारी की अन्य विधा प्रदर्शित करती है, जिसमें एकल-स्ट्रैंडेड आरएनए को अनुक्रम-विशिष्ट विधि से पहचाना जाता है। कुल मिलाकर, जिंक फिंगर्स डीएसडीएनए अनुक्रम से बाध्यकारी के माध्यम से और एसएसआरएनए अनुक्रम से बाध्यकारी के माध्यम से आरएनए को सीधे डीएनए की पहचान कर सकते हैं।<ref name="Stefl" /> | |||
=== जिंक | |||
[[File:Zinc finger rendered.png|thumb|right|alt=Zinc finger.| जिंक फिंगर: प्रोटीन के जिंक-फिंगर मोटिफ का कार्टून प्रतिनिधित्व। जिंक आयन (हरा) दो हिस्टडीन और दो सिस्टीन अमीनो एसिड अवशेषों द्वारा समन्वित होता है।]]यूकेरियोटिक जीनोम के भीतर सीसीएचएच-टाइप [[ जस्ता उंगली ]] डोमेन सबसे | |||
== भ्रूण के विकास में भूमिका == | == भ्रूण के विकास में भूमिका == | ||
[[File:CrawlingCelegans.gif|thumb|right|alt=Caenorhabditis elegans.|क्रॉलिंग कैनोर्हाडाइटिस एलिगेंस|सी. एलिगेंस उभयलिंगी कीड़ा]]आरएनए-बाध्यकारी प्रोटीन के आरएनए के ट्रांसक्रिप्शनल और [[पोस्ट-ट्रांसक्रिप्शनल विनियमन]] की विकास के | [[File:CrawlingCelegans.gif|thumb|right|alt=Caenorhabditis elegans.|क्रॉलिंग कैनोर्हाडाइटिस एलिगेंस|सी. एलिगेंस उभयलिंगी कीड़ा]]आरएनए-बाध्यकारी प्रोटीन के आरएनए के ट्रांसक्रिप्शनल और [[पोस्ट-ट्रांसक्रिप्शनल विनियमन]] की विकास के समय जीन अभिव्यक्ति के क्रम को विनियमित करने में भूमिका होती है।<ref name="Appasani2008">{{cite book |last=Appasani |first=Krishnarao |title=MicroRNAs: From Basic Science to Disease Biology |url=https://books.google.com/books?id=H_W8rqYyc9MC&pg=PA485 |year=2008 |publisher=[[Cambridge University Press]] |isbn=978-0-521-86598-2 |page=485 |access-date=12 May 2013}}</ref> नेमाटोड कैनोर्हाडाइटिस एलिगेंस पर व्यापक शोध या सी एलिगेंस ने आरएनए-बाध्यकारी प्रोटीन की [[जर्मलाइन]] और प्रारंभिक भ्रूण विकास के समय आवश्यक कारकों के रूप में पहचान की है। इस प्रकार इनके विशिष्ट कार्य में [[दैहिक (जीव विज्ञान)]] ऊतकों ([[न्यूरॉन|न्यूरॉन्स]], [[हाइपोडर्मिस]], [[मांसपेशियों]] और उत्सर्जन कोशिकाओं) के विकास के साथ-साथ विकासात्मक घटनाओं के लिए समय संकेत प्रदान करना सम्मिलित है। फिर भी, उनके आरएनए लक्ष्यों की पहचान करने में कठिनाई के कारण विकास में आरबीपी के कार्य के पीछे के तंत्र की खोज करना असाधारण रूप से चुनौतीपूर्ण है। ऐसा इसलिए है क्योंकि अधिकांशतः आरबीपी में साधारणतयः कई आरएनए लक्ष्य होते हैं।<ref name="Lee">{{cite book |last1=Lee |first1=Min-Ho |last2=Schedl |first2=Tim | name-list-style = vanc |date=18 April 2006 |chapter=RNA-binding proteins |title=वर्मबुक|pages=1–13 |chapter-url=https://www.ncbi.nlm.nih.gov/books/NBK19709/ |title-link=वर्मबुक|journal=WormBook: The Online Review of C. Elegans Biology |publisher=वर्मबुक|doi=10.1895/wormbook.1.79.1 |pmid=18050487 |pmc=4781538 }}</ref> चूंकि, यह निर्विवाद है कि आरबीपीठोस तरीके से विकासात्मक मार्गों को विनियमित करने में महत्वपूर्ण नियंत्रण रखते हैं। | ||
=== जर्मलाइन विकास === | === जर्मलाइन विकास === | ||
[[ड्रोसोफिला मेलानोगास्टर]] में, एलाव, एसएक्सएल और टीआरए-2 आरएनए-बाध्यकारी प्रोटीन एन्कोडिंग जीन हैं जो प्रारंभिक [[लिंग-निर्धारण प्रणाली]] और दैहिक यौन अवस्था के रखरखाव में महत्वपूर्ण हैं।<ref>{{cite journal | vauthors = Bandziulis RJ, Swanson MS, Dreyfuss G | title = विकास नियामकों के रूप में आरएनए-बाध्यकारी प्रोटीन| journal = Genes & Development | volume = 3 | issue = 4 | pages = 431–7 | date = April 1989 | pmid = 2470643 | doi = 10.1101/gad.3.4.431 | doi-access = free }}</ref> ये जीन ड्रोसोफिला में सेक्स-विशिष्ट विभाजन को विनियमित करके पोस्ट-ट्रांसक्रिप्शनल स्तर पर प्रभाव डालते हैं। | [[ड्रोसोफिला मेलानोगास्टर]] में, एलाव, एसएक्सएल और टीआरए-2 आरएनए-बाध्यकारी प्रोटीन एन्कोडिंग जीन हैं, जो इस प्रकार प्रारंभिक [[लिंग-निर्धारण प्रणाली]] और दैहिक यौन अवस्था के रखरखाव में महत्वपूर्ण हैं।<ref>{{cite journal | vauthors = Bandziulis RJ, Swanson MS, Dreyfuss G | title = विकास नियामकों के रूप में आरएनए-बाध्यकारी प्रोटीन| journal = Genes & Development | volume = 3 | issue = 4 | pages = 431–7 | date = April 1989 | pmid = 2470643 | doi = 10.1101/gad.3.4.431 | doi-access = free }}</ref> ये जीन ड्रोसोफिला में सेक्स-विशिष्ट विभाजन को विनियमित करके पोस्ट-ट्रांसक्रिप्शनल स्तर पर प्रभाव डालते हैं। इस प्रकार एसएक्सएल महिलाओं में कार्यात्मक टीआरए एमआरएनए उत्पन्न करने के लिए स्त्रीलिंग जीन टीआरए का धनात्मक नियमन करता है। C. एलिगेंस में, फाॅग-1, माॅग-1/-4/-5 और आरएनपी-4 सहित आरएनए-बाध्यकारी प्रोटीन जर्मलाइन और दैहिक लिंग निर्धारण नियंत्रित करते हैं। इसके अतिरिक्त, कई आरबीपी जैसे जीएलडी-1, जीएलडी-3, डीएजेड-1, पीजीएल-1 और ओएमए-1/-2 [[अर्धसूत्रीविभाजन]] [[ प्रोफेज़ |प्रोफेज़]] प्रगति, [[युग्मकजनन]], और अंडकोशिका परिपक्वता के समय अपने नियामक कार्यों को पूरा करते हैं।<ref name="Lee" /> | ||
===दैहिक विकास=== | ===दैहिक विकास=== | ||
जर्मलाइन विकास में आरबीपी के कार्यों के | जर्मलाइन विकास में आरबीपी के कार्यों के अतिरिक्त, पोस्ट-ट्रांसक्रिप्शनल नियंत्रण भी दैहिक विकास में महत्वपूर्ण भूमिका निभाता है। रोगाणु और प्रारंभिक भ्रूण विकास में सम्मिलित आरबीपी से भिन्न, दैहिक विकास में कार्य करने वाले आरबीपी एमआरएनए लक्ष्यों के ऊतक-विशिष्ट वैकल्पिक विभाजन को विनियमित करते हैं। उदाहरण के लिए, आरआरएम डोमेन वाले एमईसी-8 और यूएनसी-75 क्रमशः हाइपोडर्मिस और तंत्रिका तंत्र के क्षेत्रों में स्थानीय होते हैं।<ref name="Lee" /> इसके अतिरिक्त, अन्य आरआरएम-युक्त आरबीपी, ईएक्ससी-7, दैहिक विकास के समय भ्रूण के उत्सर्जक नलिका कोशिकाओं और पूरे तंत्रिका तंत्र में स्थानीयकरण के लिए प्रकट होता है। | ||
===तंत्रिका संबंधी विकास=== | ===तंत्रिका संबंधी विकास=== | ||
जेडबीपी1 को हिप्पोकैम्पस न्यूरॉन्स में डेंड्रिटोजेनेसिस ([[ डेन्ड्राइट | डेन्ड्राइट]] गठन) को विनियमित करने के लिए दिखाया गया था।<ref>{{cite journal | vauthors = Perycz M, Urbanska AS, Krawczyk PS, Parobczak K, Jaworski J | title = जिपकोड बाइंडिंग प्रोटीन 1 हिप्पोकैम्पस न्यूरॉन्स में डेंड्राइटिक आर्बर्स के विकास को नियंत्रित करता है| journal = The Journal of Neuroscience | volume = 31 | issue = 14 | pages = 5271–85 | date = April 2011 | pmid = 21471362 | doi = 10.1523/JNEUROSCI.2387-10.2011 | pmc = 6622686 | doi-access = free }}</ref> इस प्रकार डेन्ड्राइट निर्माण में सम्मिलित अन्य आरएनए-बाध्यकारी प्रोटीन [[PUM2|पम2]] और नैनो हैं,<ref>{{cite journal | vauthors = Ye B, Petritsch C, Clark IE, Gavis ER, Jan LY, Jan YN | title = ड्रोसोफिला परिधीय न्यूरॉन्स में डेंड्राइट मॉर्फोजेनेसिस के लिए नैनो और पुमिलियो आवश्यक हैं| journal = Current Biology | volume = 14 | issue = 4 | pages = 314–21 | date = February 2004 | pmid = 14972682 | doi = 10.1016/j.cub.2004.01.052 | doi-access = free }}</ref> जैसे एफएमआर1, [[CPEB|सीपीईबी]] और स्टॉफ़ेन (प्रोटीन) इसका प्रमुख उदाहरण हैं।<ref>{{cite journal | vauthors = Vessey JP, Macchi P, Stein JM, Mikl M, Hawker KN, Vogelsang P, Wieczorek K, Vendra G, Riefler J, Tübing F, Aparicio SA, Abel T, Kiebler MA | display-authors = 6 | title = murine Staufen1 के लिए फ़ंक्शन एलील के नुकसान से डेंड्राइटिक Staufen1-RNP डिलीवरी और डेंड्राइटिक स्पाइन मॉर्फोजेनेसिस की हानि होती है| journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 105 | issue = 42 | pages = 16374–9 | date = October 2008 | pmid = 18922781 | pmc = 2567905 | doi = 10.1073/pnas.0804583105 | bibcode = 2008PNAS..10516374V | jstor = 25465098 | doi-access = free }}</ref> | |||
== कैंसर में भूमिका == | == कैंसर में भूमिका == | ||
ट्यूमर के विकास में महत्वपूर्ण भूमिका निभाने के लिए आरबीपी उभर रहे हैं।<ref name="Human Cancers 2017">{{cite journal | vauthors = Wang ZL, Li B, Luo YX, Lin Q, Liu SR, Zhang XQ, Zhou H, Yang JH, Qu LH | display-authors = 6 | title = मानव कैंसर में आरएनए-बाध्यकारी प्रोटीन का व्यापक जीनोमिक लक्षण वर्णन| journal = Cell Reports | volume = 22 | issue = 1 | pages = 286–298 | date = January 2018 | pmid = 29298429 | doi = 10.1016/j.celrep.2017.12.035 | doi-access = free }}</ref> सैकड़ों आरबीपी मानव कैंसर में स्पष्ट रूप से विकृत हैं और सामान्य ऊतकों से संबंधित ट्यूमर में प्रमुख गिरावट दिखाते हैं।<ref name="Human Cancers 2017"/>कई आरबीपी विभिन्न प्रकार के कैंसर में भिन्न रूप से अभिव्यक्त होते हैं, उदाहरण के लिए केएचडीआरबीएस1(सैम68),<ref>{{cite journal | vauthors = Bielli P, Busà R, Paronetto MP, Sette C | title = The RNA-binding protein Sam68 is a multifunctional player in human cancer | journal = Endocrine-Related Cancer | volume = 18 | issue = 4 | pages = R91–R102 | date = August 2011 | pmid = 21565971 | doi = 10.1530/ERC-11-0041 | url = http://erc.endocrinology-journals.org/content/18/4/R91 | hdl = 2108/88068 | hdl-access = free }}</ref><ref>{{cite journal | vauthors = Liao WT, Liu JL, Wang ZG, Cui YM, Shi L, Li TT, Zhao XH, Chen XT, Ding YQ, Song LB| display-authors = 6 | title = High expression level and nuclear localization of Sam68 are associated with progression and poor prognosis in colorectal cancer | journal = BMC Gastroenterology | volume = 13 | pages = 126 | date = August 2013 | pmid = 23937454 | pmc = 3751151 | doi = 10.1186/1471-230X-13-126 }}</ref><ref>{{cite journal | vauthors = Frisone P, Pradella D, Di Matteo A, Belloni E, Ghigna C, Paronetto MP | title = SAM68: Signal Transduction and RNA Metabolism in Human Cancer | journal = BioMed Research International | volume = 2015 | pages = 528954 | date = 26 July 2015 | pmid = 26273626 | pmc = 4529925 | doi = 10.1155/2015/528954 | doi-access = free }}</ref> | ट्यूमर के विकास में महत्वपूर्ण भूमिका निभाने के लिए आरबीपी उभर रहे हैं।<ref name="Human Cancers 2017">{{cite journal | vauthors = Wang ZL, Li B, Luo YX, Lin Q, Liu SR, Zhang XQ, Zhou H, Yang JH, Qu LH | display-authors = 6 | title = मानव कैंसर में आरएनए-बाध्यकारी प्रोटीन का व्यापक जीनोमिक लक्षण वर्णन| journal = Cell Reports | volume = 22 | issue = 1 | pages = 286–298 | date = January 2018 | pmid = 29298429 | doi = 10.1016/j.celrep.2017.12.035 | doi-access = free }}</ref> सैकड़ों आरबीपी मानव कैंसर में स्पष्ट रूप से विकृत हैं, और इस प्रकार सामान्य ऊतकों से संबंधित ट्यूमर में प्रमुख गिरावट दिखाते हैं।<ref name="Human Cancers 2017"/>कई आरबीपी विभिन्न प्रकार के कैंसर में भिन्न रूप से अभिव्यक्त होते हैं, उदाहरण के लिए केएचडीआरबीएस1(सैम68),<ref>{{cite journal | vauthors = Bielli P, Busà R, Paronetto MP, Sette C | title = The RNA-binding protein Sam68 is a multifunctional player in human cancer | journal = Endocrine-Related Cancer | volume = 18 | issue = 4 | pages = R91–R102 | date = August 2011 | pmid = 21565971 | doi = 10.1530/ERC-11-0041 | url = http://erc.endocrinology-journals.org/content/18/4/R91 | hdl = 2108/88068 | hdl-access = free }}</ref><ref>{{cite journal | vauthors = Liao WT, Liu JL, Wang ZG, Cui YM, Shi L, Li TT, Zhao XH, Chen XT, Ding YQ, Song LB| display-authors = 6 | title = High expression level and nuclear localization of Sam68 are associated with progression and poor prognosis in colorectal cancer | journal = BMC Gastroenterology | volume = 13 | pages = 126 | date = August 2013 | pmid = 23937454 | pmc = 3751151 | doi = 10.1186/1471-230X-13-126 }}</ref><ref>{{cite journal | vauthors = Frisone P, Pradella D, Di Matteo A, Belloni E, Ghigna C, Paronetto MP | title = SAM68: Signal Transduction and RNA Metabolism in Human Cancer | journal = BioMed Research International | volume = 2015 | pages = 528954 | date = 26 July 2015 | pmid = 26273626 | pmc = 4529925 | doi = 10.1155/2015/528954 | doi-access = free }}</ref> ईएलएवीएल1 (हूर),<ref>{{cite journal | vauthors = Abdelmohsen K, Gorospe M | title = एचयूआर द्वारा कैंसर लक्षणों का पोस्टट्रांसक्रिप्शनल विनियमन| journal = Wiley Interdisciplinary Reviews: RNA | volume = 1 | issue = 2 | pages = 214–29 | date = 1 September 2010 | pmid = 21935886 | pmc = 3808850 | doi = 10.1002/wrna.4 }}</ref><ref>{{cite journal | vauthors = Wang J, Guo Y, Chu H, Guan Y, Bi J, Wang B | title = कैंसर की प्रगति, उपचार प्रतिक्रियाओं और पूर्वानुमान में आरएनए-बाध्यकारी प्रोटीन एचयूआर के कई कार्य| journal = International Journal of Molecular Sciences | volume = 14 | issue = 5 | pages = 10015–41 | date = May 2013 | pmid = 23665903 | pmc = 3676826 | doi = 10.3390/ijms140510015 | doi-access = free }}</ref> एफएक्सआर1<ref>{{cite journal | vauthors = Qian J, Hassanein M, Hoeksema MD, Harris BK, Zou Y, Chen H, Lu P, Eisenberg R, Wang J, Espinosa A, Ji X, Harris FT, Rahman SM, Massion PP | display-authors = 6 | title = The RNA binding protein FXR1 is a new driver in the 3q26-29 amplicon and predicts poor prognosis in human cancers | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 112 | issue = 11 | pages = 3469–74 | date = March 2015 | pmid = 25733852 | pmc = 4371932 | doi = 10.1073/pnas.1421975112 | bibcode = 2015PNAS..112.3469Q | doi-access = free }}</ref> और यूएचएमके1.<ref>{{Cite journal|last1=Feng|first1=Xing|last2=Ma|first2=Dong|last3=Zhao|first3=Jiabao|last4=Song|first4=Yongxi|last5=Zhu|first5=Yuekun|last6=Zhou|first6=Qingxin|last7=Ma|first7=Fei|last8=Liu|first8=Xing|last9=Zhong|first9=Mengya|last10=Liu|first10=Yu|last11=Xiong|first11=Yubo|date=2020-03-02|title=UHMK1 रीप्रोग्रामिंग न्यूक्लियोटाइड चयापचय के माध्यम से गैस्ट्रिक कैंसर की प्रगति को बढ़ावा देता है|journal=The EMBO Journal|volume=39|issue=5|pages=e102541|doi=10.15252/embj.2019102541|issn=1460-2075|pmc=7049804|pmid=31975428}}</ref> कुछ आरबीपी के लिए, अभिव्यक्ति में परिवर्तन कॉपी संख्या भिन्नता (सीएनवी) से संबंधित है, उदाहरण के लिए कोलोरेक्टल कैंसर कोशिकाओं में बीवाईएसएल का सीएनवी लाभ<ref name="Human Cancers 2017"/>और ईएसआरपी1, ब्रेस्ट कैंसर में सीईएलएफ3, लिवर कैंसर में आरजीएम24, फेफड़ों के कैंसर में आईजीएफ2बीपी2, आईजीएफ2बीपी3 या फेफड़ों के कैंसर में केएचडीआरबीएस2 की सीएनवी हानियाँ हैं।<ref><nowiki>{{cite journal | vauthors = Sebestyén E, Singh B, Miñana B, Pagès A, Mateo F, Pujana MA, Valcárcel J, Eyras E | display-authors = 6 | title = कई ट्यूमर में जीनोम और ट्रांसक्रिपटोम परिवर्तन के बड़े पैमाने पर विश्लेषण ने उपन्यास कैंसर-संबंधित स्प्लिसिंग नेटवर्क का खुलासा किया| journal = Genome Research | volume = 26 | issue = 6 | pages = 732–44 | date = June 2016 | pmid = 27197215 | pmc = 4889968 | doi = 10.1101/gr.199935.115 }<</nowiki></ref> इस प्रकार इन आरबीपीपर प्रोटीन को प्रभावित करने वाले उत्परिवर्तन के कारण कुछ अभिव्यक्ति परिवर्तन होते हैं, उदाहरण के लिए एनएसयूएन6, जेडसी3एच13, ईएलएसी1, [[RBMS3|आरजीएमएस3]], और जेडजीपीएटी, एसएफ3बी1, एसआरएसएफ2, आरजीएम10, यू2एएफ1, एसएफ3बी1, पीपीआरसी1, आरजीएमएक्सएल1, एचएनआरएनपीसीएल1 आदि।<ref name="Human Cancers 2017"/><ref>सेबेस्टियन 732-744</ref><ref>{{cite journal | vauthors = Yoshida K, Sanada M, Shiraishi Y, Nowak D, Nagata Y, Yamamoto R, Sato Y, Sato-Otsubo A, Kon A, Nagasaki M, Chalkidis G, Suzuki Y, Shiosaka M, Kawahata R, Yamaguchi T, Otsu M, Obara N, Sakata-Yanagimoto M, Ishiyama K, Mori H, Nolte F, Hofmann WK, Miyawaki S, Sugano S, Haferlach C, Koeffler HP, Shih LY, Haferlach T, Chiba S, Nakauchi H, Miyano S, Ogawa S | display-authors = 6 | title = माइलोडिसप्लासिया में स्प्लिसिंग मशीनरी का बार-बार मार्ग परिवर्तन| journal = Nature | volume = 478 | issue = 7367 | pages = 64–9 | date = September 2011 | pmid = 21909114 | doi = 10.1038/nature10496 | bibcode = 2011Natur.478...64Y | s2cid = 4429386 }}</ref><ref>{{cite journal | vauthors = Imielinski M, Berger AH, Hammerman PS, Hernandez B, Pugh TJ, Hodis E, Cho J, Suh J, Capelletti M, Sivachenko A, Sougnez C, Auclair D, Lawrence MS, Stojanov P, Cibulskis K, Choi K, de Waal L, Sharifnia T, Brooks A, Greulich H, Banerji S, Zander T, Seidel D, Leenders F, Ansén S, Ludwig C, Engel-Riedel W, Stoelben E, Wolf J, Goparju C, Thompson K, Winckler W, Kwiatkowski D, Johnson BE, Jänne PA, Miller VA, Pao W, Travis WD, Pass HI, Gabriel SB, Lander ES, Thomas RK, Garraway LA, Getz G, Meyerson M | display-authors = 6 | title = बड़े पैमाने पर समानांतर अनुक्रमण के साथ फेफड़े के एडेनोकार्सिनोमा के हॉलमार्क का मानचित्रण करना| language = en | journal = Cell | volume = 150 | issue = 6 | pages = 1107–20 | date = September 2012 | pmid = 22980975 | pmc = 3557932 | doi = 10.1016/j.cell.2012.08.029 }}</ref><ref>{{cite journal | vauthors = Ellis MJ, Ding L, Shen D, Luo J, Suman VJ, Wallis JW, Van Tine BA, Hoog J, Goiffon RJ, Goldstein TC, Ng S, Lin L, Crowder R, Snider J, Ballman K, Weber J, Chen K, Koboldt DC, Kandoth C, Schierding WS, McMichael JF, Miller CA, Lu C, Harris CC, McLellan MD, Wendl MC, DeSchryver K, Allred DC, Esserman L, Unzeitig G, Margenthaler J, Babiera GV, Marcom PK, Guenther JM, Leitch M, Hunt K, Olson J, Tao Y, Maher CA, Fulton LL, Fulton RS, Harrison M, Oberkfell B, Du F, Demeter R, Vickery TL, Elhammali A, Piwnica-Worms H, McDonald S, Watson M, Dooling DJ, Ota D, Chang LW, Bose R, Ley TJ, Piwnica-Worms D, Stuart JM, Wilson RK, Mardis ER | display-authors = 6 | title = संपूर्ण-जीनोम विश्लेषण एरोमाटेज निषेध के लिए स्तन कैंसर की प्रतिक्रिया की सूचना देता है| journal = Nature | volume = 486 | issue = 7403 | pages = 353–60 | date = June 2012 | pmid = 22722193 | pmc = 3383766 | doi = 10.1038/nature11143 | bibcode = 2012Natur.486..353E }}</ref> कई अध्ययनों ने आरबीपी की अभिव्यक्ति में इस परिवर्तन को कैंसर में असामान्य वैकल्पिक विभाजन से संबंधित बताया है।<ref>सेबस्टीन 732–744</ref><ref>{{cite journal | vauthors = David CJ, Manley JL | title = Alternative pre-mRNA splicing regulation in cancer: pathways and programs unhinged | journal = Genes & Development | volume = 24 | issue = 21 | pages = 2343–64 | date = November 2010 | pmid = 21041405 | pmc = 2964746 | doi = 10.1101/gad.1973010 }}</ref><ref>{{cite journal | vauthors = Fredericks AM, Cygan KJ, Brown BA, Fairbrother WG | title = RNA-Binding Proteins: Splicing Factors and Disease | journal = Biomolecules | volume = 5 | issue = 2 | pages = 893–909 | date = May 2015 | pmid = 25985083 | pmc = 4496701 | doi = 10.3390/biom5020893 | doi-access = free }}</ref> | ||
== वर्तमान शोध == | == वर्तमान शोध == | ||
[[File:Protein CIRBP PDB 1x5s.png|thumb|right|alt=CIRBP.| [[सीआईआरबीपी]]: सीआईआरबीपी प्रोटीन की संरचना।]]चूंकि आरएनए-बाध्यकारी प्रोटीन कई सेलुलर कार्यों पर महत्वपूर्ण नियंत्रण रखते हैं, वे कई शोधकर्ताओं के लिए जांच का | [[File:Protein CIRBP PDB 1x5s.png|thumb|right|alt=CIRBP.| [[सीआईआरबीपी]]: सीआईआरबीपी प्रोटीन की संरचना।]]चूंकि आरएनए-बाध्यकारी प्रोटीन कई सेलुलर कार्यों पर महत्वपूर्ण नियंत्रण रखते हैं, वे कई शोधकर्ताओं के लिए जांच का लोकप्रिय क्षेत्र रहे हैं। जैविक क्षेत्र में इसके महत्व के कारण, हाल ही में आरएनए-बाध्यकारी प्रोटीन की क्षमता के बारे में कई खोजों का अनावरण किया गया है।<ref name="Stefl" /> इस प्रकार आरएनए-बाध्यकारी प्रोटीनों की प्रायोगिक पहचान में वर्तमान समय में विकास ने आरएनए-बाध्यकारी प्रोटीनों की संख्या में महत्वपूर्ण रूप से वृद्धि की है<ref>{{cite journal | vauthors = Conrad T, Albrecht AS, de Melo Costa VR, Sauer S, Meierhofer D, Ørom UA | title = मानव कोशिका नाभिक का सीरियल इंटरएक्टिव कैप्चर| journal = Nature Communications | volume = 7 | pages = 11212 | date = April 2016 | pmid = 27040163 | pmc = 4822031 | doi = 10.1038/ncomms11212 | bibcode = 2016NatCo...711212C }}</ref><ref>{{cite journal | vauthors = Castello A, Fischer B, Eichelbaum K, Horos R, Beckmann BM, Strein C, Davey NE, Humphreys DT, Preiss T, Steinmetz LM, Krijgsveld J, Hentze MW | display-authors = 6 | title = स्तनधारी एमआरएनए-बाध्यकारी प्रोटीन के एटलस से आरएनए जीव विज्ञान में अंतर्दृष्टि| journal = Cell | volume = 149 | issue = 6 | pages = 1393–406 | date = June 2012 | pmid = 22658674 | doi = 10.1016/j.cell.2012.04.031 | doi-access = free }}</ref><ref>{{cite journal | vauthors = Baltz AG, Munschauer M, Schwanhäusser B, Vasile A, Murakawa Y, Schueler M, Youngs N, Penfold-Brown D, Drew K, Milek M, Wyler E, Bonneau R, Selbach M, Dieterich C, Landthaler M | display-authors = 6 | title = प्रोटीन-कोडिंग ट्रांस्क्रिप्ट पर एमआरएनए-बाउंड प्रोटिओम और इसकी वैश्विक अधिभोग प्रोफ़ाइल| journal = Molecular Cell | volume = 46 | issue = 5 | pages = 674–90 | date = June 2012 | pmid = 22681889 | doi = 10.1016/j.molcel.2012.05.021 | doi-access = free }}</ref> | ||
आरएनए- | आरएनए-बाध्यकारी प्रोटीन सैम68 आरएनए [[उपापचय]] के स्थानिक और लौकिक कंपार्टमेंटलाइज़ेशन को नियंत्रित करता है ताकि डेंड्राइट्स में उचित [[निष्कर्ष]] फलन प्राप्त किया जा सके। सैम68 की हानि के परिणामस्वरूप असामान्य पोस्टट्रांसक्रिप्शनल रेगुलेशन होता है और अंतत: नाजुक एक्स-जुड़े कंपकंपी / गतिभंग सिंड्रोम जैसे न्यूरोलॉजिकल विकारों की ओर जाता है। इस प्रकार सैम68 को एमआरएनए एन्कोडिंग β-एक्टिन के साथ बातचीत करने के लिए पाया गया, जो अपने [[साइटोस्केलेटल]] घटकों के साथ डेंड्राइटिक स्पाइन के सिनैप्टिक गठन को नियंत्रित करता है। इसलिए इस प्रकार सैम68 पोस्टसिनेप्टिक β-एक्टिन एमआरएनए चयापचय के नियंत्रण के माध्यम से सिनैप्स संख्या को विनियमित करने में महत्वपूर्ण भूमिका निभाता है।<ref>{{cite journal | vauthors = Klein ME, Younts TJ, Castillo PE, Jordan BA | title = RNA-binding protein Sam68 controls synapse number and local β-actin mRNA metabolism in dendrites | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 110 | issue = 8 | pages = 3125–30 | date = February 2013 | pmid = 23382180 | pmc = 3581878 | doi = 10.1073/pnas.1209811110 | bibcode = 2013PNAS..110.3125K | doi-access = free }}</ref> | ||
[[File:Protein ACTB PDB 1atn.png|thumb|left|alt=Beta-एक्टिन.| बीटा-एक्टिन: ACTB प्रोटीन की संरचना।]]न्यूरॉन-विशिष्ट सीईएलएफ परिवार आरएनए-बाध्यकारी प्रोटीन यूएनसी-75 विशेष रूप से सी एलिगेंस के न्यूरोनल कोशिकाओं में एक्सॉन 7ए चयन के लिए अपने तीन आरएनए मान्यता रूपांकनों के माध्यम से यूयूजीयूयूजीयूजीयूयूजीयू एमआरएनए खिंचाव को बांधता है। जैसा कि एक्सॉन 7ए को गैर-न्यूरोनल कोशिकाओं में कमजोर स्थलों के कारण छोड़ दिया गया है, यूएनसी-75 को विशेष रूप से एक्सॉन 7a और एक्सॉन 8 के बीच केवल न्यूरोनल कोशिकाओं में सक्रिय करने के लिए पाया गया था।<ref>{{cite journal | vauthors = Kuroyanagi H, Watanabe Y, Hagiwara M | title = CELF family RNA-binding protein UNC-75 regulates two sets of mutually exclusive exons of the unc-32 gene in neuron-specific manners in Caenorhabditis elegans | journal = PLOS Genetics | volume = 9 | issue = 2 | pages = e1003337 | year = 2013 | pmid = 23468662 | pmc = 3585155 | doi = 10.1371/journal.pgen.1003337 | editor1-last = Blumenthal | editor1-first = Tom }}</ref> | |||
कोल्ड इंड्यूसिबल आरएनए बाध्यकारी प्रोटीन सीआईआरबीपी शॉर्ट वेवलेंथ [[पराबैंगनी प्रकाश]], [[ हाइपोक्सिया (चिकित्सा) |हाइपोक्सिया (चिकित्सा)]] और [[ अल्प तपावस्था |अल्प तपावस्था]] सहित विभिन्न प्रकार के सेलुलर तनावों का सामना करने पर सेलुलर प्रतिक्रिया को नियंत्रित करने में भूमिका निभाता है। इस शोध से सूजन के साथ रोग राज्यों के जुड़ाव के संभावित प्रभाव सामने आए हैं।<ref>{{cite journal | vauthors = Brochu C, Cabrita MA, Melanson BD, Hamill JD, Lau R, Pratt MA, McKay BC | title = NF-κB-dependent role for cold-inducible RNA binding protein in regulating interleukin 1β | journal = PLOS ONE | volume = 8 | issue = 2 | pages = e57426 | year = 2013 | pmid = 23437386 | pmc = 3578848 | doi = 10.1371/journal.pone.0057426 | editor1-last = Gallouzi | bibcode = 2013PLoSO...857426B | editor1-first = Imed Eddine | doi-access = free }}</ref> इस प्रकार आरएनए-बाध्यकारी प्रोटीन एसएलआर1 के सेरीन-आर्जिनिन परिवार को [[कैनडीडा अल्बिकन्स]] में ध्रुवीकृत वृद्धि पर अत्यधिक नियंत्रण पाया गया था। इस प्रकार चूहों में एसएलआर1 म्यूटेशन के परिणामस्वरूप फिलामेंटेशन कम हो जाता है और [[अन्तःस्तरीय कोशिका]] और एंडोथेलियल [[उपकला कोशिका]] की हानि कम हो जाती है जो एसएलआर1 जंगली प्रकार के उपभेदों की तुलना में विस्तारित जीवित रहने की दर की ओर जाता है। इसलिए, इस शोध से पता चलता है कि एसआर-जैसे प्रोटीन एसएलआर1 सी. अल्बिकैंस में हाइपल गठन और विषाणु को भड़काने में भूमिका निभाता है।<ref>{{cite journal | vauthors = Ariyachet C, Solis NV, Liu Y, Prasadarao NV, Filler SG, McBride AE | title = एसआर-जैसे आरएनए-बाइंडिंग प्रोटीन एसएलआर1 कैंडिडा एल्बीकैंस फिलामेंटेशन और विषाणु को प्रभावित करता है| journal = Infection and Immunity | volume = 81 | issue = 4 | pages = 1267–76 | date = April 2013 | pmid = 23381995 | pmc = 3639594 | doi = 10.1128/IAI.00864-12 }}</ref> | |||
== यह भी देखें == | == यह भी देखें == | ||
* डीएनए बाध्यकारी प्रोटीन | * डीएनए बाध्यकारी प्रोटीन | ||
| Line 112: | Line 81: | ||
== बाहरी संबंध == | == बाहरी संबंध == | ||
{{Commons category|RNA-binding proteins}} | {{Commons category|RNA-binding proteins}} | ||
* [https://web.archive.org/web/20110222111721/http://starbase.sysu.edu.cn/ starBase platform]: a platform for decoding binding sites of | * [https://web.archive.org/web/20110222111721/http://starbase.sysu.edu.cn/ starBase platform]: a platform for decoding binding sites of आरएनए binding proteins (RBPs) from large-scale [[CLIP-Seq]] (HITS-CLIP, PAR-CLIP, iCLIP, CLASH) datasets. | ||
* [http://rbpdb.ccbr.utoronto.ca// RBPDB database]: a database of | * [http://rbpdb.ccbr.utoronto.ca// RBPDB database]: a database of आरएनए binding proteins. | ||
* [http://rnabiology.ircm.qc.ca/oRNAment/ | * [http://rnabiology.ircm.qc.ca/oRNAment/ oआरएनएment]: a database of putative RBP binding site instances in both coding and non-coding आरएनए in various species. | ||
* [https://attract.cnic.es/ ATtRACt database]: a database of | * [https://attract.cnic.es/ ATtRACt database]: a database of आरएनए binding proteins and associated motifs. | ||
* [[SplicedAid-F]]: a database of hand -cureted human | * [[SplicedAid-F]]: a database of hand -cureted human आरएनए binding proteins database. | ||
* [http://bioinfo3d.cs.tau.ac.il/RsiteDB/ RsiteDB]: | * [http://bioinfo3d.cs.tau.ac.il/RsiteDB/ RsiteDB]: आरएनए binding site database | ||
* [http://sparks-lab.org/yueyang/server/SPOT-Seq-RNA/ SPOT-Seq- | * [http://sparks-lab.org/yueyang/server/SPOT-Seq-RNA/ SPOT-Seq-आरएनए]: Template-based prediction of आरएनए binding proteins and their complex structures. | ||
* [http://sparks-lab.org/yueyang/server/SPOT-Struct-RNA/ SPOT-Struct- | * [http://sparks-lab.org/yueyang/server/SPOT-Struct-RNA/ SPOT-Struct-आरएनए]: आरएनए binding proteins prediction from 3D structures. | ||
*[https://www.encodeproject.org/ ENCODE Project]: A collection of genomic datasets (i.e. | *[https://www.encodeproject.org/ ENCODE Project]: A collection of genomic datasets (i.e. आरएनए Bind-n-seq, eCLIP, RBP targeted shआरएनए आरएनए-seq) for RBPs | ||
*[http://rnabiology.ircm.qc.ca/RBPImage/ RBP Image Database]: Images showing the cellular localization of | *[http://rnabiology.ircm.qc.ca/RBPImage/ RBP Image Database]: Images showing the cellular localization of आरबीपीin cells | ||
*[https://scbb.ihbt.res.in/RBPSpot/ RBPSpot Software]: A Deep-Learning based highly accurate software to detect RBP- | *[https://scbb.ihbt.res.in/RBPSpot/ RBPSpot Software]: A Deep-Learning based highly accurate software to detect RBP-आरएनए interaction. It also provides a module to build new RBP-आरएनए interaction models. | ||
| Line 130: | Line 99: | ||
{{RNA-binding proteins}} | {{RNA-binding proteins}} | ||
{{Ribonucleoproteins}} | {{Ribonucleoproteins}} | ||
[[Category: | [[Category:Articles with hatnote templates targeting a nonexistent page]] | ||
[[Category:CS1 English-language sources (en)]] | |||
[[Category:Collapse templates]] | |||
[[Category:Commons category link is locally defined]] | |||
[[Category:Created On 15/06/2023]] | [[Category:Created On 15/06/2023]] | ||
[[Category:Lua-based templates]] | |||
[[Category:Machine Translated Page]] | |||
[[Category:Navigational boxes| ]] | |||
[[Category:Navigational boxes without horizontal lists]] | |||
[[Category:Pages with script errors]] | |||
[[Category:Sidebars with styles needing conversion]] | |||
[[Category:Template documentation pages|Documentation/doc]] | |||
[[Category:Templates Vigyan Ready]] | |||
[[Category:Templates generating microformats]] | |||
[[Category:Templates that add a tracking category]] | |||
[[Category:Templates that are not mobile friendly]] | |||
[[Category:Templates that generate short descriptions]] | |||
[[Category:Templates using TemplateData]] | |||
[[Category:Wikipedia metatemplates]] | |||
[[Category:आरएनए-बाध्यकारी प्रोटीन| आरएनए-बाध्यकारी प्रोटीन ]] | |||
[[Category:कोशिका विज्ञान]] | |||
Latest revision as of 14:17, 5 July 2023
आरएनए बाध्यकारी प्रोटीन अधिकांशतः आरबीपी के रूप में ऐसे संक्षिप्त प्रोटीन होते हैं जो आपस में एक-दूसरे से फंसे हुए आरएनए से जुड़ते हैं,[1] इस प्रकार कोशिकाएं जीव विज्ञान में और राइबो न्यूक्लियोप्रोटीन कॉम्प्लेक्स बनाने में भाग लेते हैं।
आरबीपी में विभिन्न संरचनात्मक रूपांकन होते हैं, जैसे कि आरएनए मान्यता वाले मूल भाव (आरआरएम), डीएसआरएनए बाध्यकारी डोमेन, जिंक फिंगर और [2][3] इसमें अन्य कोशिका द्रव्य और कोशिका केंद्रक प्रोटीन हैं। चूंकि अधिकांश परिपक्व आरएनए नाभिक से अपेक्षाकृत तेज़ी से निर्यात किए जाते हैं, नाभिक में अधिकांश आरबीपी प्रोटीन और अग्रदूत एमआरएनए के क्षेत्रों के रूप में उपस्थित होते हैं। इस प्रकार पूर्व-एमआरएनए को विषम राइबोन्यूक्लियोप्रोटीन कण (एचएनआरएनपी) कहा जाता है।
विभिन्न सेलुलर प्रक्रियाओं में आरबीपी की महत्वपूर्ण भूमिका होती है जैसे: सेलुलर फलन, परिवहन और स्थानीयकरण इत्यादि। ये मुख्यतः विशेष रूप से आरएनए के पोस्ट-ट्रांसक्रिप्शनल नियंत्रण में प्रमुख भूमिका निभाते हैं, जैसे: आरएनए स्पिलिंग, पॉलीएडेनाइलेशन, एमआरएनए स्थिरीकरण, एमआरएनए उपकोशिकीय स्थानीयकरण और अनुवाद (जीव विज्ञान) इत्यादि। इस प्रकार यूकेरियोट कोशिकाएं विविध आरबीपी को अद्वितीय आरएनए-बाध्यकारी गतिविधि और प्रोटीन-प्रोटीन इंटरैक्शन के साथ व्यक्त करती हैं। यूकेरियोटिक आरबीपी डाटाबेस (यूआरबीपीडीबी) के अनुसार, मनुष्यों में 2961 जीन एन्कोडिंग आरबीपी हैं। विकास के समय, इंट्रोन्स की संख्या में वृद्धि के साथ आरबीपी की विविधता बहुत बढ़ गई। इस कारण विभिन्न प्रकार की विविधताओं के कारण सक्षम यूकेरियोटिक कोशिकाएं विभिन्न व्यवस्थाओं में आरएनए एक्सॉन का उपयोग करने के लिए, प्रत्येक आरएनए के लिए अद्वितीय आरएनपी (राइबोन्यूक्लियोप्रोटीन) को जन्म देती हैं। यद्यपि इस प्रकार जीन अभिव्यक्ति में पोस्ट-ट्रांसक्रिप्शनल विनियमन में आरबीपी की महत्वपूर्ण भूमिका है, अपेक्षाकृत कुछ आरबीपी का व्यवस्थित रूप से अध्ययन किया गया है। अब यह स्पष्ट हो गया है कि आरएनए-आरबीपी इंटरैक्शन जीवों के बीच कई जैविक प्रक्रियाओं में महत्वपूर्ण भूमिका निभाते हैं।[4][5][6]
संरचना
कई आरबीपी में मॉड्यूलर संरचनाएं होती हैं और केवल कुछ विशिष्ट मौलिक डोमेन के कई दोहराव से बने होते हैं जिनमें अधिकांशतः सीमित अनुक्रम होते हैं। विविधता की आवश्यकता को पूरा करने के लिए इन अनुक्रमों को अलग-अलग संयोजनों में व्यवस्थित किया जाता है। इस प्रकार विशिष्ट आरएनए की विशिष्ट प्रोटीन की मान्यता इन कुछ मौलिक डोमेन के पुनर्व्यवस्था के माध्यम से विकसित हुई है। प्रत्येक मूल डोमेन आरएनए को पहचानता है, अपितु इनमें से कई प्रोटीनों को कार्य करने के लिए कई सामान्य डोमेन में से की कई प्रतियों की आवश्यकता होती है।[2]
विविधता
जैसे ही परमाणु आरएनए आरएनए पोलीमरेज़ से निकलता है, आरएनए ट्रांसक्रिप्ट तुरंत आरएनए-बाध्यकारी प्रोटीन से आच्छादित हो जाते हैं जो आरएनए चयापचय के हर पहलू को नियंत्रित करते हैं और आरएनए जैवजनन, परिपक्वता, परिवहन, सेलुलर स्थानीयकरण और स्थिरता सहित कार्य करते हैं। इस प्रकार सभी आरबीपी आरएनए को बांधते हैं, चूंकि वे विभिन्न आरएनए-अनुक्रम विशिष्टताओं और समानता के साथ ऐसा करते हैं, जो आरबीपी को उनके लक्ष्यों और कार्यों के रूप में विविध होने की अनुमति देता है।[5] इन लक्ष्यों में एमआरएनए सम्मिलित होते हैं, जो इस प्रकार प्रोटीन के लिए कोड करता है, साथ ही कई कार्यात्मक गैर-कोडिंग आरएनए भी सम्मिलित हैं। एनसीआरएनए लगभग हमेशा राइबोन्यूक्लियोप्रोटीन कॉम्प्लेक्स के रूप में कार्य करते हैं न कि नग्न आरएनए के रूप में कार्य करते हैं। इस प्रकार इन गैर-कोडिंग आरएनए में माइक्रोआरएनए, छोटे हस्तक्षेप करने वाले आरएनए (siआरएनए), साथ ही स्प्लिसेसोमल छोटे परमाणु आरएनए (एसएनआरएनए) सम्मिलित किया जाता हैं।[7]
फलन
आरएनए प्रसंस्करण और संशोधन
वैकल्पिक विभाजन
वैकल्पिक विभाजन वह तंत्र है जिसके द्वारा जीन से परिपक्व एमआरएनए संदेशवाहक आरएनए के विभिन्न रूप उत्पन्न होते हैं। यह नियामक तंत्र है जिसके द्वारा एक्सॉन को एमआरएनए में सम्मिलित करने में भिन्नता से अधिक संबंधित प्रोटीन के उत्पादन की ओर ले जाती है, इस प्रकार संभावित जीनोमिक आउटपुट का विस्तार होता है। आरबीपी इस प्रक्रिया के नियमन में व्यापक रूप से कार्य करते हैं। कुछ बाध्यकारी प्रोटीन जैसे कि न्यूरोनल विशिष्ट आरएनए-बाध्यकारी प्रोटीन, अर्थात् नोवा1, आरएनए (वाईसीएवाई जहां वाई पाइरीमिडीन, यू या सी को इंगित करता है) में विशिष्ट अनुक्रम को पहचानने और बाध्य करके एचएनआरएनए के उपसमूह के वैकल्पिक विभाजन को नियंत्रित करता है।[5] ये प्रोटीन फिर इस लक्ष्य स्थल पर स्प्लिसोमल प्रोटीन की भर्ती करते हैं। एसआर प्रोटीन एसएनआरएनपी की भर्ती के माध्यम से वैकल्पिक विभाजन में अपनी भूमिका के लिए भी जाने जाते हैं, जो कि यू1 एसएनआरएनपी और यू2एएफ एसएनआरएनपी नामक स्पलाइसोसम बनाते हैं। चूंकि, आरबीपीभी इसका ही भाग हैं। इस प्रकार स्प्लिसोम एसएनआरएनए एसआर प्रोटीन सबयूनिट्स का जटिल है और यांत्रिक एजेंट के रूप में कार्य करता है जो इंट्रॉन को हटाता है और फ्लैंकिंग एक्सॉन को लिगेट करता है।[7] इस प्रकार कोर स्प्लिससम कॉम्प्लेक्स के अतिरिक्त, आरबीपी सिस-एक्टिंग आरएनए तत्वों की साइटों से भी जुड़ते हैं जो एक्सॉन को सम्मिलित करने या विभाजन के समय बहिष्करण को प्रभावित करते हैं। इन साइटों को एक्सोनिक स्प्लिसिंग एन्हांसर (ईएसई), एक्सोनिक स्प्लिसिंग साइलेंसर (ईएसएस), इंट्रोनिक स्प्लिसिंग एनहांसर (आईएसई) और इंट्रोनिक स्प्लिसिंग साइलेंसर (आईएसएस) के रूप में संदर्भित किया जाता है और उनके बंधन के स्थान के आधार पर, आरबीपी स्प्लिसिंग साइलेंसर या एन्हांसर के रूप में कार्य करते हैं।[8]
आरएनए संपादन
आरएनए संपादन के सबसे व्यापक रूप से अध्ययन किए गए रूप में एडीएआर प्रोटीन सम्मिलित है। यह प्रोटीन आरएनए के न्यूक्लियोटाइड सामग्री को बदलकर एमआरएनए ट्रांसक्रिप्ट के पोस्ट-ट्रांसक्रिप्शनल संशोधन के माध्यम से कार्य करता है। इस प्रकार यह एडीएआर द्वारा उत्प्रेरित एंजाइमिक प्रतिक्रिया में एडेनोसाइन को आइनोसीन में परिवर्तित करने के माध्यम से किया जाता है। यह प्रक्रिया जीनोम द्वारा एन्कोड किए गए आरएनए अनुक्रम को प्रभावी विधि से परिवर्तित हो जाती है और जीन उत्पादों की विविधता को बढ़ाती है। इस प्रकार अधिकांश आरएनए संपादन आरएनए के गैर-कोडिंग क्षेत्रों पर होता है; चूंकि, कुछ प्रोटीन-एन्कोडिंग आरएनए ट्रांसक्रिप्शंस को संपादन के अधीन दिखाया गया है, जिसके परिणामस्वरूप उनके प्रोटीन के अमीनो एसिड अनुक्रम में अंतर होता है। इसका उदाहरण ग्लूटामेट रिसेप्टर एमआरएनए है जहां ग्लूटामाइन को आर्गिनिन में परिवर्तित किया जाता है जिससे प्रोटीन की कार्यक्षमता में परिवर्तन होता है।[5]
पॉलीएडेनिलेशन
पॉलीएडेनाइलेशन तीन प्रमुख अअनुवादित क्षेत्र के भीतर एएयूएएए अनुक्रम के डाउनस्ट्रीम लगभग 20 आधारों के आरएनए प्रतिलेख में एडिनाइलेट अवशेषों की एक टेल के अतिरिक्त प्राप्त होती है। इस प्रकार एमआरएनए के पॉलीएडेनाइलेशन का इसके परमाणु परिवहन, अनुवाद दक्षता और स्थिरता पर गहरा प्रभाव पड़ता है। इन सभी के साथ-साथ पॉलीएडेनाइलेशन की प्रक्रिया विशिष्ट आरबीपी के बंधन पर निर्भर करती है। इस प्रकार कुछ अपवादों के साथ सभी यूकेरियोटिक एमआरएनए को लगभग 200 न्यूक्लियोटाइड्स की 3' पॉली (ए) टेल प्राप्त करने के लिए संसाधित किया जाता है। इस प्रकार इस प्रक्रिया में आवश्यक प्रोटीन क्षेत्रों में से सीपीएसएफ है। सीपीएसएफ 3' टेल (एएयूएएए) अनुक्रम से जुड़ता है और साथ में पॉली (ए)पॉली (ए) -बाध्यकारी प्रोटीन नामक अन्य प्रोटीन के साथ, पॉली (ए) पोलीमरेज़ की गतिविधि को भरती और उत्तेजित करता है। पॉली (ए) पोलीमरेज़ अपने आप में निष्क्रिय है और इन अन्य प्रोटीनों को ठीक से कार्य करने के लिए बाध्य करने की आवश्यकता है।[5]
निर्यात
प्रसंस्करण पूरा होने के पश्चात एमआरएनए को सेल न्यूक्लियस से साइटोप्लाज्म तक ले जाने की आवश्यकता होती है। यह तीन-चरण की प्रक्रिया है जिसमें नाभिक में कार्गो-वाहक क्षेत्र की पीढ़ी सम्मिलित है, जिसके बाद परमाणु छिद्र क्षेत्र के माध्यम से क्षेत्र का स्थानांतरण होता है और अंत में कार्गो को साइटोप्लाज्म में छोड़ दिया जाता है। इस प्रकार वाहक को बाद में पुनर्नवीनीकरण किया जाता है। टैप/एनएक्सएफ1:p15 हेटेरोडिमर को एमआरएनए निर्यात में प्रमुख खिलाड़ी माना जाता है। ज़ेनोपस लाविस मेंढकों में टैप की ओवर-एक्सप्रेशन उन प्रतिलेखों के निर्यात को बढ़ाती है जो अन्यथा अक्षम रूप से निर्यात किए जाते हैं। चूंकि टैप को एडेप्टर प्रोटीन की आवश्यकता होती है क्योंकि यह एमआरएनए के साथ सीधे संपर्क करने में असमर्थ है। इस कारण एली/आरईएफ प्रोटीन एमआरएनए भर्ती टीएपी से संपर्क करता है, और बांध कर रखता है।[5]
एमआरएनए स्थानीयकरण
स्थानिक रूप से विनियमित प्रोटीन उत्पादन की अनुमति देकर जीन अभिव्यक्ति के नियमन के लिए एमआरएनए स्थानीयकरण महत्वपूर्ण है। एमआरएनए स्थानीयकरण के माध्यम से प्रोटीन को सेल के अपने इच्छित लक्ष्य स्थल में अनुवादित किया जाता है। प्रारंभिक विकास के समय यह विशेष रूप से महत्वपूर्ण है जब तेजी से सेल दरार अलग-अलग कोशिकाओं को एमआरएनए के विभिन्न संयोजन देते हैं जो बाद में अलग-अलग सेल भाग्य को जन्म दे सकते हैं। आरबीपी इस एमआरएनए के स्थानीयकरण में महत्वपूर्ण हैं जो सुनिश्चित करता है कि प्रोटीन केवल उनके इच्छित क्षेत्रों में अनुवादित हैं। इस प्रकार इनमें से प्रोटीन जेडबीपी1 है। जेडबीपी1 प्रतिलेखन के स्थल पर बीटा एक्टिन एमआरएनए से जुड़ता है और एमआरएनए के साथ साइटोप्लाज्म में चला जाता है। इसके पश्चात यह इस एमआरएनए को कई असममित सेल प्रकारों के लैमेला (कोशिका जीव विज्ञान) क्षेत्र में स्थानांतरित करता है जहां इसका अनुवाद किया जा सकता है।[5] इस प्रकार 2008 में यह प्रस्तावित किया गया था कि एफएमआर1 सुसंस्कृत हिप्पोकैम्पस न्यूरॉन्स के न्यूरोनल डेंड्राइट्स में कई डेंड्राइटिक एमआरएनए के उत्तेजना-प्रेरित स्थानीयकरण में सम्मिलित था।[9] सीए1 हिप्पोकैम्पस न्यूरॉन्स के माइक्रोडिसेक्टेड डेंड्राइट्स में उपस्थित एफएमआरP-बाउंड आरएनए के अधिकांशतः वर्तमान समय के अध्ययनों से भिन्न प्रकार से इसके विरुद्ध एफएमआरपी-रिक्त माउस में स्थानीयकरण में कोई परिवर्तन नहीं हुआ हैं।[10]
अनुवाद
ट्रांसलेशनल विनियमन जीन अभिव्यक्ति को नियंत्रित करने के लिए तीव्र तंत्र प्रदान करता है। इस प्रकार ट्रांसक्रिप्शनल स्तर पर जीन अभिव्यक्ति को नियंत्रित करने के अतिरिक्त, एमआरएनए पहले से ही लिखित है अपितु राइबोसोम की भर्ती नियंत्रित है। जब सिग्नल ट्रांसलेशन को सक्रिय करता है तो यह तेजी से प्रोटीन उत्पन्न करता है। इस प्रकार जेडबीपी1 बी-एक्टिन एमआरएनए के स्थानीयकरण में अपनी भूमिका के अतिरिक्त अनुवाद दीक्षा को अवरुद्ध करके बीटा-एक्टिन एमआरएनए के अनुवाद संबंधी दमन में भी सम्मिलित है। राइबोसोम को ठीक से बाँधने और अनुवाद प्रारंभ करने की अनुमति देने के लिए जेडबीपी1 को एमआरएनए से हटाया जाना चाहिए।[5]
प्रोटीन-आरएनए इंटरैक्शन
आरएनए-बाध्यकारी प्रोटीन अपने अनुक्रमों, संरचनाओं, रूपांकनों और आरएनए संशोधनों को पहचानकर अपने आरएनए लक्ष्यों की अत्यधिक विशिष्ट पहचान प्रदर्शित करते हैं।[11] इस प्रकार आरएनए-बाध्यकारी प्रोटीन के विशिष्ट बंधन उन्हें अपने लक्ष्यों को अलग करने और आरएनए प्रतिलेख की पीढ़ी, परिपक्वता और जीवन काल के नियंत्रण के माध्यम से विभिन्न प्रकार के सेलुलर कार्यों को विनियमित करने की अनुमति देते हैं। यह अंतःक्रिया ट्रांसक्रिप्शन के समय प्रारंभ होती है क्योंकि कुछ आरबीपी गिरावट तक आरएनए के लिए बाध्य रहते हैं, जबकि अन्य आरएनए स्पिलिंग, प्रसंस्करण, परिवहन और स्थानीयकरण को विनियमित करने के लिए केवल क्षणिक रूप से आरएनए से जुड़ते हैं।[12] विभिन्न प्रकार के ऊतकों और जीवों में आरएनए-बाध्यकारी प्रोटीनों के प्रत्यक्ष आरएनए बाध्यकारी साइटों की पहचान करने के लिए क्रॉस-लिंकिंग इम्यूनोप्रिसिपेशन (सीएलआईपी) विधियों का उपयोग किया जाता है। इस खंड में, सबसे व्यापक रूप से अध्ययन किए गए आरएनए-बाध्यकारी डोमेन (आरएनए-रिकग्निशन मोटिफ, डबल-स्ट्रैंडेड आरएनए-बाध्यकारी मोटिफ, जिंक-फिंगर मोटिफ) के तीन वर्गों पर चर्चा की जाती हैं।
आरएनए-रिकग्निशन मोटिफ (आरआरएम)
आरएनए रिकग्निशन मोटिफ, जो सबसे साधारण आरएनए-बाध्यकारी मोटिफ है, 75-85 एमिनो एसिड का छोटा प्रोटीन डोमेन है जो दो α-हेलीकॉप्स के खिलाफ चार-फंसे हुए β-शीट बनाता है। यह मान्यता मूल भाव कई सेलुलर कार्यों में विशेष रूप से एमआरएनए / rआरएनए प्रसंस्करण, विभाजन, अनुवाद विनियमन, आरएनए निर्यात और आरएनए स्थिरता में अपनी भूमिका निभाता है। एनएमआर स्पेक्ट्रोस्कोपी और एक्स - रे क्रिस्टलोग्राफी के माध्यम से आरआरएम की दस संरचनाओं की पहचान की गई है। इस प्रकार ये संरचनाएं आरआरएम की प्रोटीन-आरएनए मान्यता की गहनता को दर्शाती हैं क्योंकि इसमें प्रोटीन-आरएनए इंटरैक्शन के अतिरिक्त आरएनए-आरएनए और प्रोटीन-प्रोटीन इंटरैक्शन सम्मिलित हैं। उनकी जटिलता के अतिरिक्त, सभी दस संरचनाओं में कुछ सामान्य विशेषताएं हैं। सभी आरआरएम की मुख्य प्रोटीन सतहों की चार-फंसे β-शीट को आरएनए के साथ बातचीत करने के लिए पाया गया, जो साधारणतौर पर विशिष्ट तरीके से दो या तीन न्यूक्लियोटाइड्स से संपर्क करता है। इसके अतिरिक्त, अंतर-डोमेन लिंकर और इस प्रकार आरएनए के बीच और यह स्वयं आरआरएम के बीच बातचीत के माध्यम से भिन्नता के प्रति मजबूत आरएनए बंधन संबंध और विशिष्टता प्राप्त की जाती है। आरआरएम की यह नमनीयता बताती है कि आरआरएम सबसे प्रचुर मात्रा में डोमेन क्यों है और यह विभिन्न जैविक कार्यों में महत्वपूर्ण भूमिका क्यों निभाता है।[12]
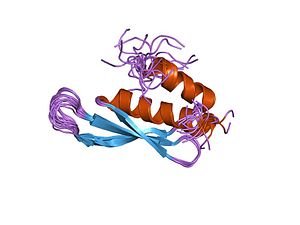
डबल-स्ट्रैंडेड आरएनए-बाध्यकारी मोटिफ
| Double-stranded RNA-binding motif | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| Identifiers | |||||||||
| Symbol | drrm | ||||||||
| Pfam | PF14709 | ||||||||
| Pfam clan | CL0196 | ||||||||
| InterPro | IPR014720 | ||||||||
| CATH | 1di2 | ||||||||
| SCOP2 | 1di2 / SCOPe / SUPFAM | ||||||||
| |||||||||
| Use the Pfam clan for the homologous superfamily. | |||||||||
डबल-स्ट्रैंडेड आरएनए-बाध्यकारी मोटिफ (डीएसआरएम, डीएसआरबीडी), 70-75 एमिनो-एसिड डोमेन, आरएनए प्रसंस्करण, आरएनए सबसेलुलर लोकलाइजेशन, आरएनए हस्तक्षेप, आरएनए संपादन और ट्रांसलेशनल दमन में महत्वपूर्ण भूमिका निभाता है। इस प्रकार 2005 तक हल की गई डोमेन की सभी तीन संरचनाओं में एकत्रित करने वाली विशेषताएँ हैं जो बताती हैं कि कैसे डीएसआरएमएस केवल डीएसडीएनए के अतिरिक्त डीएसआरएनए से जुड़ते हैं। डीएसआरएम आरएनए डुप्लेक्स के साथ α-हेलीकॉप्टर और β1-β2 लूप दोनों के माध्यम से बातचीत करने के लिए पाए गए हैं। इसके अतिरिक्त, सभी तीन डीएसआरबीएम संरचनाएं प्रमुख संरचना और छोटे संरचना के चीनी-फॉस्फेट रीढ़ के साथ संपर्क बनाती हैं, जो इस प्रकार अल्फा हेलिक्स 2 के ए N- टर्मिनस क्षेत्र के साथ β1-β2 लूप द्वारा मध्यस्थ होता है। यह बातचीत है आरएनए डबल हेलिक्स के आकार के लिए अद्वितीय अनुकूलन क्योंकि इसमें 2'-हाइड्रॉक्सिल और फॉस्फेट ऑक्सीजन सम्मिलित है। इस प्रकार डीएसआरबीएम के बीच सामान्य संरचनात्मक विशेषताओं के अतिरिक्त, वे अलग-अलग रासायनिक ढांचे का प्रदर्शन करते हैं, जो आरएनए संरचनाओं के लिए विविधता के लिए विशिष्टता की अनुमति देता है जिसमें स्टेम-लूप, आंतरिक लूप, उभार या बेमेल सम्मिलित हैं।[12]
जिंक फिंगर
यूकेरियोटिक जीनोम के भीतर सीसीएचएच-टाइप जिंक फिंगर डोमेन सबसे साधारण डीएनए-बाध्यकारी डोमेन हैं। डीएनए की उच्च अनुक्रम-विशिष्ट पहचान प्राप्त करने के लिए, मॉड्यूलर फैशन में कई जिंक फिंगर का उपयोग किया जाता है। जिंक फिंगर ββα प्रोटीन फोल्ड प्रदर्शित करती हैं जिसमें β-हेयरपिन और α-हेलिक्स के माध्यम से Zn2+
आयन जुड़ जाते हैं। इसके अतिरिक्त, प्रमुख संरचना में डीएनए आधारों के साथ α-हेलिक्स की प्रोटीन साइड-चेन के बीच की बातचीत डीएनए-अनुक्रम-विशिष्ट पहचान के लिए अनुमति देती है। डीएनए की व्यापक मान्यता के अतिरिक्त, हाल ही में ऐसी खोजें हुई हैं कि ज़िंक फिंगर में आरएनए को पहचानने की क्षमता भी होती है। इस प्रकार सीसीएचएच जिंक फिंगर्स के अतिरिक्त, सीसीसीएच जिंक फिंगर्स को हाल ही में इंटरमॉलिक्युलर हाइड्रोजन बंध और आरएनए बेस के वाटसन-क्रिक किनारों के बीच बातचीत के माध्यम से एकल-फंसे हुए आरएनए की अनुक्रम-विशिष्ट पहचान को नियोजित करने के लिए खोजा गया था। इस प्रकार सीसीएचएच-टाइप जिंक फिंगर्स आरएनए बाध्यकारी के दो तरीके अपनाते हैं। सबसे पहले, जिंक फिंगर दोहरी कुंडली की रीढ़ के साथ गैर-विशिष्ट बातचीत करती हैं, जबकि दूसरी मोड जिंक फिंगर को विशेष रूप से अलग-अलग आधारों को पहचानने की अनुमति देती है जो बाहर निकलते हैं। सीसीएचएच-प्रकार से भिन्न, सीसीसीएच-प्रकार की जिंक फिंगर आरएनए बाध्यकारी की अन्य विधा प्रदर्शित करती है, जिसमें एकल-स्ट्रैंडेड आरएनए को अनुक्रम-विशिष्ट विधि से पहचाना जाता है। कुल मिलाकर, जिंक फिंगर्स डीएसडीएनए अनुक्रम से बाध्यकारी के माध्यम से और एसएसआरएनए अनुक्रम से बाध्यकारी के माध्यम से आरएनए को सीधे डीएनए की पहचान कर सकते हैं।[12]
भ्रूण के विकास में भूमिका
आरएनए-बाध्यकारी प्रोटीन के आरएनए के ट्रांसक्रिप्शनल और पोस्ट-ट्रांसक्रिप्शनल विनियमन की विकास के समय जीन अभिव्यक्ति के क्रम को विनियमित करने में भूमिका होती है।[13] नेमाटोड कैनोर्हाडाइटिस एलिगेंस पर व्यापक शोध या सी एलिगेंस ने आरएनए-बाध्यकारी प्रोटीन की जर्मलाइन और प्रारंभिक भ्रूण विकास के समय आवश्यक कारकों के रूप में पहचान की है। इस प्रकार इनके विशिष्ट कार्य में दैहिक (जीव विज्ञान) ऊतकों (न्यूरॉन्स, हाइपोडर्मिस, मांसपेशियों और उत्सर्जन कोशिकाओं) के विकास के साथ-साथ विकासात्मक घटनाओं के लिए समय संकेत प्रदान करना सम्मिलित है। फिर भी, उनके आरएनए लक्ष्यों की पहचान करने में कठिनाई के कारण विकास में आरबीपी के कार्य के पीछे के तंत्र की खोज करना असाधारण रूप से चुनौतीपूर्ण है। ऐसा इसलिए है क्योंकि अधिकांशतः आरबीपी में साधारणतयः कई आरएनए लक्ष्य होते हैं।[14] चूंकि, यह निर्विवाद है कि आरबीपीठोस तरीके से विकासात्मक मार्गों को विनियमित करने में महत्वपूर्ण नियंत्रण रखते हैं।
जर्मलाइन विकास
ड्रोसोफिला मेलानोगास्टर में, एलाव, एसएक्सएल और टीआरए-2 आरएनए-बाध्यकारी प्रोटीन एन्कोडिंग जीन हैं, जो इस प्रकार प्रारंभिक लिंग-निर्धारण प्रणाली और दैहिक यौन अवस्था के रखरखाव में महत्वपूर्ण हैं।[15] ये जीन ड्रोसोफिला में सेक्स-विशिष्ट विभाजन को विनियमित करके पोस्ट-ट्रांसक्रिप्शनल स्तर पर प्रभाव डालते हैं। इस प्रकार एसएक्सएल महिलाओं में कार्यात्मक टीआरए एमआरएनए उत्पन्न करने के लिए स्त्रीलिंग जीन टीआरए का धनात्मक नियमन करता है। C. एलिगेंस में, फाॅग-1, माॅग-1/-4/-5 और आरएनपी-4 सहित आरएनए-बाध्यकारी प्रोटीन जर्मलाइन और दैहिक लिंग निर्धारण नियंत्रित करते हैं। इसके अतिरिक्त, कई आरबीपी जैसे जीएलडी-1, जीएलडी-3, डीएजेड-1, पीजीएल-1 और ओएमए-1/-2 अर्धसूत्रीविभाजन प्रोफेज़ प्रगति, युग्मकजनन, और अंडकोशिका परिपक्वता के समय अपने नियामक कार्यों को पूरा करते हैं।[14]
दैहिक विकास
जर्मलाइन विकास में आरबीपी के कार्यों के अतिरिक्त, पोस्ट-ट्रांसक्रिप्शनल नियंत्रण भी दैहिक विकास में महत्वपूर्ण भूमिका निभाता है। रोगाणु और प्रारंभिक भ्रूण विकास में सम्मिलित आरबीपी से भिन्न, दैहिक विकास में कार्य करने वाले आरबीपी एमआरएनए लक्ष्यों के ऊतक-विशिष्ट वैकल्पिक विभाजन को विनियमित करते हैं। उदाहरण के लिए, आरआरएम डोमेन वाले एमईसी-8 और यूएनसी-75 क्रमशः हाइपोडर्मिस और तंत्रिका तंत्र के क्षेत्रों में स्थानीय होते हैं।[14] इसके अतिरिक्त, अन्य आरआरएम-युक्त आरबीपी, ईएक्ससी-7, दैहिक विकास के समय भ्रूण के उत्सर्जक नलिका कोशिकाओं और पूरे तंत्रिका तंत्र में स्थानीयकरण के लिए प्रकट होता है।
तंत्रिका संबंधी विकास
जेडबीपी1 को हिप्पोकैम्पस न्यूरॉन्स में डेंड्रिटोजेनेसिस ( डेन्ड्राइट गठन) को विनियमित करने के लिए दिखाया गया था।[16] इस प्रकार डेन्ड्राइट निर्माण में सम्मिलित अन्य आरएनए-बाध्यकारी प्रोटीन पम2 और नैनो हैं,[17] जैसे एफएमआर1, सीपीईबी और स्टॉफ़ेन (प्रोटीन) इसका प्रमुख उदाहरण हैं।[18]
कैंसर में भूमिका
ट्यूमर के विकास में महत्वपूर्ण भूमिका निभाने के लिए आरबीपी उभर रहे हैं।[19] सैकड़ों आरबीपी मानव कैंसर में स्पष्ट रूप से विकृत हैं, और इस प्रकार सामान्य ऊतकों से संबंधित ट्यूमर में प्रमुख गिरावट दिखाते हैं।[19]कई आरबीपी विभिन्न प्रकार के कैंसर में भिन्न रूप से अभिव्यक्त होते हैं, उदाहरण के लिए केएचडीआरबीएस1(सैम68),[20][21][22] ईएलएवीएल1 (हूर),[23][24] एफएक्सआर1[25] और यूएचएमके1.[26] कुछ आरबीपी के लिए, अभिव्यक्ति में परिवर्तन कॉपी संख्या भिन्नता (सीएनवी) से संबंधित है, उदाहरण के लिए कोलोरेक्टल कैंसर कोशिकाओं में बीवाईएसएल का सीएनवी लाभ[19]और ईएसआरपी1, ब्रेस्ट कैंसर में सीईएलएफ3, लिवर कैंसर में आरजीएम24, फेफड़ों के कैंसर में आईजीएफ2बीपी2, आईजीएफ2बीपी3 या फेफड़ों के कैंसर में केएचडीआरबीएस2 की सीएनवी हानियाँ हैं।[27] इस प्रकार इन आरबीपीपर प्रोटीन को प्रभावित करने वाले उत्परिवर्तन के कारण कुछ अभिव्यक्ति परिवर्तन होते हैं, उदाहरण के लिए एनएसयूएन6, जेडसी3एच13, ईएलएसी1, आरजीएमएस3, और जेडजीपीएटी, एसएफ3बी1, एसआरएसएफ2, आरजीएम10, यू2एएफ1, एसएफ3बी1, पीपीआरसी1, आरजीएमएक्सएल1, एचएनआरएनपीसीएल1 आदि।[19][28][29][30][31] कई अध्ययनों ने आरबीपी की अभिव्यक्ति में इस परिवर्तन को कैंसर में असामान्य वैकल्पिक विभाजन से संबंधित बताया है।[32][33][34]
वर्तमान शोध

चूंकि आरएनए-बाध्यकारी प्रोटीन कई सेलुलर कार्यों पर महत्वपूर्ण नियंत्रण रखते हैं, वे कई शोधकर्ताओं के लिए जांच का लोकप्रिय क्षेत्र रहे हैं। जैविक क्षेत्र में इसके महत्व के कारण, हाल ही में आरएनए-बाध्यकारी प्रोटीन की क्षमता के बारे में कई खोजों का अनावरण किया गया है।[12] इस प्रकार आरएनए-बाध्यकारी प्रोटीनों की प्रायोगिक पहचान में वर्तमान समय में विकास ने आरएनए-बाध्यकारी प्रोटीनों की संख्या में महत्वपूर्ण रूप से वृद्धि की है[35][36][37]
आरएनए-बाध्यकारी प्रोटीन सैम68 आरएनए उपापचय के स्थानिक और लौकिक कंपार्टमेंटलाइज़ेशन को नियंत्रित करता है ताकि डेंड्राइट्स में उचित निष्कर्ष फलन प्राप्त किया जा सके। सैम68 की हानि के परिणामस्वरूप असामान्य पोस्टट्रांसक्रिप्शनल रेगुलेशन होता है और अंतत: नाजुक एक्स-जुड़े कंपकंपी / गतिभंग सिंड्रोम जैसे न्यूरोलॉजिकल विकारों की ओर जाता है। इस प्रकार सैम68 को एमआरएनए एन्कोडिंग β-एक्टिन के साथ बातचीत करने के लिए पाया गया, जो अपने साइटोस्केलेटल घटकों के साथ डेंड्राइटिक स्पाइन के सिनैप्टिक गठन को नियंत्रित करता है। इसलिए इस प्रकार सैम68 पोस्टसिनेप्टिक β-एक्टिन एमआरएनए चयापचय के नियंत्रण के माध्यम से सिनैप्स संख्या को विनियमित करने में महत्वपूर्ण भूमिका निभाता है।[38]
न्यूरॉन-विशिष्ट सीईएलएफ परिवार आरएनए-बाध्यकारी प्रोटीन यूएनसी-75 विशेष रूप से सी एलिगेंस के न्यूरोनल कोशिकाओं में एक्सॉन 7ए चयन के लिए अपने तीन आरएनए मान्यता रूपांकनों के माध्यम से यूयूजीयूयूजीयूजीयूयूजीयू एमआरएनए खिंचाव को बांधता है। जैसा कि एक्सॉन 7ए को गैर-न्यूरोनल कोशिकाओं में कमजोर स्थलों के कारण छोड़ दिया गया है, यूएनसी-75 को विशेष रूप से एक्सॉन 7a और एक्सॉन 8 के बीच केवल न्यूरोनल कोशिकाओं में सक्रिय करने के लिए पाया गया था।[39]
कोल्ड इंड्यूसिबल आरएनए बाध्यकारी प्रोटीन सीआईआरबीपी शॉर्ट वेवलेंथ पराबैंगनी प्रकाश, हाइपोक्सिया (चिकित्सा) और अल्प तपावस्था सहित विभिन्न प्रकार के सेलुलर तनावों का सामना करने पर सेलुलर प्रतिक्रिया को नियंत्रित करने में भूमिका निभाता है। इस शोध से सूजन के साथ रोग राज्यों के जुड़ाव के संभावित प्रभाव सामने आए हैं।[40] इस प्रकार आरएनए-बाध्यकारी प्रोटीन एसएलआर1 के सेरीन-आर्जिनिन परिवार को कैनडीडा अल्बिकन्स में ध्रुवीकृत वृद्धि पर अत्यधिक नियंत्रण पाया गया था। इस प्रकार चूहों में एसएलआर1 म्यूटेशन के परिणामस्वरूप फिलामेंटेशन कम हो जाता है और अन्तःस्तरीय कोशिका और एंडोथेलियल उपकला कोशिका की हानि कम हो जाती है जो एसएलआर1 जंगली प्रकार के उपभेदों की तुलना में विस्तारित जीवित रहने की दर की ओर जाता है। इसलिए, इस शोध से पता चलता है कि एसआर-जैसे प्रोटीन एसएलआर1 सी. अल्बिकैंस में हाइपल गठन और विषाणु को भड़काने में भूमिका निभाता है।[41]
यह भी देखें
- डीएनए बाध्यकारी प्रोटीन
- आरएनए-बाध्यकारी प्रोटीन डेटाबेस
- राइबोन्यूक्लियोप्रोटीन
बाहरी संबंध
- starBase platform: a platform for decoding binding sites of आरएनए binding proteins (RBPs) from large-scale CLIP-Seq (HITS-CLIP, PAR-CLIP, iCLIP, CLASH) datasets.
- RBPDB database: a database of आरएनए binding proteins.
- oआरएनएment: a database of putative RBP binding site instances in both coding and non-coding आरएनए in various species.
- ATtRACt database: a database of आरएनए binding proteins and associated motifs.
- SplicedAid-F: a database of hand -cureted human आरएनए binding proteins database.
- RsiteDB: आरएनए binding site database
- SPOT-Seq-आरएनए: Template-based prediction of आरएनए binding proteins and their complex structures.
- SPOT-Struct-आरएनए: आरएनए binding proteins prediction from 3D structures.
- ENCODE Project: A collection of genomic datasets (i.e. आरएनए Bind-n-seq, eCLIP, RBP targeted shआरएनए आरएनए-seq) for RBPs
- RBP Image Database: Images showing the cellular localization of आरबीपीin cells
- RBPSpot Software: A Deep-Learning based highly accurate software to detect RBP-आरएनए interaction. It also provides a module to build new RBP-आरएनए interaction models.
संदर्भ
- ↑ RNA-Binding+Proteins at the US National Library of Medicine Medical Subject Headings (MeSH)
- ↑ 2.0 2.1 Lunde BM, Moore C, Varani G (June 2007). "RNA-binding proteins: modular design for efficient function". Nature Reviews. Molecular Cell Biology. 8 (6): 479–90. doi:10.1038/nrm2178. PMC 5507177. PMID 17473849.
- ↑ Liu, S; Li, B; Liang, Q; Liu, A; Qu, L; Yang, J (November 2020). "आरएनए-प्रोटीन इंटरैक्शन का वर्गीकरण और कार्य।". Wiley Interdisciplinary Reviews. RNA. 11 (6): e1601. doi:10.1002/wrna.1601. PMID 32488992. S2CID 219284021.
- ↑ Hogan DJ, Riordan DP, Gerber AP, Herschlag D, Brown PO (October 2008). "विविध आरएनए-बाध्यकारी प्रोटीन आरएनए के कार्यात्मक रूप से संबंधित सेटों के साथ परस्पर क्रिया करते हैं, एक व्यापक नियामक प्रणाली का सुझाव देते हैं". PLOS Biology. 6 (10): e255. doi:10.1371/journal.pbio.0060255. PMC 2573929. PMID 18959479.
- ↑ 5.0 5.1 5.2 5.3 5.4 5.5 5.6 5.7 Glisovic T, Bachorik JL, Yong J, Dreyfuss G (June 2008). "आरएनए-बाध्यकारी प्रोटीन और पोस्ट-ट्रांसक्रिप्शनल जीन विनियमन". FEBS Letters. 582 (14): 1977–86. doi:10.1016/j.febslet.2008.03.004. PMC 2858862. PMID 18342629.
- ↑ Liu, S; Li, B; Liang, Q; Liu, A; Qu, L; Yang, J (November 2020). "आरएनए-प्रोटीन इंटरैक्शन का वर्गीकरण और कार्य।". Wiley Interdisciplinary Reviews. RNA. 11 (6): e1601. doi:10.1002/wrna.1601. PMID 32488992. S2CID 219284021.
- ↑ 7.0 7.1 Matera AG, Terns RM, Terns MP (March 2007). "Non-coding RNAs: lessons from the small nuclear and small nucleolar RNAs". Nature Reviews. Molecular Cell Biology. 8 (3): 209–20. doi:10.1038/nrm2124. PMID 17318225. S2CID 30268055.
- ↑ Fu XD, Ares M (October 2014). "आरएनए-बाइंडिंग प्रोटीन द्वारा वैकल्पिक स्प्लिसिंग का संदर्भ-निर्भर नियंत्रण". Nature Reviews. Genetics. 15 (10): 689–701. doi:10.1038/nrg3778. PMC 4440546. PMID 25112293.
- ↑ Dictenberg JB, Swanger SA, Antar LN, Singer RH, Bassell GJ (June 2008). "गतिविधि-निर्भर डेंड्राइटिक एमआरएनए ट्रांसपोर्ट में एफएमआरपी के लिए एक सीधी भूमिका फ़िलाओपोडियल-स्पाइन मॉर्फोजेनेसिस को नाजुक एक्स सिंड्रोम से जोड़ती है". Developmental Cell. 14 (6): 926–39. doi:10.1016/j.devcel.2008.04.003. PMC 2453222. PMID 18539120.
- ↑ Hale, Caryn R.; Sawicka, Kirsty; Mora, Kevin; Fak, John J.; Kang, Jin Joo; Cutrim, Paula; Cialowicz, Katarzyna; Carroll, Thomas S.; Darnell, Robert B. (2021-12-23). "FMRP सेल बॉडी और CA1 पिरामिड न्यूरॉन्स के डेंड्राइट्स में अलग-अलग कार्यों को एन्कोडिंग करने वाले mRNAs को नियंत्रित करता है". eLife. 10: e71892. doi:10.7554/eLife.71892. ISSN 2050-084X. PMC 8820740. PMID 34939924.
- ↑ Liu, S; Li, B; Liang, Q; Liu, A; Qu, L; Yang, J (November 2020). "आरएनए-प्रोटीन इंटरैक्शन का वर्गीकरण और कार्य।". Wiley Interdisciplinary Reviews. RNA. 11 (6): e1601. doi:10.1002/wrna.1601. PMID 32488992. S2CID 219284021.
- ↑ 12.0 12.1 12.2 12.3 12.4 Stefl R, Skrisovska L, Allain FH (January 2005). "राइबोन्यूक्लियोप्रोटीन कण में प्रोटीन द्वारा आरएनए अनुक्रम- और आकार-निर्भर पहचान". EMBO Reports. 6 (1): 33–8. doi:10.1038/sj.embor.7400325. PMC 1299235. PMID 15643449.
- ↑ Appasani, Krishnarao (2008). MicroRNAs: From Basic Science to Disease Biology. Cambridge University Press. p. 485. ISBN 978-0-521-86598-2. Retrieved 12 May 2013.
- ↑ 14.0 14.1 14.2 Lee M, Schedl T (18 April 2006). "RNA-binding proteins". वर्मबुक. pp. 1–13. doi:10.1895/wormbook.1.79.1. PMC 4781538. PMID 18050487.
{{cite book}}:|journal=ignored (help) - ↑ Bandziulis RJ, Swanson MS, Dreyfuss G (April 1989). "विकास नियामकों के रूप में आरएनए-बाध्यकारी प्रोटीन". Genes & Development. 3 (4): 431–7. doi:10.1101/gad.3.4.431. PMID 2470643.
- ↑ Perycz M, Urbanska AS, Krawczyk PS, Parobczak K, Jaworski J (April 2011). "जिपकोड बाइंडिंग प्रोटीन 1 हिप्पोकैम्पस न्यूरॉन्स में डेंड्राइटिक आर्बर्स के विकास को नियंत्रित करता है". The Journal of Neuroscience. 31 (14): 5271–85. doi:10.1523/JNEUROSCI.2387-10.2011. PMC 6622686. PMID 21471362.
- ↑ Ye B, Petritsch C, Clark IE, Gavis ER, Jan LY, Jan YN (February 2004). "ड्रोसोफिला परिधीय न्यूरॉन्स में डेंड्राइट मॉर्फोजेनेसिस के लिए नैनो और पुमिलियो आवश्यक हैं". Current Biology. 14 (4): 314–21. doi:10.1016/j.cub.2004.01.052. PMID 14972682.
- ↑ Vessey JP, Macchi P, Stein JM, Mikl M, Hawker KN, Vogelsang P, et al. (October 2008). "murine Staufen1 के लिए फ़ंक्शन एलील के नुकसान से डेंड्राइटिक Staufen1-RNP डिलीवरी और डेंड्राइटिक स्पाइन मॉर्फोजेनेसिस की हानि होती है". Proceedings of the National Academy of Sciences of the United States of America. 105 (42): 16374–9. Bibcode:2008PNAS..10516374V. doi:10.1073/pnas.0804583105. JSTOR 25465098. PMC 2567905. PMID 18922781.
- ↑ 19.0 19.1 19.2 19.3 Wang ZL, Li B, Luo YX, Lin Q, Liu SR, Zhang XQ, et al. (January 2018). "मानव कैंसर में आरएनए-बाध्यकारी प्रोटीन का व्यापक जीनोमिक लक्षण वर्णन". Cell Reports. 22 (1): 286–298. doi:10.1016/j.celrep.2017.12.035. PMID 29298429.
- ↑ Bielli P, Busà R, Paronetto MP, Sette C (August 2011). "The RNA-binding protein Sam68 is a multifunctional player in human cancer". Endocrine-Related Cancer. 18 (4): R91–R102. doi:10.1530/ERC-11-0041. hdl:2108/88068. PMID 21565971.
- ↑ Liao WT, Liu JL, Wang ZG, Cui YM, Shi L, Li TT, et al. (August 2013). "High expression level and nuclear localization of Sam68 are associated with progression and poor prognosis in colorectal cancer". BMC Gastroenterology. 13: 126. doi:10.1186/1471-230X-13-126. PMC 3751151. PMID 23937454.
- ↑ Frisone P, Pradella D, Di Matteo A, Belloni E, Ghigna C, Paronetto MP (26 July 2015). "SAM68: Signal Transduction and RNA Metabolism in Human Cancer". BioMed Research International. 2015: 528954. doi:10.1155/2015/528954. PMC 4529925. PMID 26273626.
- ↑ Abdelmohsen K, Gorospe M (1 September 2010). "एचयूआर द्वारा कैंसर लक्षणों का पोस्टट्रांसक्रिप्शनल विनियमन". Wiley Interdisciplinary Reviews: RNA. 1 (2): 214–29. doi:10.1002/wrna.4. PMC 3808850. PMID 21935886.
- ↑ Wang J, Guo Y, Chu H, Guan Y, Bi J, Wang B (May 2013). "कैंसर की प्रगति, उपचार प्रतिक्रियाओं और पूर्वानुमान में आरएनए-बाध्यकारी प्रोटीन एचयूआर के कई कार्य". International Journal of Molecular Sciences. 14 (5): 10015–41. doi:10.3390/ijms140510015. PMC 3676826. PMID 23665903.
- ↑ Qian J, Hassanein M, Hoeksema MD, Harris BK, Zou Y, Chen H, et al. (March 2015). "The RNA binding protein FXR1 is a new driver in the 3q26-29 amplicon and predicts poor prognosis in human cancers". Proceedings of the National Academy of Sciences of the United States of America. 112 (11): 3469–74. Bibcode:2015PNAS..112.3469Q. doi:10.1073/pnas.1421975112. PMC 4371932. PMID 25733852.
- ↑ Feng, Xing; Ma, Dong; Zhao, Jiabao; Song, Yongxi; Zhu, Yuekun; Zhou, Qingxin; Ma, Fei; Liu, Xing; Zhong, Mengya; Liu, Yu; Xiong, Yubo (2020-03-02). "UHMK1 रीप्रोग्रामिंग न्यूक्लियोटाइड चयापचय के माध्यम से गैस्ट्रिक कैंसर की प्रगति को बढ़ावा देता है". The EMBO Journal. 39 (5): e102541. doi:10.15252/embj.2019102541. ISSN 1460-2075. PMC 7049804. PMID 31975428.
- ↑ {{cite journal | vauthors = Sebestyén E, Singh B, Miñana B, Pagès A, Mateo F, Pujana MA, Valcárcel J, Eyras E | display-authors = 6 | title = कई ट्यूमर में जीनोम और ट्रांसक्रिपटोम परिवर्तन के बड़े पैमाने पर विश्लेषण ने उपन्यास कैंसर-संबंधित स्प्लिसिंग नेटवर्क का खुलासा किया| journal = Genome Research | volume = 26 | issue = 6 | pages = 732–44 | date = June 2016 | pmid = 27197215 | pmc = 4889968 | doi = 10.1101/gr.199935.115 }<
- ↑ सेबेस्टियन 732-744
- ↑ Yoshida K, Sanada M, Shiraishi Y, Nowak D, Nagata Y, Yamamoto R, et al. (September 2011). "माइलोडिसप्लासिया में स्प्लिसिंग मशीनरी का बार-बार मार्ग परिवर्तन". Nature. 478 (7367): 64–9. Bibcode:2011Natur.478...64Y. doi:10.1038/nature10496. PMID 21909114. S2CID 4429386.
- ↑ Imielinski M, Berger AH, Hammerman PS, Hernandez B, Pugh TJ, Hodis E, et al. (September 2012). "बड़े पैमाने पर समानांतर अनुक्रमण के साथ फेफड़े के एडेनोकार्सिनोमा के हॉलमार्क का मानचित्रण करना". Cell (in English). 150 (6): 1107–20. doi:10.1016/j.cell.2012.08.029. PMC 3557932. PMID 22980975.
- ↑ Ellis MJ, Ding L, Shen D, Luo J, Suman VJ, Wallis JW, et al. (June 2012). "संपूर्ण-जीनोम विश्लेषण एरोमाटेज निषेध के लिए स्तन कैंसर की प्रतिक्रिया की सूचना देता है". Nature. 486 (7403): 353–60. Bibcode:2012Natur.486..353E. doi:10.1038/nature11143. PMC 3383766. PMID 22722193.
- ↑ सेबस्टीन 732–744
- ↑ David CJ, Manley JL (November 2010). "Alternative pre-mRNA splicing regulation in cancer: pathways and programs unhinged". Genes & Development. 24 (21): 2343–64. doi:10.1101/gad.1973010. PMC 2964746. PMID 21041405.
- ↑ Fredericks AM, Cygan KJ, Brown BA, Fairbrother WG (May 2015). "RNA-Binding Proteins: Splicing Factors and Disease". Biomolecules. 5 (2): 893–909. doi:10.3390/biom5020893. PMC 4496701. PMID 25985083.
- ↑ Conrad T, Albrecht AS, de Melo Costa VR, Sauer S, Meierhofer D, Ørom UA (April 2016). "मानव कोशिका नाभिक का सीरियल इंटरएक्टिव कैप्चर". Nature Communications. 7: 11212. Bibcode:2016NatCo...711212C. doi:10.1038/ncomms11212. PMC 4822031. PMID 27040163.
- ↑ Castello A, Fischer B, Eichelbaum K, Horos R, Beckmann BM, Strein C, et al. (June 2012). "स्तनधारी एमआरएनए-बाध्यकारी प्रोटीन के एटलस से आरएनए जीव विज्ञान में अंतर्दृष्टि". Cell. 149 (6): 1393–406. doi:10.1016/j.cell.2012.04.031. PMID 22658674.
- ↑ Baltz AG, Munschauer M, Schwanhäusser B, Vasile A, Murakawa Y, Schueler M, et al. (June 2012). "प्रोटीन-कोडिंग ट्रांस्क्रिप्ट पर एमआरएनए-बाउंड प्रोटिओम और इसकी वैश्विक अधिभोग प्रोफ़ाइल". Molecular Cell. 46 (5): 674–90. doi:10.1016/j.molcel.2012.05.021. PMID 22681889.
- ↑ Klein ME, Younts TJ, Castillo PE, Jordan BA (February 2013). "RNA-binding protein Sam68 controls synapse number and local β-actin mRNA metabolism in dendrites". Proceedings of the National Academy of Sciences of the United States of America. 110 (8): 3125–30. Bibcode:2013PNAS..110.3125K. doi:10.1073/pnas.1209811110. PMC 3581878. PMID 23382180.
- ↑ Kuroyanagi H, Watanabe Y, Hagiwara M (2013). Blumenthal T (ed.). "CELF family RNA-binding protein UNC-75 regulates two sets of mutually exclusive exons of the unc-32 gene in neuron-specific manners in Caenorhabditis elegans". PLOS Genetics. 9 (2): e1003337. doi:10.1371/journal.pgen.1003337. PMC 3585155. PMID 23468662.
- ↑ Brochu C, Cabrita MA, Melanson BD, Hamill JD, Lau R, Pratt MA, McKay BC (2013). Gallouzi IE (ed.). "NF-κB-dependent role for cold-inducible RNA binding protein in regulating interleukin 1β". PLOS ONE. 8 (2): e57426. Bibcode:2013PLoSO...857426B. doi:10.1371/journal.pone.0057426. PMC 3578848. PMID 23437386.
- ↑ Ariyachet C, Solis NV, Liu Y, Prasadarao NV, Filler SG, McBride AE (April 2013). "एसआर-जैसे आरएनए-बाइंडिंग प्रोटीन एसएलआर1 कैंडिडा एल्बीकैंस फिलामेंटेशन और विषाणु को प्रभावित करता है". Infection and Immunity. 81 (4): 1267–76. doi:10.1128/IAI.00864-12. PMC 3639594. PMID 23381995.