जीन द्विगुणन: Difference between revisions
m (16 revisions imported from alpha:जीन_डुप्लीकेशन) |
m (Abhishekkshukla moved page जीन डुप्लीकेशन to जीन द्विगुणन without leaving a redirect) |
||
| (One intermediate revision by one other user not shown) | |||
| Line 174: | Line 174: | ||
{{Self-replicating organic structures}} | {{Self-replicating organic structures}} | ||
{{DEFAULTSORT:Gene Duplication}} | {{DEFAULTSORT:Gene Duplication}} | ||
[[Category:Articles with hatnote templates targeting a nonexistent page|Gene Duplication]] | |||
[[Category:CS1 English-language sources (en)]] | |||
[[Category: | [[Category:Collapse templates|Gene Duplication]] | ||
[[Category:Created On 10/07/2023]] | [[Category:Created On 10/07/2023|Gene Duplication]] | ||
[[Category:Vigyan Ready]] | [[Category:Lua-based templates|Gene Duplication]] | ||
[[Category:Machine Translated Page|Gene Duplication]] | |||
[[Category:Multi-column templates|Gene Duplication]] | |||
[[Category:Navigational boxes| ]] | |||
[[Category:Navigational boxes without horizontal lists|Gene Duplication]] | |||
[[Category:Pages using div col with small parameter|Gene Duplication]] | |||
[[Category:Pages with script errors|Gene Duplication]] | |||
[[Category:Sidebars with styles needing conversion|Gene Duplication]] | |||
[[Category:Template documentation pages|Documentation/doc]] | |||
[[Category:Templates Vigyan Ready|Gene Duplication]] | |||
[[Category:Templates generating microformats|Gene Duplication]] | |||
[[Category:Templates that add a tracking category|Gene Duplication]] | |||
[[Category:Templates that are not mobile friendly|Gene Duplication]] | |||
[[Category:Templates that generate short descriptions|Gene Duplication]] | |||
[[Category:Templates using TemplateData|Gene Duplication]] | |||
[[Category:Templates using under-protected Lua modules|Gene Duplication]] | |||
[[Category:Wikipedia fully protected templates|Div col]] | |||
[[Category:Wikipedia metatemplates|Gene Duplication]] | |||
[[Category:आणविक विकास|Gene Duplication]] | |||
[[Category:आनुवंशिक जानकारी का संशोधन|Gene Duplication]] | |||
[[Category:आनुवंशिकी अवधारणाएँ|Gene Duplication]] | |||
[[Category:उत्परिवर्तन|Gene Duplication]] | |||
[[Category:विकासवादी जीवविज्ञान अवधारणाएँ|Gene Duplication]] | |||
Latest revision as of 12:27, 8 September 2023
जीन डुप्लीकेशन (या क्रोमोसोमल डुप्लीकेशन या जीन प्रवर्धन) ऐसी प्रमुख प्रणाली है जिसके माध्यम से आणविक विकास के समय नई जीन सामग्री उत्पन्न होती है। इसे डीएनए के उस क्षेत्र के किसी भी डुप्लीकेशन के रूप में परिभाषित किया जा सकता है जिसमें जीन उपस्थित होता है। जीन डुप्लीकेशन डीएनए प्रतिकृति और डीएनए त्रुटिनिवारण मशीनरी में कई प्रकार की त्रुटियों के साथ-साथ स्वार्थपरायण जीन तत्वों द्वारा आकस्मिक अधिकार के परिणामस्वरूप उत्पन्न हो सकता है। जीन डुप्लीकेशन के सामान्य स्रोतों में एक्टोपिक पुनर्संयोजन, रेट्रोट्रांसपोसन परिणाम, एन्यूप्लोइडी, पॉलीप्लोइडी और प्रतिकृति स्लिपेज सम्मिलित हैं।[1]
डुप्लीकेशन की प्रणाली
एक्टोपिक पुनर्संयोजन
डुप्लीकेशन ऐसी घटना से उत्पन्न होता है जिसे असमान क्रॉसिंग-ओवर कहा जाता है जो कि त्रुटिपूर्ण संरेखित समजात गुणसूत्रों के मध्य अर्धसूत्रीविभाजन के समय होता है। ऐसा होने की संभावना दो गुणसूत्रों के मध्य डुप्लीकेशन वाले तत्वों के विभाजन की डिग्री पर निर्भर करती है। इस पुनर्संयोजन के उत्पाद विनिमय स्थल पर डुप्लीकेशन और पारस्परिक विलोपन हैं। एक्टोपिक पुनर्संयोजन सामान्यतः डुप्लिकेट ब्रेकप्वाइंट पर अनुक्रम समानता द्वारा मध्यस्थ होता है, जो प्रत्यक्ष डुप्लीकेशन बनाता है। डुप्लीकेशन जीन तत्व जैसे ट्रांसपोज़ेबल तत्व डुप्लीकेशन डीएनए का स्रोत प्रदान करते हैं जो पुनर्संयोजन की सुविधा प्रदान कर सकते हैं, और वे प्रायः पौधों और स्तनधारियों में डुप्लीकेशन ब्रेकप्वाइंट पर पाए जाते हैं।[2]
प्रतिकृति स्लिपेज
प्रतिकृति स्लिपेज डीएनए प्रतिकृति में ऐसी त्रुटि है जो लघु जीन अनुक्रमों के डुप्लीकेशन का उत्पादन कर सकती है। प्रतिकृति के समय डीएनए पोलीमरेज़ डीएनए की प्रतिलिपि बनाना प्रारंभ कर देता है। प्रतिकृति प्रक्रिया के समय कुछ बिंदु पर, पोलीमरेज़ डीएनए से भिन्न हो जाता है और प्रतिकृति रुक जाती है। जब पोलीमरेज़ डीएनए स्ट्रैंड से दोबारा जुड़ता है, तो यह प्रतिकृति स्ट्रैंड को त्रुटिपूर्ण स्थिति में संरेखित करता है और संयोग से एक ही सेक्शन को एक से अधिक बार कॉपी करता है। प्रतिकृति स्लिपेज को प्रायः डुप्लीकेशन किए गए अनुक्रमों द्वारा भी सुविधाजनक बनाया जाता है, किन्तु इसके लिए समानता के केवल कुछ आधारों की आवश्यकता होती है।
रेट्रोट्रांसपोज़िशन
रेट्रोट्रांसपोज़न, मुख्य रूप से लाइन1, कभी-कभी सेलुलर एमआरएनए पर कार्य कर सकता है। प्रतिलेखों को डीएनए में विपरीत प्रतिलेखित किया जाता है और जीनोम में यादृच्छिक स्थान पर उत्पन्न किया जाता है, जिससे रेट्रोजेन का निर्माण होता है। परिणामी अनुक्रम में सामान्यतः इंट्रॉन की अल्पता होती है और प्रायः पॉली, अनुक्रम होते हैं जो जीनोम में भी एकीकृत होते हैं। कई रेट्रोजीन अपने पैतृक जीन अनुक्रमों की तुलना में जीन विनियमन में परिवर्तन प्रदर्शित करते हैं, जिसके परिणामस्वरूप कभी-कभी नए कार्य होते हैं। क्रोमोसोमल विकास को आकार देने के लिए रेट्रोजीन विभिन्न गुणसूत्रों के मध्य घूर्णन कर सकते हैं।[3]
एन्यूप्लोइडी
एन्यूप्लोइडी तब होता है जब एकल गुणसूत्र पर नॉनडिसजंक्शन के परिणामस्वरूप गुणसूत्रों की असामान्य संख्या उत्पन्न होती है। एन्यूप्लोइडी प्रायः हानिकारक होती है और स्तनधारियों में नियमित रूप से सहज गर्भपात (गर्भपात) हो जाता है। कुछ एन्यूप्लोइड व्यक्ति व्यवहार्य होते हैं, उदाहरण के लिए मनुष्यों में ट्राइसॉमी 21, जो डाउन सिंड्रोम की ओर ले जाता है। एन्यूप्लोइडी प्रायः जीन की मात्रा को ऐसी विधियों से परिवर्तित कर देता है जो जीव के लिए हानिकारक होते हैं; इसलिए, इसके जनसंख्या में विस्तारित होने की संभावना नहीं है।
पॉलीप्लोइडी
पॉलीप्लोइडी, या संपूर्ण जीनोम डुप्लीकेशन अर्धसूत्रीविभाजन के समय नॉनडिसजंक्शन का उत्पाद होता है जिसके परिणामस्वरूप पूर्ण जीनोम की अतिरिक्त प्रतियां बनती हैं। पॉलीप्लोइडी पौधों में सामान्य है, किन्तु यह जानवरों में भी हुआ है, जिसमें कशेरुक भाग में पूर्ण जीनोम डुप्लीकेशन (2आर परिकल्पना) के दो युग होते हैं, जो मनुष्यों तक पहुंचते हैं।[4] यह हेमियास्कोमाइसीट यीस्ट ~100 माइआ में भी हुआ है।[5][6]
पूर्ण जीनोम डुप्लीकेशन के पश्चात, जीनोम अस्थिरता, व्यापक जीन हानि, न्यूक्लियोटाइड प्रतिस्थापन के उच्च स्तर और नियामक नेटवर्क रीवायरिंग की अपेक्षाकृत अल्प अवधि होती है।[7][8] इसके अतिरिक्त, जीन मात्रा प्रभाव महत्वपूर्ण भूमिका निभाते हैं।[9] इस प्रकार, अधिकांश डुप्लिकेट छोटी अवधि के अंदर लुप्त हो जाते हैं, चूँकि, डुप्लिकेट का बड़ा भाग शेष रह जाता है।[10] रोचक विषय यह है कि नियमन में सम्मिलित जीनों को प्राथमिकता से निरंतर रखा जाता है।[11][12] इसके अतिरिक्त, नियामक जीन, विशेष रूप से हॉक्स जीन, के प्रतिधारण ने अनुकूली नवाचार को उत्पन्न किया है।
डुप्लिकेट जीन के प्रतिलेखन के स्तर पर तीव्रता से विकास और कार्यात्मक विचलन सामान्यतः लघु प्रतिलेखन कारक बाइंडिंग रूपांकनों में बिंदु उत्परिवर्तन द्वारा देखा गया है।[13][14] इसके अतिरिक्त, प्रोटीन फॉस्फोराइलेशन मोटिफ्स का तीव्रता से विकास, जो सामान्यतः तीव्रता से विकसित होने वाले आंतरिक रूप से अव्यवस्थित क्षेत्रों में अंतर्निहित होता है, डुप्लिकेट जीन के अस्तित्व और तीव्रता से अनुकूलन/नियोफंक्शनलाइजेशन के लिए योगदान कारक है।[15] इस प्रकार, जीन विनियमन (कम से कम पोस्ट-ट्रांसलेशनल स्तर पर) और जीनोम विकास के मध्य लिंक उपस्थित प्रतीत होता है।[15]
पॉलीप्लोइडी भी प्रजातिकरण का प्रसिद्ध स्रोत है, क्योंकि संतान, जिनमें मूल प्रजातियों की तुलना में गुणसूत्रों की संख्या भिन्न होती है, प्रायः गैर-पॉलीप्लॉइड जीवों के साथ प्रजनन करने में असमर्थ होती हैं। संपूर्ण जीनोम डुप्लीकेशन को एन्यूप्लोइडी की तुलना में कम हानिकारक माना जाता है क्योंकि व्यक्तिगत जीन की सापेक्ष मात्रा समान होनी चाहिए।
विकासवादी घटना के रूप में
जीन डुप्लीकेशन की दर
जीनोम की तुलना से ज्ञात हुआ है कि परीक्षण की गई अधिकांश प्रजातियों में जीन डुप्लीकेशन सामान्य है। इसका संकेत मनुष्यों या फल मक्खियों के जीनोम में परिवर्तनशील प्रतिलिपि संख्याओं (कॉपी संख्या भिन्नता) से होता है।[16][17][18] चूँकि, इस प्रकार के डुप्लीकेशन की दर को मापना कठिन हो गया है। वर्तमान के अध्ययनों से सी एलिगेंस में जीन डुप्लीकेशन की जीनोम-व्यापी दर का प्रथम प्रत्यक्ष अनुमान प्राप्त हुआ। प्रथम बहुकोशिकीय यूकेरियोट जिसके लिए ऐसा अनुमान उपलब्ध हुआ। सी एलिगेंस में जीन डुप्लीकेशन दर 10−7 डुप्लीकेशन/जीन/पीढ़ी, अर्थात, 10 मिलियन कृमियों की जनसंख्या में, प्रति पीढ़ी जीन डुप्लीकेशन होगा। यह दर इस प्रजाति में प्रति न्यूक्लियोटाइड साइट पर बिंदु उत्परिवर्तन की सहज दर से दो गुना अधिक है।[19] प्राचीन (अप्रत्यक्ष) अध्ययनों ने बैक्टीरिया, ड्रोसोफिला और मनुष्यों में स्थान-विशिष्ट डुप्लीकेशन दर 10−3 से 10−7/जीन/पीढ़ी तक बताई गई है।[20][21][22]
नियोफ़ंक्शनलाइज़ेशन
जीन डुप्लीकेशन जीन नवीनता का आवश्यक स्रोत है जो विकासवादी नवाचार को उत्पन्न कर सकता है। डुप्लीकेशन जीन अतिरेक उत्पन्न करता है, जहां जीन की दूसरी प्रति प्रायः शुद्ध चयन से मुक्त होती है - अर्थात, इसके उत्परिवर्तन का इसके मेजबान जीव पर कोई हानिकारक प्रभाव नहीं पड़ता है। यदि जीन की एक प्रति में उत्परिवर्तन होता है जो उसके मूल कार्य को प्रभावित करता है, तो दूसरी प्रति 'अतिरिक्त भाग' के रूप में कार्य कर सकती है और उचित प्रकार से कार्य करना निरंतर रख सकती है। इस प्रकार, डुप्लिकेट जीन जीवों की पीढ़ियों के समय कार्यात्मक एकल-प्रतिलिपि जीन की तुलना में तीव्रता से उत्परिवर्तन एकत्र करते हैं, और दो प्रतियों में से एक के लिए नया और भिन्न कार्य विकसित करना संभव है। इस प्रकार के नियोफंक्शनलाइजेशन के कुछ उदाहरण बर्फ की मछली के परिवार में डुप्लिकेट पाचन जीन का एंटीफ्रीज जीन में स्पष्ट उत्परिवर्तन और डुप्लिकेशन से उपन्यास सांप जहर जीन की ओर अग्रसर होता है।[23] और सूअरों में 1 बीटा-हाइड्रॉक्सीटेस्टोस्टेरोन का संश्लेषण होता है।[24]
माना जाता है कि जीन डुप्लीकेशन विकास में प्रमुख भूमिका निभाता है; यह रुख वैज्ञानिक समुदाय के सदस्यों द्वारा 100 से अधिक वर्षों से अपनाया गया है।[25] सुसुमु ओहनो अपनी क्लासिक पुस्तक इवोल्यूशन बाय जीन डुप्लिकेशन (1970) में इस सिद्धांत के सबसे प्रसिद्ध डेवलपर्स में से थे।[26] ओहनो ने तर्क दिया कि सार्वभौमिक सामान्य पूर्वज के उद्भव के पश्चात से जीन डुप्लीकेशन सबसे महत्वपूर्ण विकासवादी शक्ति है।[27] प्रमुख जीनोम डुप्लीकेशन की घटनाएं अधिक सामान्य हो सकती हैं। ऐसा माना जाता है कि लगभग 100 मिलियन वर्ष पूर्व संपूर्ण यीस्ट जीनोम का डुप्लीकेशन हुआ था।[28] पौधे विपुल जीनोम अनुलिपित्र हैं। उदाहरण के लिए, गेहूं हेक्साप्लोइड (एक प्रकार का पॉलीप्लॉइड) है, जिसका अर्थ है कि इसके जीनोम की छह प्रतियां हैं।
सबफ़ंक्शनलाइज़ेशन
डुप्लिकेट जीन के लिए संभावित भाग्य यह है कि दोनों प्रतियां अपक्षयी उत्परिवर्तन एकत्र करने के लिए समान रूप से स्वतंत्र हैं, जब तक कि कोई भी दोष दूसरी प्रतिलिपि द्वारा पूरक हो। यह तटस्थ सबफ़ंक्शनलाइज़ेशन (रचनात्मक तटस्थ विकास की प्रक्रिया) या डीडीसी (दोहराव-अध:करण-पूरक) प्रारूप की ओर ले जाता है,[29][30] जिसमें मूल जीन की कार्यक्षमता दो प्रतियों के मध्य वितरित की जाती है। कोई भी जीन नष्ट नहीं हो सकता, क्योंकि दोनों अब महत्वपूर्ण गैर-अनावश्यक कार्य करते हैं, किन्तु अंततः कोई भी नवीन कार्यक्षमता प्राप्त करने में सक्षम नहीं है।
सबफ़ंक्शनलाइज़ेशन तटस्थ प्रक्रियाओं के माध्यम से हो सकता है जिसमें उत्परिवर्तन बिना किसी हानिकारक या लाभकारी प्रभाव के एकत्र होते हैं। चूँकि, कुछ स्थितियों में स्पष्ट अनुकूली लाभों के साथ सबफ़ंक्शनलाइज़ेशन हो सकता है। यदि पैतृक जीन प्लियोट्रोपिक है और दो कार्य करता है, तो प्रायः इन दोनों कार्यों में से किसी एक को दूसरे कार्य को प्रभावित किए बिना परिवर्तित नहीं किया जा सकता है। इस प्रकार, पैतृक कार्यों को दो भिन्न-भिन्न जीनों में विभाजित करने से उप-कार्यों के अनुकूली विशेषज्ञता की अनुमति मिल सकती है, जिससे अनुकूली लाभ मिलता है।[31]
हानि
प्रायः परिणामी जीनोमिक भिन्नता जीन मात्रा पर निर्भर न्यूरोलॉजिकल विकारों जैसे रेट-लाइक सिंड्रोम और पेलिज़ियस-मर्ज़बैकर रोग की ओर ले जाती है।[32] इस प्रकार के हानिकारक उत्परिवर्तन जनसंख्या से लुप्त हो जाने की संभावना है और इन्हें संरक्षित नहीं किया जाएगा या नवीन कार्यों का विकास नहीं किया जाएगा। चूँकि, कई डुप्लीकेशन, वास्तव में, हानिकारक या लाभकारी नहीं हैं, और ये तटस्थ अनुक्रम लुप्त हो सकते हैं या जीन बहाव के माध्यम से यादृच्छिक उतार-चढ़ाव के माध्यम से जनसंख्या में विस्तारित हो सकते हैं।
अनुक्रमित जीनोम में डुप्लीकेशन की पहचान करना
मानदंड और एकल जीनोम स्कैन
जीन डुप्लीकेशन की घटना के पश्चात उपस्थित दो जीनों को पैरालॉग कहा जाता है और सामान्यतः समान कार्य और संरचना वाले प्रोटीन के लिए कोड होते हैं। इसके विपरीत, ऑर्थोलॉगस जीन विभिन्न प्रजातियों में उपस्थित होते हैं, जो मूल रूप से एक ही पैतृक अनुक्रम से प्राप्त होते हैं। (आनुवांशिकी में अनुक्रमों की समरूपता देखें)।
जैविक अनुसंधान में पैरालॉग और ऑर्थोलॉग के मध्य अंतर करना महत्वपूर्ण (किन्तु प्रायः कठिन) होता है। मानव जीन फ़ंक्शन पर प्रयोग प्रायः अन्य प्रजातियों पर किए जा सकते हैं यदि मानव जीन का होमोलॉग उस प्रजाति के जीनोम में पाया जा सकता है, किन्तु केवल तभी जब होमोलॉग ऑर्थोलॉगस हो। यदि वे परलोक हैं और जीन डुप्लीकेशन की घटना से उत्पन्न हुए हैं, तो उनके कार्यों के अधिक भिन्न होने की संभावना है। डुप्लिकेट जीन की एक या अधिक प्रतियां जो एक जीन परिवार का गठन करती हैं, ट्रांसपोज़ेबल तत्वों के सम्मिलन से प्रभावित हो सकती हैं जो उनके मध्य उनके अनुक्रम में महत्वपूर्ण भिन्नता का कारण बनती हैं और अंततः भिन्न विकास के लिए उत्तरदायी हो सकती हैं। यह उनके अनुक्रमों में कम या कोई समानता नहीं होने के कारण जीन डुप्लिकेट के होमोलॉग के मध्य जीन रूपांतरण की संभावना और दर को भी प्रस्तुत कर सकता है।
सभी एनोटेटेड जीन प्रारूपों की एक दूसरे से अनुक्रम तुलना के माध्यम से एकल जीनोम में पैरालॉग की पहचान की जा सकती है। इस प्रकार की तुलना प्राचीन डुप्लीकेशन की पहचान करने के लिए अनुवादित अमीनो अम्ल अनुक्रमों (जैसे BLASTp, tBLASTx) पर या अधिक वर्तमान डुप्लीकेशन की पहचान करने के लिए डीएनए न्यूक्लियोटाइड अनुक्रमों (जैसे BLASTn, मेगाब्लास्ट) पर की जा सकती है। जीन डुप्लीकेशन की पहचान करने के लिए अधिकांश अध्ययनों में पारस्परिक-सर्वश्रेष्ठ-हिट या फ़ज़ी पारस्परिक-सर्वश्रेष्ठ-हिट की आवश्यकता होती है, जहां अनुक्रम तुलना में प्रत्येक पैरालॉग को दूसरे का सबसे उचित युग्मन होना चाहिए।[33]
अधिकांश जीन डुप्लीकेशन कम प्रतिलिपि डुप्लीकेशन (एलसीआर) के रूप में उपस्थित होते हैं, अन्यथा ट्रांसपोज़ेबल तत्वों के जैसे अत्यधिक डुप्लीकेशन वाले अनुक्रम होते हैं। वे अधिकतर क्रोमोसोम के पेरीसेंट्रोनोमिक, सबटेलोमेरिक और इंटरस्टिशियल क्षेत्रों में पाए जाते हैं। कई एलसीआर, अपने आकार (>1Kb), समानता और अभिविन्यास के कारण, डुप्लीकेशन और विलोपन के लिए अतिसंवेदनशील होते हैं।
जीनोमिक माइक्रोएरे डुप्लीकेशन को ज्ञात करते हैं
जीनोमिक माइक्रोएरे जैसी प्रौद्योगिकी, जिन्हें एरे तुलनात्मक जीनोमिक हाइब्रिडाइजेशन (एरे सीजीएच) भी कहा जाता है, इसका उपयोग जीनोमिक डीएनए प्रतिरूपों से उच्च थ्रूपुट फैशन में क्रोमोसोमल असामान्यताओं, जैसे कि माइक्रोडुप्लीकेशन, को ज्ञात करने के लिए किया जाता है। विशेष रूप से, डीएनए माइक्रोएरे प्रौद्योगिकी एक साथ कई उपचारों या प्रायोगिक स्थितियों में हजारों जीनों की अभिव्यक्ति के स्तर का निरिक्षण कर सकती है, जिससे जीन डुप्लीकेशन या प्रजातिकरण के पश्चात जीन विनियमन के विकासवादी अध्ययन में अधिक सुविधा होती है।[34][35]
अगली पीढ़ी का अनुक्रमण
अगली पीढ़ी के अनुक्रमण प्लेटफार्मों के उपयोग के माध्यम से जीन डुप्लीकेशन की भी पहचान की जा सकती है। जीनोमिक रीसेक्वेंसिंग डेटा में डुप्लीकेशन की पहचान करने का सबसे सरल साधन युग्मित-अंत अनुक्रमण रीडिंग का उपयोग है। अग्रानुक्रम डुप्लीकेशन को पढ़ने वाले जोड़े को अनुक्रमित करके प्रदर्शित किया जाता है जो असामान्य अभिविन्यास में मैप करते हैं। बढ़े हुए अनुक्रम कवरेज और असामान्य मानचित्रण अभिविन्यास के संयोजन के माध्यम से, जीनोमिक अनुक्रमण डेटा में डुप्लीकेशन की पहचान करना संभव है।
नामपद्धति
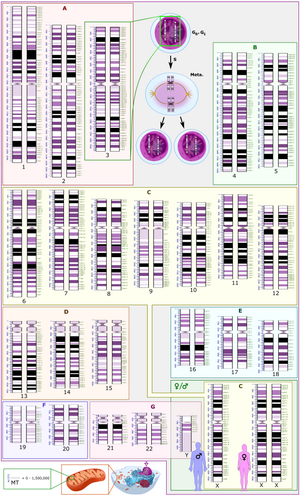
मानव साइटोजेनोमिक नामकरण के लिए अंतर्राष्ट्रीय प्रणाली (आईएससीएन) मानव गुणसूत्र नामकरण के लिए अंतरराष्ट्रीय मानक है, जिसमें मानव गुणसूत्र और गुणसूत्र असामान्यताओं के विवरण में उपयोग किए जाने वाले बैंड नाम, प्रतीक और संक्षिप्त शब्द सम्मिलित हैं। संक्षिप्ताक्षरों में गुणसूत्र के भागों के डुप्लीकेशन के लिए डुप सम्मिलित है।[36] उदाहरण के लिए, डुप(17पी12) चारकोट-मैरी-टूथ रोग प्रकार 1ए का कारण बनता है।[37]
प्रवर्धन के रूप में
जीन डुप्लीकेशन से किसी प्रजाति के जीनोम में स्थायी परिवर्तन होना आवश्यक नहीं है। वास्तव में, ऐसे परिवर्तन प्रायः प्रारंभिक मेजबान जीव से आगे नहीं रहते हैं। आणविक आनुवंशिकी दृष्टिकोण से, जीन प्रवर्धन उन कई प्रकारों में से है जिसमें जीन को अत्यधिक अभिव्यक्त किया जा सकता है। जीन प्रवर्धन कृत्रिम रूप से हो सकता है, जैसे कि एंजाइमों का उपयोग करके विट्रो में डीएनए के छोटे स्ट्रैंड को बढ़ाने के लिए पोलीमरेज़ चेन रिएक्शन प्रौद्योगिकी का उपयोग किया जाता है, या यह स्वाभाविक रूप से हो सकता है, जैसा कि ऊपर वर्णित है। यदि यह प्राकृतिक डुप्लीकेशन है, तो यह अभी भी रोगाणु कोशिका के अतिरिक्त दैहिक कोशिका में हो सकता है (जो स्थायी विकासवादी परिवर्तन के लिए आवश्यक होगा)।
कैंसर में भूमिका
ओंकोजीन का डुप्लीकेशन कई प्रकार के कैंसर का सामान्य कारण है। ऐसी स्थितियों में जीन डुप्लीकेशन दैहिक कोशिका में होता है और केवल कैंसर कोशिकाओं के जीनोम को प्रभावित करता है, पूर्ण जीव को नहीं, पश्चात की संतानों को तो बिल्कुल भी प्रभावित नहीं करता है। वर्तमान में व्यापक रोगी-स्तरीय वर्गीकरण और टीसीजीए समूहों में ड्राइवर घटनाओं के परिमाणीकरण से ज्ञात हुआ है कि प्रति ट्यूमर औसतन 12 ड्राइवर घटनाएं होती हैं, जिनमें से 1.5 ऑन्कोजीन के प्रवर्धन हैं।[38]
| कैंसर का प्रकार | संबद्ध जीन
प्रवर्धन |
इसकी प्रधानता
विस्तारण कैंसर के प्रकार में (प्रतिशत) |
|---|---|---|
| स्तन कैंसर | एमवाईसी | 20%[39] |
| ईआरबीबी2 (एचईआर2) | 20%[39] | |
| सीसीएनडी1 (साइक्लिन डी1) | 15–20%[39] | |
| एफजीएफआर1 | 12%[39] | |
| एफजीएफआर2 | 12%[39] | |
| सर्वाइकल कैंसर | एमवाईसी | 25–50%[39] |
| ईआरबीबी2 | 20%[39] | |
| कोलोरेक्टल कैंसर | एचआरएएस | 30%[39] |
| केआरएएस | 20%[39] | |
| एमवाईबी | 15–20%[39] | |
| एसोफेजल कैंसर | एमवाईसी | 40%[39] |
| सीसीएनडी1 | 25%[39] | |
| एमडीएम2 | 13%[39] | |
| अमाशय का कैंसर | सीसीएनई (साइक्लिन ई) | 15%[39] |
| केआरएएस | 10%[39] | |
| एमइटी | 10%[39] | |
| ग्लयोब्लास्टोमा | ईआरबीबी1 (ईजीएफआर) | 33–50%[39] |
| सीडीके4 | 15%[39] | |
| सिर और गर्दन का कैंसर | सीसीएनडी1 | 50%[39] |
| ईआरबीबी1 | 10%[39] | |
| एमवाईसी | 7–10%[39] | |
| हेपेटोसेल्यूलर कैंसर | सीसीएनडी1 | 13%[39] |
| न्यूरोब्लास्टोमा | एमवाईसीएन | 20–25%[39] |
| अंडाशयी कैंसर | एमवाईसी | 20–30%[39] |
| ईआरबीबी2 | 15–30%[39] | |
| एकेटी2 | 12%[39] | |
| सार्कोमा | एमडीएम2 | 10–30%[39] |
| सीडीके4 | 10%[39] | |
| लघु कोशिका फेफड़ों का कैंसर | एमवाईसी | 15–20%[39] |
संपूर्ण-जीनोम डुप्लीकेशन का उपयोग प्रायः कैंसर में होता है, सबसे सामान्य प्रकार के कैंसर के 30% से 36% ट्यूमर में इसको ज्ञात किया जाता है।[40][41] कार्सिनोजेनेसिस में उनकी त्रुटिहीन भूमिका स्पष्ट नहीं है, किन्तु कुछ स्थितियों में वे क्रोमैटिन पृथक्करण की हानि का कारण बनते हैं जिससे क्रोमैटिन संरचना में परिवर्तन होता है जो विपरीत में ऑन्कोजेनिक एपिजेनेटिक और ट्रांसक्रिप्शनल संशोधनों को उत्पन्न करता है।[42]
यह भी देखें
संदर्भ
- ↑ Zhang J (2003). "जीन दोहराव द्वारा विकास: एक अद्यतन" (PDF). Trends in Ecology & Evolution. 18 (6): 292–8. doi:10.1016/S0169-5347(03)00033-8.
- ↑ "जीन दोहराव की परिभाषा". medterms medical dictionary. MedicineNet. 2012-03-19.
- ↑ Miller, Duncan; Chen, Jianhai; Liang, Jiangtao; Betrán, Esther; Long, Manyuan; Sharakhov, Igor V. (2022-05-28). "मलेरिया के मच्छरों में सेक्स क्रोमोसोम के विकास द्वारा आकारित रेट्रोजीन दोहराव और अभिव्यक्ति पैटर्न". Genes. 13 (6): 968. doi:10.3390/genes13060968. ISSN 2073-4425. PMC 9222922. PMID 35741730.
- ↑ Dehal P, Boore JL (October 2005). "पैतृक कशेरुक में संपूर्ण जीनोम दोहराव के दो दौर". PLOS Biology. 3 (10): e314. doi:10.1371/journal.pbio.0030314. PMC 1197285. PMID 16128622.
- ↑ Wolfe, K. H.; Shields, D. C. (1997-06-12). "संपूर्ण यीस्ट जीनोम के प्राचीन दोहराव के लिए आणविक साक्ष्य". Nature. 387 (6634): 708–713. Bibcode:1997Natur.387..708W. doi:10.1038/42711. ISSN 0028-0836. PMID 9192896. S2CID 4307263.
- ↑ Kellis, Manolis; Birren, Bruce W.; Lander, Eric S. (2004-04-08). "यीस्ट सैक्रोमाइसेस सेरेविसिया में प्राचीन जीनोम दोहराव का प्रमाण और विकासवादी विश्लेषण". Nature. 428 (6983): 617–624. Bibcode:2004Natur.428..617K. doi:10.1038/nature02424. ISSN 1476-4687. PMID 15004568. S2CID 4422074.
- ↑ Otto, Sarah P. (2007-11-02). "पॉलीप्लोइडी के विकासवादी परिणाम". Cell. 131 (3): 452–462. doi:10.1016/j.cell.2007.10.022. ISSN 0092-8674. PMID 17981114. S2CID 10054182.
- ↑ Conant, Gavin C.; Wolfe, Kenneth H. (April 2006). "जीनोम दोहराव के बाद यीस्ट सह-अभिव्यक्ति नेटवर्क का कार्यात्मक विभाजन". PLOS Biology. 4 (4): e109. doi:10.1371/journal.pbio.0040109. ISSN 1545-7885. PMC 1420641. PMID 16555924.
- ↑ Papp, Balázs; Pál, Csaba; Hurst, Laurence D. (2003-07-10). "खुराक संवेदनशीलता और खमीर में जीन परिवारों का विकास". Nature. 424 (6945): 194–197. Bibcode:2003Natur.424..194P. doi:10.1038/nature01771. ISSN 1476-4687. PMID 12853957. S2CID 4382441.
- ↑ Lynch, M.; Conery, J. S. (2000-11-10). "डुप्लिकेट जीन का विकासवादी भाग्य और परिणाम". Science. 290 (5494): 1151–1155. Bibcode:2000Sci...290.1151L. doi:10.1126/science.290.5494.1151. ISSN 0036-8075. PMID 11073452.
- ↑ Freeling, Michael; Thomas, Brian C. (July 2006). "टेट्राप्लोइडी की तरह जीन-संतुलित दोहराव, रूपात्मक जटिलता को बढ़ाने के लिए पूर्वानुमानित ड्राइव प्रदान करता है". Genome Research. 16 (7): 805–814. doi:10.1101/gr.3681406. ISSN 1088-9051. PMID 16818725.
- ↑ Davis, Jerel C.; Petrov, Dmitri A. (October 2005). "Do disparate mechanisms of duplication add similar genes to the genome?". Trends in Genetics. 21 (10): 548–551. doi:10.1016/j.tig.2005.07.008. ISSN 0168-9525. PMID 16098632.
- ↑ Casneuf, Tineke; De Bodt, Stefanie; Raes, Jeroen; Maere, Steven; Van de Peer, Yves (2006). "फूल वाले पौधे अरेबिडोप्सिस थालियाना में जीन और जीनोम दोहराव के बाद जीन अभिव्यक्ति का गैर-यादृच्छिक विचलन". Genome Biology. 7 (2): R13. doi:10.1186/gb-2006-7-2-r13. ISSN 1474-760X. PMC 1431724. PMID 16507168.
- ↑ Li, Wen-Hsiung; Yang, Jing; Gu, Xun (November 2005). "डुप्लिकेट जीन के बीच अभिव्यक्ति विचलन". Trends in Genetics. 21 (11): 602–607. doi:10.1016/j.tig.2005.08.006. ISSN 0168-9525. PMID 16140417.
- ↑ 15.0 15.1 Amoutzias, Grigoris D.; He, Ying; Gordon, Jonathan; Mossialos, Dimitris; Oliver, Stephen G.; Van de Peer, Yves (2010-02-16). "पोस्टट्रांसलेशनल विनियमन डुप्लिकेट जीन के भाग्य को प्रभावित करता है". Proceedings of the National Academy of Sciences of the United States of America. 107 (7): 2967–2971. Bibcode:2010PNAS..107.2967A. doi:10.1073/pnas.0911603107. ISSN 1091-6490. PMC 2840353. PMID 20080574.
- ↑ Sebat J, Lakshmi B, Troge J, Alexander J, Young J, Lundin P, et al. (July 2004). "मानव जीनोम में बड़े पैमाने पर प्रतिलिपि संख्या बहुरूपता". Science. 305 (5683): 525–8. Bibcode:2004Sci...305..525S. doi:10.1126/science.1098918. PMID 15273396. S2CID 20357402.
- ↑ Iafrate AJ, Feuk L, Rivera MN, Listewnik ML, Donahoe PK, Qi Y, et al. (September 2004). "मानव जीनोम में बड़े पैमाने पर भिन्नता का पता लगाना". Nature Genetics. 36 (9): 949–51. doi:10.1038/ng1416. PMID 15286789.
- ↑ Emerson JJ, Cardoso-Moreira M, Borevitz JO, Long M (June 2008). "प्राकृतिक चयन ड्रोसोफिला मेलानोगास्टर में प्रतिलिपि-संख्या बहुरूपता के जीनोम-विस्तृत पैटर्न को आकार देता है". Science. 320 (5883): 1629–31. Bibcode:2008Sci...320.1629E. doi:10.1126/science.1158078. PMID 18535209. S2CID 206512885.
- ↑ Lipinski KJ, Farslow JC, Fitzpatrick KA, Lynch M, Katju V, Bergthorsson U (February 2011). "कैनोर्हाडाइटिस एलिगेंस में जीन दोहराव की उच्च सहज दर". Current Biology. 21 (4): 306–10. doi:10.1016/j.cub.2011.01.026. PMC 3056611. PMID 21295484.
- ↑ Anderson P, Roth J (May 1981). "साल्मोनेला टाइफिम्यूरियम में सहज अग्रानुक्रम आनुवंशिक दोहराव आरआरएनए (आरआरएन) सिस्ट्रोन के बीच असमान पुनर्संयोजन से उत्पन्न होता है". Proceedings of the National Academy of Sciences of the United States of America. 78 (5): 3113–7. Bibcode:1981PNAS...78.3113A. doi:10.1073/pnas.78.5.3113. PMC 319510. PMID 6789329.
- ↑ Watanabe Y, Takahashi A, Itoh M, Takano-Shimizu T (March 2009). "ड्रोसोफिला मेलानोगास्टर की नर और मादा जर्मलाइन कोशिकाओं में सहज डे नोवो उत्परिवर्तन का आणविक स्पेक्ट्रम". Genetics. 181 (3): 1035–43. doi:10.1534/genetics.108.093385. PMC 2651040. PMID 19114461.
- ↑ Turner DJ, Miretti M, Rajan D, Fiegler H, Carter NP, Blayney ML, et al. (January 2008). "डे नोवो मेयोटिक विलोपन और दोहराव की रोगाणु दर कई जीनोमिक विकारों का कारण बनती है". Nature Genetics. 40 (1): 90–5. doi:10.1038/ng.2007.40. PMC 2669897. PMID 18059269.
- ↑ Lynch VJ (January 2007). "Inventing an arsenal: adaptive evolution and neofunctionalization of snake venom phospholipase A2 genes". BMC Evolutionary Biology. 7: 2. doi:10.1186/1471-2148-7-2. PMC 1783844. PMID 17233905.
- ↑ Conant GC, Wolfe KH (December 2008). "Turning a hobby into a job: how duplicated genes find new functions". Nature Reviews. Genetics. 9 (12): 938–50. doi:10.1038/nrg2482. PMID 19015656. S2CID 1240225.
- ↑ Taylor JS, Raes J (2004). "दोहराव और विचलन: नए जीन और पुराने विचारों का विकास". Annual Review of Genetics. 38: 615–43. doi:10.1146/annurev.genet.38.072902.092831. PMID 15568988.
- ↑ Ohno, S. (1970). जीन दोहराव द्वारा विकास. Springer-Verlag. ISBN 978-0-04-575015-3.
- ↑ Ohno, S. (1967). सेक्स क्रोमोसोम और सेक्स-लिंक्ड जीन. Springer-Verlag. ISBN 978-91-554-5776-1.
- ↑ Kellis M, Birren BW, Lander ES (April 2004). "यीस्ट सैक्रोमाइसेस सेरेविसिया में प्राचीन जीनोम दोहराव का प्रमाण और विकासवादी विश्लेषण". Nature. 428 (6983): 617–24. Bibcode:2004Natur.428..617K. doi:10.1038/nature02424. PMID 15004568. S2CID 4422074.
- ↑ Force A, Lynch M, Pickett FB, Amores A, Yan YL, Postlethwait J (April 1999). "पूरक, अपक्षयी उत्परिवर्तन द्वारा डुप्लिकेट जीन का संरक्षण". Genetics. 151 (4): 1531–45. doi:10.1093/genetics/151.4.1531. PMC 1460548. PMID 10101175.
- ↑ Stoltzfus A (August 1999). "रचनात्मक तटस्थ विकास की संभावना पर". Journal of Molecular Evolution. 49 (2): 169–81. Bibcode:1999JMolE..49..169S. CiteSeerX 10.1.1.466.5042. doi:10.1007/PL00006540. PMID 10441669. S2CID 1743092.
- ↑ Des Marais DL, Rausher MD (August 2008). "एंथोसायनिन पाथवे जीन में दोहराव के बाद अनुकूली संघर्ष से बचें". Nature. 454 (7205): 762–5. Bibcode:2008Natur.454..762D. doi:10.1038/nature07092. PMID 18594508. S2CID 418964.
- ↑ Lee JA, Lupski JR (October 2006). "तंत्रिका तंत्र विकारों के कारण के रूप में जीनोमिक पुनर्व्यवस्था और जीन कॉपी-संख्या परिवर्तन". Neuron. 52 (1): 103–21. doi:10.1016/j.neuron.2006.09.027. PMID 17015230. S2CID 22412305.
- ↑ Hahn MW, Han MV, Han SG (November 2007). "Gene family evolution across 12 Drosophila genomes". PLOS Genetics. 3 (11): e197. doi:10.1371/journal.pgen.0030197. PMC 2065885. PMID 17997610.
- ↑ Mao R, Pevsner J (2005). "मानसिक मंदता में गुणसूत्र संबंधी असामान्यताओं का अध्ययन करने के लिए जीनोमिक माइक्रोएरे का उपयोग". Mental Retardation and Developmental Disabilities Research Reviews. 11 (4): 279–85. doi:10.1002/mrdd.20082. PMID 16240409.
- ↑ Gu X, Zhang Z, Huang W (January 2005). "यीस्ट जीन दोहराव के बाद अभिव्यक्ति और नियामक विचलन का तेजी से विकास". Proceedings of the National Academy of Sciences of the United States of America. 102 (3): 707–12. Bibcode:2005PNAS..102..707G. doi:10.1073/pnas.0409186102. PMC 545572. PMID 15647348.
- ↑ "आईएससीएन प्रतीक और संक्षिप्त शर्तें". Coriell Institute for Medical Research. Retrieved 2022-10-27.
- ↑ Cassandra L. Kniffin. "HARCOT-MARIE-TOOTH DISEASE, DEMYELINATING, TYPE 1A; CMT1A". OMIM. Updated : 4/23/2014
- ↑ Vyatkin, Alexey D.; Otnyukov, Danila V.; Leonov, Sergey V.; Belikov, Aleksey V. (14 January 2022). "TCGA PanCanAtlas समूहों में ड्राइवर घटनाओं का व्यापक रोगी-स्तरीय वर्गीकरण और परिमाणीकरण". PLOS Genetics. 18 (1): e1009996. doi:10.1371/journal.pgen.1009996. PMC 8759692. PMID 35030162.
- ↑ 39.00 39.01 39.02 39.03 39.04 39.05 39.06 39.07 39.08 39.09 39.10 39.11 39.12 39.13 39.14 39.15 39.16 39.17 39.18 39.19 39.20 39.21 39.22 39.23 39.24 39.25 39.26 39.27 39.28 Kinzler KW, Vogelstein B (2002). The genetic basis of human cancer. McGraw-Hill. p. 116. ISBN 978-0-07-137050-9.
- ↑ Bielski, Craig M.; Zehir, Ahmet; Penson, Alexander V.; Donoghue, Mark T. A.; Chatila, Walid; Armenia, Joshua; Chang, Matthew T.; Schram, Alison M.; Jonsson, Philip; Bandlamudi, Chaitanya; Razavi, Pedram; Iyer, Gopa; Robson, Mark E.; Stadler, Zsofia K.; Schultz, Nikolaus (2018). "जीनोम दोहरीकरण उन्नत कैंसर के विकास और पूर्वानुमान को आकार देता है". Nature Genetics (in English). 50 (8): 1189–1195. doi:10.1038/s41588-018-0165-1. ISSN 1546-1718.
- ↑ Quinton, Ryan J.; DiDomizio, Amanda; Vittoria, Marc A.; Kotýnková, Kristýna; Ticas, Carlos J.; Patel, Sheena; Koga, Yusuke; Vakhshoorzadeh, Jasmine; Hermance, Nicole; Kuroda, Taruho S.; Parulekar, Neha; Taylor, Alison M.; Manning, Amity L.; Campbell, Joshua D.; Ganem, Neil J. (2021). "संपूर्ण-जीनोम दोहरीकरण ट्यूमर कोशिकाओं पर अद्वितीय आनुवंशिक कमजोरियाँ प्रदान करता है". Nature (in English). 590 (7846): 492–497. doi:10.1038/s41586-020-03133-3. ISSN 1476-4687.
- ↑ Lambuta, Ruxandra A.; Nanni, Luca; Liu, Yuanlong; Diaz-Miyar, Juan; Iyer, Arvind; Tavernari, Daniele; Katanayeva, Natalya; Ciriello, Giovanni; Oricchio, Elisa (2023-03-15). "संपूर्ण-जीनोम दोहरीकरण से क्रोमैटिन पृथक्करण का ऑन्कोजेनिक नुकसान होता है". Nature (in English): 1–9. doi:10.1038/s41586-023-05794-2. ISSN 1476-4687.

