अभिकलनात्मक जीवविज्ञान (कम्प्यूटेशनल बायोलॉजी): Difference between revisions
No edit summary |
m (24 revisions imported from alpha:अभिकलनात्मक_जीवविज्ञान_(कम्प्यूटेशनल_बायोलॉजी)) |
||
| (16 intermediate revisions by 3 users not shown) | |||
| Line 1: | Line 1: | ||
{{short description|Branch of biology}} | {{short description|Branch of biology}} | ||
[[File:Human Genome Project Timeline (26964377742).jpg|thumb|548x548px|यह समयरेखा 1865 के पश्चात् से आनुवंशिकी के संदर्भ में [[मानव जीनोम परियोजना|ह्यूमन जीनोम प्रोजेक्ट]] की वर्ष-दर-वर्ष प्रगति को प्रदर्शित करती है। 1990 से प्रारम्भ होकर, 1999 तक, क्रोमोजोम 22 पूर्ण प्रकार से सीक्वेन्सड फर्स्ट ह्यूमन क्रोमोजोम बन गया।]]'''कम्प्यूटेशनल बायोलॉजी''' | [[File:Human Genome Project Timeline (26964377742).jpg|thumb|548x548px|यह समयरेखा 1865 के पश्चात् से आनुवंशिकी के संदर्भ में [[मानव जीनोम परियोजना|ह्यूमन जीनोम प्रोजेक्ट]] की वर्ष-दर-वर्ष प्रगति को प्रदर्शित करती है। 1990 से प्रारम्भ होकर, 1999 तक, क्रोमोजोम 22 पूर्ण प्रकार से सीक्वेन्सड फर्स्ट ह्यूमन क्रोमोजोम बन गया।]]'''अभिकलनात्मक जीवविज्ञान (कम्प्यूटेशनल बायोलॉजी)''' बायोलॉजिकल सिस्टम्स और रिलेशनशिप का अध्ययन करने के लिए [[डेटा विश्लेषण|डेटा एनालिसिस]], मैथमेटिकल मॉडलिंग और [[कंप्यूटर सिमुलेशन]] के उपयोग को संदर्भित करती है।<ref name="nih" /> [[कंप्यूटर विज्ञान|कंप्यूटर साइंस]], जीव विज्ञान, और बिग डेटा का इंटरसेक्शन, इस क्षेत्र में अनुप्रयुक्त गणित, [[रसायन विज्ञान]] और [[आनुवंशिकी]] में भी आधार है।<ref name="brown" /> यह [[जैविक कंप्यूटिंग|बायोलॉजिकल कंप्यूटिंग]] से भिन्न है, [[कंप्यूटर]] इंजीनियरिंग का उपक्षेत्र जो कंप्यूटर बनाने के लिए बायोइंजीनियरिंग का उपयोग करता है। | ||
== इतिहास == | == इतिहास == | ||
बायोइन्फार्मेटिक्स, | बायोइन्फार्मेटिक्स, बायोलॉजिकल सिस्टम्स में इन्फार्मेटिक्स प्रोसेसों का एनालिसिस, 1970 के दशक में प्रारम्भ हुआ था। इस समय, [[कृत्रिम होशियारी|आर्टिफिशियल इंटेलिजेंस]] में अनुसंधान नए [[एल्गोरिदम]] जेनेरेट करने के लिए ह्यूमन ब्रेन के [[नेटवर्क मॉडल]] का उपयोग कर रहा था। बायोलॉजिकल डेटा के इस उपयोग ने जैविक शोधकर्ताओं को अपने क्षेत्र में लार्ज डेटा सेटों को इवैल्युएट और कम्पेयर करने के लिए कंप्यूटर का उपयोग करने के लिए प्रेरित किया था।<ref name="Hogeweg 2011">{{cite journal |last=Hogeweg |first=Paulien |date=7 March 2011 |title=The Roots of Bioinformatics in Theoretical Biology |journal=PLOS Computational Biology |series=3 |volume=7 |issue=3 |pages=e1002021 |bibcode=2011PLSCB...7E2021H |doi=10.1371/journal.pcbi.1002021 |pmc=3068925 |pmid=21483479}}</ref> 1982 तक, शोधकर्ताओं ने [[छिद्रित कार्ड|पंच कार्ड]] के माध्यम से इनफार्मेशन शेयर की थी। 1980 के दशक के अंत तक डेटा के अमाउंट में तीव्रता से वृद्धि हुई, जिससे क्विकली इंटरप्रेटिंग रिलेवेंट इनफार्मेशन के लिए नई कम्प्यूटेशनल विधियों की आवश्यकता हुई।<ref name="Hogeweg 2011"/> | ||
कम्प्यूटेशनल बायोलॉजी का संभवतः सबसे प्रसिद्ध उदाहरण, ह्यूमन जीनोम प्रोजेक्ट, आधिकारिक रूप से 1990 में प्रारम्भ हुआ था।<ref name=":0">{{Cite web |date=22 December 2020 |title=The Human Genome Project |url=https://www.genome.gov/human-genome-project |access-date=13 April 2022 |website=The Human Genome Project}}</ref> 2003 तक, प्रोजेक्ट ने अपने प्रारंभिक लक्ष्यों को पूर्ण करते हुए ह्यूमन जीनोम का लगभग 85% मानचित्रण कर लिया था।<ref>{{Cite web |title=Human Genome Project FAQ |url=https://www.genome.gov/human-genome-project/Completion-FAQ |access-date=2022-04-20 |website=Genome.gov |language=en}}</ref> यद्यपि, कार्य प्रारम्भ रहा, और 2021 के स्तर तक पूर्ण जीनोम तक पहुँच गया, जिसमें केवल 0.3% शेष आधार संभावित अभिप्रायों से आच्छादित थे।<ref>{{Cite web |title=T2T-CHM13v1.1 - Genome - Assembly - NCBI |url=https://www.ncbi.nlm.nih.gov/assembly/GCA_009914755.3 |access-date=2022-04-20 |website=www.ncbi.nlm.nih.gov}}</ref><ref>{{Cite web |title=Genome List - Genome - NCBI |url=https://www.ncbi.nlm.nih.gov/genome/browse/#!/eukaryotes/51/ |access-date=2022-04-20 |website=www.ncbi.nlm.nih.gov}}</ref> मिसिंग Y क्रोमोजोम जनवरी 2022 में ऐड किया गया था। | कम्प्यूटेशनल बायोलॉजी का संभवतः सबसे प्रसिद्ध उदाहरण, ह्यूमन जीनोम प्रोजेक्ट, आधिकारिक रूप से 1990 में प्रारम्भ हुआ था।<ref name=":0">{{Cite web |date=22 December 2020 |title=The Human Genome Project |url=https://www.genome.gov/human-genome-project |access-date=13 April 2022 |website=The Human Genome Project}}</ref> 2003 तक, प्रोजेक्ट ने अपने प्रारंभिक लक्ष्यों को पूर्ण करते हुए ह्यूमन जीनोम का लगभग 85% मानचित्रण कर लिया था।<ref>{{Cite web |title=Human Genome Project FAQ |url=https://www.genome.gov/human-genome-project/Completion-FAQ |access-date=2022-04-20 |website=Genome.gov |language=en}}</ref> यद्यपि, कार्य प्रारम्भ रहा, और 2021 के स्तर तक पूर्ण जीनोम तक पहुँच गया, जिसमें केवल 0.3% शेष आधार संभावित अभिप्रायों से आच्छादित थे।<ref>{{Cite web |title=T2T-CHM13v1.1 - Genome - Assembly - NCBI |url=https://www.ncbi.nlm.nih.gov/assembly/GCA_009914755.3 |access-date=2022-04-20 |website=www.ncbi.nlm.nih.gov}}</ref><ref>{{Cite web |title=Genome List - Genome - NCBI |url=https://www.ncbi.nlm.nih.gov/genome/browse/#!/eukaryotes/51/ |access-date=2022-04-20 |website=www.ncbi.nlm.nih.gov}}</ref> मिसिंग Y क्रोमोजोम जनवरी 2022 में ऐड किया गया था। | ||
| Line 25: | Line 25: | ||
इन गणितीय दृष्टिकोणों ने बायोलॉजिकल डेटा को स्टोर करने, रिट्रीव करने और एनालाइज़ करने के लिए [[डेटाबेस]] और अन्य विधियों के निर्माण को सक्षम किया है, जिसे बायोइन्फार्मेटिक्स के रूप में जाना जाता है। सामान्यतः, इस प्रोसेस में जेनेटिक्स और [[जीन|जीन्स]] का एनालाइज़ेशन सम्मिलित होता है। | इन गणितीय दृष्टिकोणों ने बायोलॉजिकल डेटा को स्टोर करने, रिट्रीव करने और एनालाइज़ करने के लिए [[डेटाबेस]] और अन्य विधियों के निर्माण को सक्षम किया है, जिसे बायोइन्फार्मेटिक्स के रूप में जाना जाता है। सामान्यतः, इस प्रोसेस में जेनेटिक्स और [[जीन|जीन्स]] का एनालाइज़ेशन सम्मिलित होता है। | ||
लार्ज डेटासेट को गेदर करने और उसका एनालाइज़ेशन करने से [[डेटा खनन|डेटा माइनिंग]] और कम्प्यूटेशनल बायोमॉडलिंग जैसे बढ़ते शोध क्षेत्रों के लिए स्थान बन गया है,<ref name="nlcb.wordpress.com">{{Cite web |date=2013-02-18 |title=The Sub-fields of Computational Biology |url=https://nlcb.wordpress.com/2013/02/17/the-sub-fields-of-computational-biology/ |access-date=2022-04-18 |website=Ninh Laboratory of Computational Biology |language=en}}</ref> जो [[कंप्यूटर मॉडल]] और बायोलॉजिकल सिस्टम्स के [[संवर्धित वास्तविकता|विसुअल सिमुलेशन]] के निर्माण को संदर्भित करता है। यह शोधकर्ताओं को यह अनुमान लगाने की अनुमति देता है कि ऐसे सिस्टम्स डिफरेंट एनवायरनमेन्ट्स पर किस प्रकार रियेक्ट करते हैं, जो यह निर्धारित करने के लिए उपयोगी है कि क्या कोई सिस्टम एक्सटर्नल और इंटरनल पर्टरबेशन्स के विरुद्ध अपने स्टेट और फंक्शन्स को मेन्टेन रख सकता है।<ref name= Kitano 2002 206–10 >{{cite journal |last=Kitano |first=Hiroaki |date=14 November 2002 |title= | लार्ज डेटासेट को गेदर करने और उसका एनालाइज़ेशन करने से [[डेटा खनन|डेटा माइनिंग]] और कम्प्यूटेशनल बायोमॉडलिंग जैसे बढ़ते शोध क्षेत्रों के लिए स्थान बन गया है,<ref name="nlcb.wordpress.com">{{Cite web |date=2013-02-18 |title=The Sub-fields of Computational Biology |url=https://nlcb.wordpress.com/2013/02/17/the-sub-fields-of-computational-biology/ |access-date=2022-04-18 |website=Ninh Laboratory of Computational Biology |language=en}}</ref> जो [[कंप्यूटर मॉडल]] और बायोलॉजिकल सिस्टम्स के [[संवर्धित वास्तविकता|विसुअल सिमुलेशन]] के निर्माण को संदर्भित करता है। यह शोधकर्ताओं को यह अनुमान लगाने की अनुमति देता है कि ऐसे सिस्टम्स डिफरेंट एनवायरनमेन्ट्स पर किस प्रकार रियेक्ट करते हैं, जो यह निर्धारित करने के लिए उपयोगी है कि क्या कोई सिस्टम एक्सटर्नल और इंटरनल पर्टरबेशन्स के विरुद्ध अपने स्टेट और फंक्शन्स को मेन्टेन रख सकता है।<ref name="Kitano 2002 206–10">{{cite journal|last=Kitano|first=Hiroaki|date=14 November 2002|title=Computational systems biology|journal=Nature|volume=420|issue=6912|pages=206–10|bibcode=2002Natur.420..206K|doi=10.1038/nature01254|pmid=12432404|id={{ProQuest|204483859}}|s2cid=4401115}}</ref> कम्प्यूटेशनल बायोलॉजी ने प्रथम सिद्धांतों से [[प्राकृतिक चयन|एवोल्यूशनरी सिस्टम्स]] के [[जनसंख्या वृद्धि|पापुलेशन जेनेटिक्स]] मॉडल का निर्माण करके एवोल्यूशनरी बायोलॉजी की सहायता की है जिससे यह अनुमान लगाया जा सके कि क्या विकसित होने की संभावना है। | ||
=== जीनोमिक्स === | === जीनोमिक्स === | ||
[[File:Genome viewer screenshot small.png|thumbnail|right| | [[File:Genome viewer screenshot small.png|thumbnail|right|पार्शियली सीक्वेन्सड जीनोम।]] | ||
{{main|कम्प्यूटेशनल जीनोमिक्स}} | {{main|कम्प्यूटेशनल जीनोमिक्स}} | ||
कम्प्यूटेशनल [[जीनोम|जीनोमिक्स]] कोशिका ([[जीव]] विज्ञान) और जीवों के जीनोम का अध्ययन है। ह्यूमन जीनोम प्रोजेक्ट कम्प्यूटेशनल जीनोमिक्स का उदाहरण है। यह प्रोजेक्ट सम्पूर्ण ह्यूमन जीनोम को डेटा के एक सेट में सीक्वेंस करना चाहता है। पूर्ण रूप से प्रस्तावित होने पर, यह डॉक्टरों को व्यक्तिगत रोगी के जीनोम को एनालाइज़ करने की अनुमति दे सकता है।<ref>{{cite magazine|title=Genome Sequencing to the Rest of Us|url=http://www.scientificamerican.com/article.cfm?id=personal-genome-sequencing|magazine=Scientific American}}</ref> यह व्यक्तिगत चिकित्सा की संभावना को उन्मुक्त करता है, किसी व्यक्ति के प्री-एक्सिस्टिंग जेनेटिक पैटर्न के आधार पर उपचार निर्धारित करता है। शोधकर्ता जानवरों, पौधों, [[जीवाणु|जीवाणुओं]] और अन्य सभी प्रकार के जीवन के जीनोम को सीक्वेंस करने का प्रयास कर रहे हैं। | कम्प्यूटेशनल [[जीनोम|जीनोमिक्स]] कोशिका ([[जीव]] विज्ञान) और जीवों के जीनोम का अध्ययन है। ह्यूमन जीनोम प्रोजेक्ट कम्प्यूटेशनल जीनोमिक्स का उदाहरण है। यह प्रोजेक्ट सम्पूर्ण ह्यूमन जीनोम को डेटा के एक सेट में सीक्वेंस करना चाहता है। पूर्ण रूप से प्रस्तावित होने पर, यह डॉक्टरों को व्यक्तिगत रोगी के जीनोम को एनालाइज़ करने की अनुमति दे सकता है।<ref>{{cite magazine|title=Genome Sequencing to the Rest of Us|url=http://www.scientificamerican.com/article.cfm?id=personal-genome-sequencing|magazine=Scientific American}}</ref> यह व्यक्तिगत चिकित्सा की संभावना को उन्मुक्त करता है, किसी व्यक्ति के प्री-एक्सिस्टिंग जेनेटिक पैटर्न के आधार पर उपचार निर्धारित करता है। शोधकर्ता जानवरों, पौधों, [[जीवाणु|जीवाणुओं]] और अन्य सभी प्रकार के जीवन के जीनोम को सीक्वेंस करने का प्रयास कर रहे हैं। | ||
जिन मुख्य प्रकारों में जीनोम की उपमा की जाती है उनमें से एक [[अनुक्रम समरूपता|सीक्वेंस होमोलॉजी]] है। होमोलॉजी विभिन्न जीवों में जैविक संरचनाओं और न्यूक्लियोटाइड सीक्वेंसेस का अध्ययन है जो सामान्य [[पूर्वज]] से आते हैं। शोध से ज्ञात होता है कि न्यूली सीक्वेन्सड प्रोकैरियोट जीनोम में 80 से 90% जीनों की पहचान इस प्रकार से की जा सकती है।<ref name= Koonin 2001 155–158 /> | जिन मुख्य प्रकारों में जीनोम की उपमा की जाती है उनमें से एक [[अनुक्रम समरूपता|सीक्वेंस होमोलॉजी]] है। होमोलॉजी विभिन्न जीवों में जैविक संरचनाओं और न्यूक्लियोटाइड सीक्वेंसेस का अध्ययन है जो सामान्य [[पूर्वज]] से आते हैं। शोध से ज्ञात होता है कि न्यूली सीक्वेन्सड प्रोकैरियोट जीनोम में 80 से 90% जीनों की पहचान इस प्रकार से की जा सकती है।<ref name="Koonin 2001 155–158">{{cite journal|last=Koonin|first=Eugene|date=6 March 2001|title=Computational Genomics|journal=Curr. Biol.|volume=11|issue=5|pages=155–158|doi=10.1016/S0960-9822(01)00081-1|pmid=11267880|doi-access=free|s2cid=17202180}}</ref> | ||
[[अनुक्रम संरेखण]] | [[अनुक्रम संरेखण|सीक्वेंस एलाइनमेंट]] बायोलॉजिकल सीक्वेंसेस या जीनों के मध्य समानता की अपेक्षा और ज्ञात करने की प्रक्रिया है। सीक्वेंस एलाइनमेंट कई बायोइन्फार्मेटिक्स अनुप्रयोगों में उपयोगी है, जैसे कि दो जीनों की सबसे लंबी सामान्य अनुगामी समस्या की गणना करना या कुछ रोगों के वेरिएंट की उपमा करना। | ||
कम्प्यूटेशनल जीनोमिक्स में | कम्प्यूटेशनल जीनोमिक्स में अनटच प्रोजेक्ट इंटरजेनिक रिजनों का एनालिसिस है, जिसमें ह्यूमन जीनोम का लगभग 97% भाग सम्मिलित है। शोधकर्ता कम्प्यूटेशनल और सांख्यिकीय विधियों के विकास और [[ENCODE|एनकोड]] तथा रोडमैप एपिजेनोमिक्स प्रोजेक्ट जैसे बड़े संघ प्रोजेक्ट्स के माध्यम से ह्यूमन जीनोम के नॉन-कोडिंग रिजनों के फंक्शन्स को समझने के लिए कार्य कर रहे हैं। | ||
[[अणु]], कोशिका (जीव विज्ञान), और जीव स्तर पर किसी जीव के जीव विज्ञान में व्यक्तिगत जीन | [[अणु]], कोशिका (जीव विज्ञान), और जीव स्तर पर किसी जीव के जीव विज्ञान में व्यक्तिगत जीन किस प्रकार योगदान करते हैं, इसका अध्ययन करना [[जीन ओन्टोलॉजी]] के रूप में जाना जाता है। [[जीन ओन्टोलॉजी कंसोर्टियम]] का मिशन मॉलिक्यूलर लेवल से लेकर लार्जर पाथवेज़, सेलुलर और ऑर्गैनिस्म-लेवल सिस्टम्स तक [[शाही सेना|बायोलॉजिकल सिस्टम्स]] का अप-टू-डेट, कम्प्रेहैन्सिव, कम्प्यूटेशनल मॉडल विकसित करना है। जीन ओन्टोलॉजी रिसोर्स मनुष्यों से लेकर जीवाणुओं तक, कई भिन्न-भिन्न जीवों से जीन के कार्यों (या जीन द्वारा उत्पादित [[प्रोटीन]] और नॉन-कोडिंग आरएनए अणुओं) के संबंध में करंट साइंटिफिक नॉलेज का कम्प्यूटेशनल रिप्रजेंटेशन प्रदान करता है। | ||
3डी जीनोमिक्स कम्प्यूटेशनल बायोलॉजी में उपधारा है जो [[यूकेरियोटिक सेल]] के भीतर जीन के आर्गेनाईजेशन और इंटरेक्शन पर केंद्रित है। 3डी जीनोमिक डेटा गेदर करने के लिए उपयोग की जाने वाली विधि जीनोम आर्किटेक्चर मैपिंग (जीएएम) के माध्यम से है। जीएएम [[क्रायोसेक्शनिंग]], डीएनए का परीक्षण करने के लिए नाभिक से स्ट्रिप प्राप्त करने की प्रक्रिया, लेजर माइक्रोडिसेक्शन के साथ संयोजन करके जीनोम में [[क्रोमेटिन]] और डीएनए की 3डी दूरी को मापता है। न्यूक्लेयर प्रोफ़ाइल केवल यह स्ट्रिप अथवा स्लाइस है जो नाभिक से ली गई है। प्रत्येक न्यूक्लेयर प्रोफ़ाइल में जीनोमिक विंडो होती हैं, जो डीएनए की आधार इकाई [[न्यूक्लियोटाइड]] के कुछ सीक्वेंस होते हैं। जीएएम पूर्ण सेल में काम्प्लेक्स, मल्टी एनहांसर क्रोमैटिन कॉन्टेक्ट्स के जीनोम नेटवर्क को कैप्चर करता है। | |||
कम्प्यूटेशनल [[ | |||
कम्प्यूटेशनल [[neuropsychiatry]] | ===न्यूरोसाइंस=== | ||
{{main|कम्प्यूटेशनल न्यूरोसाइंस}} | |||
कम्प्यूटेशनल [[तंत्रिका विज्ञान|न्यूरोसाइंस]] [[तंत्रिका तंत्र|नर्वस सिस्टम]] के इनफार्मेशन प्रोसेसिंग गुणों के संदर्भ में ब्रेन के कार्य का अध्ययन है। न्यूरोसाइंस का सबसेट, यह न्यूरोलॉजिकल सिस्टम के विशिष्ट दृश्यों का परीक्षण करने के लिए ब्रेन को मॉडल करता है।<ref>{{Cite web |title=Computational Neuroscience | Neuroscience |url=http://www.bu.edu/neuro/academics/graduate/curriculum/computational-neuroscience/ |website=www.bu.edu}}</ref> ब्रेन के मॉडल में सम्मिलित हैं: | |||
* रीयलिस्टिक ब्रेन मॉडल: ये मॉडल ब्रेन के प्रत्येक एस्पेक्ट का प्रतिनिधित्व करते हैं, जिसमें कोशीय स्तर पर जितना संभव हो उतना विवरण सम्मिलित होता है। रीयलिस्टिक मॉडल ब्रेन के संबंध में सबसे अधिक इनफार्मेशन प्रदान करते हैं, किन्तु एरर के लिए सबसे बड़ा मार्जिन भी होता है। ब्रेन मॉडल में अधिक वेरिएबल्स अधिक एरर होने की संभावना उत्पन्न करते हैं। ये मॉडल कोशीय संरचना के उन भागों के संबंध में नहीं बताते हैं जिनके संबंध में वैज्ञानिक नहीं जानते हैं। रीयलिस्टिक ब्रेन मॉडल कम्प्यूटेशनल रूप से सबसे हैवी और इम्प्लीमेंट करने के लिए अधिक बहुमूल्य हैं।<ref name="Sejnowski 1988">{{cite journal|last=Sejnowski|first=Terrence |author2=Christof Koch |author3=Patricia S. Churchland|title=Computational Neuroscience|journal=Science |date=9 September 1988|volume=241|series=4871|issue=4871 |pages=1299–306 |doi=10.1126/science.3045969 |pmid=3045969 |bibcode=1988Sci...241.1299S }}</ref> | |||
*सिम्प्लिफायिंग ब्रेन मॉडल: ये मॉडल न्यूरोलॉजिकल सिस्टम की स्पेसिफिक फिजिकल प्रॉपर्टी का आकलन करने के लिए मॉडल के स्कोप को सीमित करने पर विचार करते हैं। यह इंटेंसिव कम्प्यूटेशनल प्रोब्लेम्स को सॉल्व करने की अनुमति देता है, और रीयलिस्टिक ब्रेन मॉडल से पोटेंशियल एरर के अमाउंट को कम करता है।<ref name="Sejnowski 1988" /> वर्तमान में ऐसी गणनाओं की गति में वृद्धि के लिए उपयोग किए जाने वाले एल्गोरिदम और डेटा स्ट्रक्चर्स में संशोधन करना कम्प्यूटेशनल न्यूरोसाइंटिस्ट का कार्य है। | |||
कम्प्यूटेशनल [[neuropsychiatry|न्यूरोसाइकिएट्री]] इमर्जिंग फील्ड है जो [[मानसिक विकार|मानसिक विकारों]] में सम्मिलित ब्रेन मैकेनिज्म की मैथमेटिकल और कंप्यूटर-असिस्टेड मॉडलिंग का उपयोग करता है। कई उपक्रमों ने प्रदर्शित किया है कि कम्प्यूटेशनल मॉडलिंग न्यूरोनल सर्किट के अध्ययन में महत्वपूर्ण योगदान है जो मानसिक कार्यों और शिथिलताओं को उत्पन्न कर सकता है।<ref>{{cite journal |last1=Dauvermann |first1=Maria R. |last2=Whalley |first2=Heather C. |last3=Schmidt |first3=Andrã© |last4=Lee |first4=Graham L. |last5=Romaniuk |first5=Liana |last6=Roberts |first6=Neil |last7=Johnstone |first7=Eve C. |last8=Lawrie |first8=Stephen M. |last9=Moorhead |first9=Thomas W. J. |year=2014 |title=Computational Neuropsychiatry – Schizophrenia as a Cognitive Brain Network Disorder |journal=Frontiers in Psychiatry |volume=5 |pages=30 |doi=10.3389/fpsyt.2014.00030 |pmc=3971172 |pmid=24723894 |doi-access=free}}</ref><ref>{{cite journal |last1=Tretter |first1=F. |last2=Albus |first2=M. |date=December 2007 |title='Computational Neuropsychiatry' of Working Memory Disorders in Schizophrenia: The Network Connectivity in Prefrontal Cortex - Data and Models |journal=Pharmacopsychiatry |volume=40 |issue=S 1 |pages=S2–S16 |doi=10.1055/S-2007-993139 |s2cid=18574327}}</ref><ref>{{cite journal |last1=Marin-Sanguino |first1=A. |last2=Mendoza |first2=E. |year=2008 |title=Hybrid Modeling in Computational Neuropsychiatry |journal=Pharmacopsychiatry |volume=41 |pages=S85–S88 |doi=10.1055/s-2008-1081464 |pmid=18756425}}</ref> | |||
'''फार्माकोलॉजी''' | '''फार्माकोलॉजी''' | ||
{{main| | {{main|फार्माकोलॉजी}} | ||
कम्प्यूटेशनल फार्माकोलॉजी विशिष्ट [[जीनोटाइप]] और | कम्प्यूटेशनल फार्माकोलॉजी विशिष्ट [[जीनोटाइप]] और रोगों और फिर स्क्रीनिंग ड्रग डाटा के मध्य संबंध प्राप्त के लिए जीनोमिक डेटा के प्रभावों का अध्ययन है।<ref>{{cite web|last=Price|first=Michael|title=Computational Biologists: The Next Pharma Scientists?|url=https://www.science.org/content/article/computational-biologists-next-pharma-scientists|date=2012-04-13}}</ref> [[दवा उद्योग|फार्मास्युटिकल उद्योग]] को ड्रग डेटा का एनालिसिस करने की विधियों में परिवर्तन करने की आवश्यकता है। फार्माकोलॉजिस्ट ड्रग की प्रभावशीलता से संबंधित रासायनिक और जीनोमिक डेटा की उपमा करने के लिए [[Microsoft Excel|माइक्रोसॉफ्ट एक्सेल]] का उपयोग करने में सक्षम थे। यद्यपि, उद्योग उस स्थिति तक पहुँच गया है जिसे एक्सेल बैरिकेड कहा जाता है। यह [[स्प्रेडशीट]] पर एक्सेसिबल सेल्स की सीमित संख्या से उत्पन्न होता है। इस विकास ने कम्प्यूटेशनल फार्माकोलॉजी की आवश्यकता को उत्पन्न किया था। वैज्ञानिक और शोधकर्ता इन मैसिव [[डेटा सेट|डेटा सेट्स]] का एनालिसिस करने के लिए कम्प्यूटेशनल विधि विकसित करते हैं। यह नोटेबल डेटा पॉइंट्स के मध्य कुशल उपमा की अनुमति देता है और अधिक एक्यूरेट ड्रग्स को विकसित करने की अनुमति देता है।<ref name="Walter">{{cite web|last=Jessen|first=Walter|title=Pharma's shifting strategy means more jobs for computational biologists|url=http://medcitynews.com/2012/04/pharmas-shifting-strategy-means-more-jobs-for-computational-biologists/|date=2012-04-15}}</ref> | ||
एनालिस्टों का अनुमान है कि यदि पेटेंट के कारण प्रमुख ड्रग्स विफल हो जाते हैं, तो मार्किट में उपस्थित ड्रगों को परिवर्तित करने के लिए कम्प्यूटेशनल बायोलॉजी की आवश्यकता होगी। कम्प्यूटेशनल बायोलॉजी में डॉक्टरेट छात्रों को डॉक्टरेट के पश्चात् के पदों को लेने के अतिरिक्त उद्योग में करियर बनाने के लिए प्रोत्साहित किया जा रहा है। यह नए ड्रग के उत्पादन के लिए आवश्यक बिग डेटा सेट के अधिक योग्य एनालिस्टों की आवश्यकता वाली प्रमुख ड्रग कंपनियों का प्रत्यक्ष परिणाम है।<ref name="Walter" /> | |||
== | इसी प्रकार, कम्प्यूटेशनल ऑन्कोलॉजी का उद्देश्य एल्गोरिथम एप्रोच के माध्यम से [[कैंसर]] में भविष्य के [[उत्परिवर्तन]] को निर्धारित करना है। इस क्षेत्र में अनुसंधान ने उच्च-थ्रूपुट मापन का उपयोग करने के लिए प्रेरित किया है जो [[रोबोटिक|रोबोटिक्स]] और अन्य सेंसिंग डिवाइसों का उपयोग करते हुए लाखों डेटा पॉइंट्स हैं। यह डेटा डीएनए, आरएनए और अन्य जैविक संरचनाओं से एकत्र किया जाता है। फोकस के क्षेत्रों में [[सूजन|ट्यूमर]] की विशेषताओं का निर्धारण करना, अणुओं का विश्लेषण करना जो कैंसर उत्पन्न करने में नियतात्मक हैं, और यह समझना कि ह्यूमन जीनोम ट्यूमर और कैंसर के कारण से कैसे संबंधित है।<ref>{{cite journal |last1=Barbolosi |first1=Dominique |last2=Ciccolini |first2=Joseph |last3=Lacarelle |first3=Bruno |last4=Barlesi |first4=Fabrice |last5=Andre |first5=Nicolas |year=2016 |title=Computational oncology--mathematical modelling of drug regimens for precision medicine |journal=Nature Reviews Clinical Oncology |volume=13 |issue=4 |pages=242–254 |doi=10.1038/nrclinonc.2015.204 |pmid=26598946 |s2cid=22492353}}</ref><ref>{{cite journal |last=Yakhini |first=Zohar |year=2011 |title=Cancer Computational Biology |journal=BMC Bioinformatics |volume=12 |pages=120 |doi=10.1186/1471-2105-12-120 |pmc=3111371 |pmid=21521513}}</ref> | ||
== टेक्निक्स == | |||
कम्प्यूटेशनल जीवविज्ञानी अपने शोध करने के लिए सॉफ्टवेयर और एल्गोरिदम की विस्तृत श्रृंखला का उपयोग करते हैं। | कम्प्यूटेशनल जीवविज्ञानी अपने शोध करने के लिए सॉफ्टवेयर और एल्गोरिदम की विस्तृत श्रृंखला का उपयोग करते हैं। | ||
=== | === अनसुपरवाइसड लर्निंग === | ||
[[अनियंत्रित शिक्षा]] | [[अनियंत्रित शिक्षा|अनसुपरवाइसड लर्निंग]] एल्गोरिदम है जो अनलेबेल्ड डेटा में पैटर्न फाइंड करता है। उदाहरण k-मीन्स क्लस्टरिंग है, जिसका उद्देश्य n डेटा पॉइंट्स को k समूहों में विभाजित करना है, जिसमें प्रत्येक डेटा पॉइंट निकटतम माध्य वाले क्लस्टर से संबंधित है। अन्य वर्जन [[k-मेडोइड्स]] एल्गोरिथम है, जो क्लस्टर सेण्टर या क्लस्टर सेंट्रोइड को सेलेक्ट करते समय, सेट में इसके डेटा पॉइंट्स में से एक को सेलेक्ट करेगा। | ||
[[File:Jmatrix.png|thumb| | [[File:Jmatrix.png|thumb|न्यूक्लेयर प्रोफाइल की जैकार्ड डिस्टेंस का हीट-मैप]]एल्गोरिथ्म इन चरणों का पालन करता है: | ||
# यादृच्छिक रूप से k | # यादृच्छिक रूप से k डिस्टिंक्ट डेटा पॉइंट्स को सेलेक्ट करें। ये इनिशियल क्लस्टर हैं। | ||
# प्रत्येक | # प्रत्येक पॉइंट और प्रत्येक 'k' क्लस्टर के मध्य की दूरी को मापें। (यह प्रत्येक पॉइंट k से पॉइंट्स की दूरी है)। | ||
# प्रत्येक | # प्रत्येक पॉइंट को निकटतम क्लस्टर में असाइन करें। | ||
# प्रत्येक क्लस्टर (मेडॉइड) | # प्रत्येक क्लस्टर (मेडॉइड) के सेंटर को फाइंड करें। | ||
# तब तक | # तब तक रिपीट करें जब तक कि क्लस्टर नहीं चेंज नहीं होते हैं। | ||
# प्रत्येक क्लस्टर के भीतर | # प्रत्येक क्लस्टर के भीतर वेरिएशन ऐड करके क्लस्टरिंग की गुणवत्ता का आकलन करें। | ||
# k | # k की विभिन्न वैल्यूज के साथ प्रोसेस को रिपीट करें। | ||
# 'k' के लिए | # उस प्लॉट में "एल्बो" फाइंड करके 'k' के लिए बेस्ट वैल्यू पिक करें, जिसकी k वैल्यू में सबसे कम वेरिएशन है। | ||
जीव विज्ञान में इसका उदाहरण जीनोम | जीव विज्ञान में इसका उदाहरण जीनोम की 3डी मैपिंग में प्रयोग किया जाता है। क्रोमोसोम 13 के माउस के हिस्ट1 रीजन की इनफार्मेशन [[सभी के लिए जीन अभिव्यक्ति|जीन एक्सप्रेशन ओमनीबस]] से प्राप्त की जाती है।<ref>[https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE64881]</ref> इस इनफार्मेशन में डेटा सम्मिलित है जिस पर न्यूक्लेयर प्रोफाइल कुछ जीनोमिक रीजन में दिखाई देते हैं। इस इनफार्मेशन के साथ, सभी लोकी के मध्य सामान्यीकृत दूरी फाइंड करने के लिए [[जैकार्ड दूरी|जैकार्ड इंडेक्स]] का उपयोग किया जा सकता है। | ||
=== ग्राफ एनालिटिक्स === | === ग्राफ एनालिटिक्स === | ||
ग्राफ़ एनालिटिक्स, या [[नेटवर्क सिद्धांत]], ग्राफ़ का अध्ययन है जो विभिन्न | ग्राफ़ एनालिटिक्स, या [[नेटवर्क सिद्धांत|नेटवर्क थ्योरी]], ग्राफ़ का अध्ययन है जो विभिन्न ऑब्जेक्ट्स के मध्य कनेक्शन का प्रतिनिधित्व करता है। जीव विज्ञान में ग्राफ़ सभी प्रकार के नेटवर्क का प्रतिनिधित्व कर सकते हैं जैसे प्रोटीन-प्रोटीन इंटरैक्शन या प्रोटीन-प्रोटीन इंटरैक्शन नेटवर्क, रेगुलेटरी नेटवर्क, मेटाबोलिक और बायोकैमिकल नेटवर्क इत्यादि। इन नेटवर्कों का एनालिसिस करने के कई प्रकार हैं। जिसमें ग्राफ़ में सेंट्रलिटी को देखना सम्मिलित है। ग्राफ़ में सेंट्रलिटी फाइंड करना नोड रैंकिंग को उनकी लोकप्रियता या ग्राफ़ में सेंट्रलिटी प्रदान करता है। यह ज्ञात करने में उपयोगी हो सकता है कि कौन से नोड सबसे महत्वपूर्ण हैं। जीव विज्ञान में यह कई प्रकार से अधिक उपयोगी हो सकता है। उदाहरण के लिए, यदि हमारे निकट निश्चित समय अवधि में जीन की गतिविधि पर डेटा होना था, तो हम यह देखने के लिए डिग्री सेंट्रलिटी का उपयोग कर सकते हैं कि कौन से जीन पूर्ण नेटवर्क में सबसे अधिक सक्रिय हैं, या कौन से जीन पूर्ण नेटवर्क में दूसरों के साथ सबसे अधिक इंटरैक्ट करते हैं। इससे हमें यह समझने में सहायता मिल सकती है कि कुछ जीन नेटवर्क में क्या भूमिका निभाते हैं। | ||
ग्राफ़ में | ग्राफ़ में सेंट्रलिटी की गणना करने की कई विधियां हैं, जिनमें से सभी सेंट्रलिटी पर विभिन्न प्रकार की इनफार्मेशन दे सकती हैं। जीव विज्ञान में सेंट्रलिटी को फाइंड करना कई भिन्न-भिन्न परिस्थितियों में प्रारम्भ किया जा सकता है, जिनमें से कुछ जीन रेगुलेटरी, प्रोटीन इंटरैक्शन और मेटाबोलिक नेटवर्क हैं।<ref name=":3">{{Cite journal |last1=Koschützki |first1=Dirk |last2=Schreiber |first2=Falk |date=2008-05-15 |title=Centrality Analysis Methods for Biological Networks and Their Application to Gene Regulatory Networks |journal=Gene Regulation and Systems Biology |volume=2 |pages=193–201 |doi=10.4137/grsb.s702 |issn=1177-6250 |pmc=2733090 |pmid=19787083}}</ref> | ||
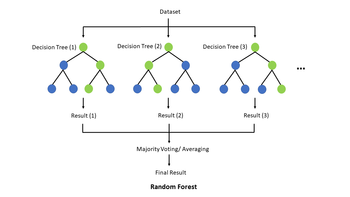
''' | '''सुपरवाइसड लर्निंग''' | ||
[[पर्यवेक्षित अध्ययन]] | [[पर्यवेक्षित अध्ययन|सुपरवाइसड लर्निंग]] एल्गोरिदम है जो लेबेल्ड डेटा से लर्न करता है और यह लर्न करता है कि अनलेबेल्ड फ्यूचर डेटा को लेबल कैसे असाइन किया जाए। जीव विज्ञान में सुपरवाइसड लर्निंग तब सहायक हो सकती है जब हमारे निकट डेटा हो जिसे हम कैटेगोराइज़ करना जानते हों और हम अधिक डेटा को उन कैटेगोरिज़ में कैटेगोराइज़ करना चाहेंगे।[[File:Random forest explain.png|thumb|350x350px|आरेख सिंपल रैंडम फारेस्ट प्रदर्शित कर रहा है।]]सामान्य सुपरवाइसड लर्निंग एल्गोरिथ्म रैंडम फारेस्ट है, जो डेटासेट को क्लास्सिफ़ाय करने के लिए मॉडल को ट्रेन करने के लिए कई डिसीज़न ट्रीज का उपयोग करता है। रैंडम फारेस्ट के आधार पर, डिसीज़न ट्री स्ट्रक्चर है जिसका उद्देश्य उस डेटा की कुछ ज्ञात विशेषताओं का उपयोग करके डेटा के कुछ सेट को क्लास्सिफ़ाय करना या लेबल करना है। इसका व्यावहारिक जैविक उदाहरण व्यक्ति के जेनेटिक डेटा को लेना और यह भविष्यवाणी करना होगा कि क्या वह व्यक्ति निश्चित रोग या कैंसर विकसित करने के लिए पूर्वनिर्धारित है या नहीं है। प्रत्येक इंटरनल नोड पर एल्गोरिद्म फीचर के लिए डेटासेट को चेक करता है, पूर्व उदाहरण में स्पेसिफिक जीन, और फिर परिणाम के आधार पर लेफ्ट अथवा राइट ब्रांचेज सम्मिलित हैं। फिर प्रत्येक लीफ नोड पर, डिसीजन ट्री डेटासेट को क्लास लेबल प्रदान करता है। इसलिए व्यवहार में, एल्गोरिथ्म डिसीजन ट्री के माध्यम से इनपुट डेटासेट के आधार पर स्पेसिफिक रूट-टू-लीफ पाथ पर चलता है, जिसके परिणामस्वरूप उस डेटासेट का क्लासिफिकेशन होता है। सामान्यतः, डिसीजन ट्री में टार्गेट वेरिएबल्स होते हैं जो डिस्क्रीट वैल्यूज लेते हैं, जैसे हां/नहीं, जिस स्थिति में इसे [[वर्गीकरण चार्ट|क्लासिफिकेशन ट्री]] के रूप में संदर्भित किया जाता है, किन्तु यदि टारगेट वेरिएबल कंटीन्यूअस है तो इसे [[प्रतिगमन वृक्ष|रिग्रेशन ट्री]] कहा जाता है। डिसीजन ट्री का निर्माण करने के लिए, इसे प्रथम ट्रेनिंग सेट का उपयोग करके ट्रेन किया जाना चाहिए जिससे यह आइडेंटीफाई किया सके कि कौन से फीचर टारगेट वेरिएबल के बेस्ट प्रेडिक्टर्स हैं। | ||
=== [[खुला स्रोत सॉफ्टवेयर]] === | === [[खुला स्रोत सॉफ्टवेयर|ओपन सोर्स सॉफ्टवेयर]] === | ||
ओपन सोर्स सॉफ्टवेयर कम्प्यूटेशनल बायोलॉजी के लिए | ओपन सोर्स सॉफ्टवेयर कम्प्यूटेशनल बायोलॉजी के लिए प्लेटफार्म प्रदान करता है जहां कोई भी रीसर्च में डेवेलप्ड सॉफ्टवेयर एक्सेस कर सकता है और उससे लाभ प्राप्त कर सकता है। पीएलओएस ओपन सोर्स सॉफ़्टवेयर के उपयोग के चार मुख्य कारण बताता है: | ||
* | *रेप्रोडूसिबिलिटी: यह शोधकर्ताओं को बायोलॉजिकल डेटा के मध्य संबंधों की गणना करने के लिए उपयोग की जाने वाली त्रुटिहीन विधियों का उपयोग करने की अनुमति देता है। | ||
* | *फास्टर डेवलपमेंट: डेवलपर्स और शोधकर्ताओं को सामान्य कार्यों के लिए एक्सिस्टिंग कोड को पुनः बनाने की आवश्यकता नहीं है। इसके अतिरिक्त वे बड़े प्रोजेक्ट्स के डेवलपमेंट और इम्प्लीमेंटेशन पर टाइम सेव करने के लिए प्री-एक्सिस्टिंग प्रोग्राम्स का उपयोग कर सकते हैं। | ||
* | * इंक्रीज़्ड क्वालिटी: समान विषय का अध्ययन करने वाले कई शोधकर्ताओं के इनपुट प्राप्त होने से अशौरेन्स की लेयर प्राप्त होती है कि कोड में एरर नहीं होंगे। | ||
* | *लॉन्ग-टर्म अवेलेबिलिटी: ओपन सोर्स प्रोग्राम किसी व्यवसाय या पेटेंट से बंधे नहीं हैं। यह उन्हें कई [[वेब पृष्ठ|वेब पेजेज]] पर पोस्ट करने की अनुमति देता है और यह सुनिश्चित करता है कि वे भविष्य में अवेलेबल हों।<ref>{{cite journal|journal=PLOS Computational Biology| doi=10.1371/journal.pcbi.1002799 | volume=8| issue=11 |title=The PLOS Computational Biology Software Section|pages=e1002799|year=2012|last1=Prlić|first1=Andreas| last2=Lapp | first2=Hilmar |pmc=3510099| bibcode=2012PLSCB...8E2799P }}</ref> | ||
== | == रिसर्च == | ||
कम्प्यूटेशनल बायोलॉजी से संबंधित कई बड़े | कम्प्यूटेशनल बायोलॉजी से संबंधित कई बड़े कॉन्फरेन्सेस हैं। कुछ उल्लेखनीय उदाहरण, इंटेलिजेंट सिस्टम फॉर मॉलिक्यूलर बायोलॉजी, यूरोपियन कांफ्रेंस ऑन कम्प्यूटेशनल बायोलॉजी और रिसर्च इन कम्प्यूटेशनल मॉलिक्यूलर बायोलॉजी हैं। | ||
कम्प्यूटेशनल बायोलॉजी के लिए समर्पित कई पत्रिकाएँ भी हैं। कुछ उल्लेखनीय उदाहरणों में | कम्प्यूटेशनल बायोलॉजी के लिए समर्पित कई पत्रिकाएँ भी हैं। कुछ उल्लेखनीय उदाहरणों में कम्प्यूटेशनल बायोलॉजी के जर्नल और पीएलओएस कम्प्यूटेशनल बायोलॉजी, पीर-रिव्यू ओपन एक्सेस जर्नल जिसमें कम्प्यूटेशनल बायोलॉजी के क्षेत्र में कई नोटेबल रिसर्च प्रोजेक्ट्स सम्मिलित हैं। वे [[सॉफ़्टवेयर]] पर रिव्यू प्रदान करते हैं, ओपन सोर्स सॉफ्टवेयर के लिए ट्यूटोरियल और अपकमिंग कम्प्यूटेशनल बायोलॉजी कॉन्फरेन्सेस पर इनफार्मेशन डिस्प्ले करते हैं। | ||
== संबंधित क्षेत्र == | == संबंधित क्षेत्र == | ||
कम्प्यूटेशनल बायोलॉजी, बायोइन्फार्मेटिक्स और [[गणितीय और सैद्धांतिक जीव विज्ञान]] [[जीवन विज्ञान की सूची]] के लिए सभी | कम्प्यूटेशनल बायोलॉजी, बायोइन्फार्मेटिक्स और [[गणितीय और सैद्धांतिक जीव विज्ञान|गणितीय तथा सैद्धांतिक जीव विज्ञान,]] [[जीवन विज्ञान की सूची|जीवन विज्ञान]] के लिए सभी इंटरडिसिप्लिनरी एप्प्रोच हैं जो गणित और इनफार्मेशन साइंस जैसे मात्रात्मक विषयों से आते हैं। [[नेशनल इंस्टीट्यूट ऑफ हेल्थ]] कम्प्यूटेशनल/मैथमेटिकल बायोलॉजी को जीव विज्ञान में सैद्धांतिक और प्रयोगात्मक प्रश्नों को संबोधित करने के लिए कम्प्यूटेशनल/मैथमेटिकल एप्प्रोच के उपयोग के रूप में वर्णित करता है, और इसके विपरीत, बायोइन्फार्मेटिक्स काम्प्लेक्स लाइफ-साइंस डेटा को समझने के लिए इनफार्मेशन साइंस के एप्लीकेशन के रूप में है।<ref name="nih"/> | ||
विशेष रूप से, एनआईएच परिभाषित करता है | विशेष रूप से, एनआईएच परिभाषित करता है | ||
{{blockquote| | {{blockquote|कम्प्यूटेशनल बायोलॉजी: बायोलॉजिकल, बिहेवियरल, और सोशल सिस्टम्स के अध्ययन के लिए डेटा-एनालिटिकल और सैद्धांतिक विधियों, गणितीय मॉडलिंग और कम्प्यूटेशनल सिमुलेशन टेक्निक्स का डेवलपमेंट और एप्लीकेशन है।<ref name="nih"/>}} | ||
{{blockquote| | {{blockquote|बायोइन्फार्मेटिक्स: बायोलॉजिकल, मेडिकल, बिहेवियरल और हेल्थ डेटा के उपयोग में वृद्धि के लिए कम्प्यूटेशनल टूल और एप्प्रोच की रिसर्च, डेवलपमेंट, या एप्लीकेशन, जिसमें ऐसे डेटा को एक्वायर करना, स्टोर करना, ऑर्गनाइज़ करना, एनालाइज करना या विसुअलाइज करना सम्मिलित है।.<ref name="nih"/>}} | ||
जबकि प्रत्येक क्षेत्र भिन्न है, उनके इंटरफ़ेस पर महत्वपूर्ण ओवरलैप हो सकता है,<ref name="nih"/>इतना अधिक कि कई लोगों के लिए, बायोइन्फार्मेटिक्स और कम्प्यूटेशनल बायोलॉजी ऐसे शब्द हैं जिनका परस्पर विनिमय किया जाता है। | जबकि प्रत्येक क्षेत्र भिन्न है, उनके इंटरफ़ेस पर महत्वपूर्ण ओवरलैप हो सकता है,<ref name="nih"/> इतना अधिक कि कई लोगों के लिए, बायोइन्फार्मेटिक्स और कम्प्यूटेशनल बायोलॉजी ऐसे शब्द हैं जिनका परस्पर विनिमय किया जाता है। | ||
कम्प्यूटेशनल बायोलॉजी और [[विकासवादी संगणना]] | कम्प्यूटेशनल बायोलॉजी और [[विकासवादी संगणना|एवोलुशनरी कम्प्यूटेशन]] शब्दों का समान नाम है, किन्तु उन्हें भ्रमित नहीं होना चाहिए। कम्प्यूटेशनल बायोलॉजी के विपरीत, एवोलुशनरी कम्प्यूटेशन बायोलॉजिकल डेटा के मॉडलिंग और एनालिसिस से संबंधित नहीं है। इसके अतिरिक्त यह विभिन्न प्रजातियों में विकास के विचारों के आधार पर एल्गोरिदम बनाता है। कभी-कभी [[जेनेटिक एल्गोरिद्म]] के रूप में संदर्भित, इस क्षेत्र का शोध कम्प्यूटेशनल बायोलॉजी पर प्रयुक्त किया जा सकता है। जबकि एवोलुशनरी कम्प्यूटेशन स्वाभाविक रूप से कम्प्यूटेशनल बायोलॉजी का अंश नहीं है, कम्प्यूटेशनल एवोलुशनरी बायोलॉजी इसका उपक्षेत्र है।<ref>{{cite journal |last=Foster |first=James |date=June 2001 |title=Evolutionary Computation |journal=Nature Reviews Genetics |volume=2 |issue=6 |pages=428–436 |doi=10.1038/35076523 |pmid=11389459 |s2cid=205017006}}</ref> | ||
== यह भी देखें == | == यह भी देखें == | ||
{{columns-list|colwidth=30em|* | {{columns-list|colwidth=30em|* आर्टिफिशियल लाइफ | ||
* | * बायोइन्फार्मेटिक्स | ||
* | * बायोलॉजिकल कंप्यूटिंग | ||
* | * बायोलॉजिकल सिमुलेशन | ||
* | * बायोसिमुलेशन | ||
* | * बायोस्टेटिस्टिक्स | ||
* | * कम्प्यूटेशनल ऑडियोलॉजी | ||
* | * कम्प्यूटेशनल केमिस्ट्री | ||
* | * कम्प्यूटेशनल साइंस | ||
* | * कम्प्यूटेशनल हिस्ट्री | ||
* | * डीएनए सिक्वेंसिंग | ||
* | * फंक्शनल जीनोमिक्स | ||
* | * इंटरनेशनल सोसाइटी फॉर कम्प्यूटेशनल बायोलॉजी | ||
* | * बायोइन्फार्मेटिक्स संस्थानों की लिस्ट | ||
* | * बायोलॉजिकल डेटाबेसेस की लिस्ट | ||
* | * मैथमेटिकल बायोलॉजी | ||
* | * मोंटे कार्लो मेथड | ||
* | * मॉलिक्यूलर मॉडलिंग | ||
* | * नेटवर्क बायोलॉजी | ||
* | * फाइलोंजेनेटिक्स | ||
* | * प्रोटिओमिक्स | ||
* | * स्ट्रक्चरल जीनोमिक्स | ||
* | * सिंथेटिक बायोलॉजी | ||
* | * सिस्टम्स बायोलॉजी | ||
}} | }} | ||
[[Category:Articles with hatnote templates targeting a nonexistent page|Computational Biology]] | |||
[[Category:Created On 18/02/2023|Computational Biology]] | |||
[[Category:Machine Translated Page|Computational Biology]] | |||
[[Category:Pages with reference errors|Computational Biology]] | |||
[[Category:Pages with script errors|Computational Biology]] | |||
[[Category:Short description with empty Wikidata description|Computational Biology]] | |||
[[Category:Template documentation pages|Short description/doc]] | |||
[[Category:Templates Vigyan Ready|Computational Biology]] | |||
[[Category:Templates that add a tracking category|Computational Biology]] | |||
[[Category:Templates that generate short descriptions|Computational Biology]] | |||
== संदर्भ == | == संदर्भ == | ||
| Line 182: | Line 180: | ||
[[Category: Machine Translated Page]] | [[Category: Machine Translated Page]] | ||
[[Category:Created On 18/02/2023]] | [[Category:Created On 18/02/2023]] | ||
[[Category:Vigyan Ready]] | |||
Latest revision as of 21:49, 10 October 2023
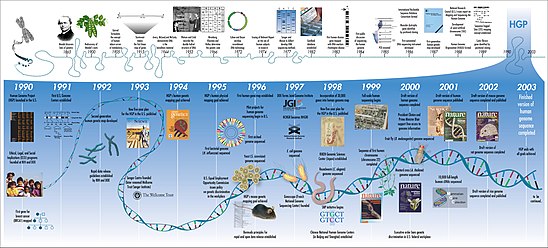
अभिकलनात्मक जीवविज्ञान (कम्प्यूटेशनल बायोलॉजी) बायोलॉजिकल सिस्टम्स और रिलेशनशिप का अध्ययन करने के लिए डेटा एनालिसिस, मैथमेटिकल मॉडलिंग और कंप्यूटर सिमुलेशन के उपयोग को संदर्भित करती है।[1] कंप्यूटर साइंस, जीव विज्ञान, और बिग डेटा का इंटरसेक्शन, इस क्षेत्र में अनुप्रयुक्त गणित, रसायन विज्ञान और आनुवंशिकी में भी आधार है।[2] यह बायोलॉजिकल कंप्यूटिंग से भिन्न है, कंप्यूटर इंजीनियरिंग का उपक्षेत्र जो कंप्यूटर बनाने के लिए बायोइंजीनियरिंग का उपयोग करता है।
इतिहास
बायोइन्फार्मेटिक्स, बायोलॉजिकल सिस्टम्स में इन्फार्मेटिक्स प्रोसेसों का एनालिसिस, 1970 के दशक में प्रारम्भ हुआ था। इस समय, आर्टिफिशियल इंटेलिजेंस में अनुसंधान नए एल्गोरिदम जेनेरेट करने के लिए ह्यूमन ब्रेन के नेटवर्क मॉडल का उपयोग कर रहा था। बायोलॉजिकल डेटा के इस उपयोग ने जैविक शोधकर्ताओं को अपने क्षेत्र में लार्ज डेटा सेटों को इवैल्युएट और कम्पेयर करने के लिए कंप्यूटर का उपयोग करने के लिए प्रेरित किया था।[3] 1982 तक, शोधकर्ताओं ने पंच कार्ड के माध्यम से इनफार्मेशन शेयर की थी। 1980 के दशक के अंत तक डेटा के अमाउंट में तीव्रता से वृद्धि हुई, जिससे क्विकली इंटरप्रेटिंग रिलेवेंट इनफार्मेशन के लिए नई कम्प्यूटेशनल विधियों की आवश्यकता हुई।[3]
कम्प्यूटेशनल बायोलॉजी का संभवतः सबसे प्रसिद्ध उदाहरण, ह्यूमन जीनोम प्रोजेक्ट, आधिकारिक रूप से 1990 में प्रारम्भ हुआ था।[4] 2003 तक, प्रोजेक्ट ने अपने प्रारंभिक लक्ष्यों को पूर्ण करते हुए ह्यूमन जीनोम का लगभग 85% मानचित्रण कर लिया था।[5] यद्यपि, कार्य प्रारम्भ रहा, और 2021 के स्तर तक पूर्ण जीनोम तक पहुँच गया, जिसमें केवल 0.3% शेष आधार संभावित अभिप्रायों से आच्छादित थे।[6][7] मिसिंग Y क्रोमोजोम जनवरी 2022 में ऐड किया गया था।
1990 के दशक के उत्तरार्ध से, कम्प्यूटेशनल बायोलॉजी, जीव विज्ञान का महत्वपूर्ण अंश बन गया है, जिससे कई उपक्षेत्र बन गए हैं।[8] वर्तमान में, इंटरनेशनल सोसाइटी फॉर कम्प्यूटेशनल बायोलॉजी 21 भिन्न-भिन्न 'कम्युनिटीज ऑफ़ स्पेशल इंटरेस्ट' को मान्यता देती है, और प्रत्येक बड़े क्षेत्र के खंड का प्रतिनिधित्व करती है।[9] ह्यूमन जीनोम के सीक्वेंस में सहायता करने के अतिरिक्त, कम्प्यूटेशनल बायोलॉजी ने ह्यूमन ब्रेन, जीनोम आर्किटेक्चर मैपिंग और मॉडल बायोलॉजिकल सिस्टम्स के एक्यूरेट मॉडल बनाने में सहायता की है।[3]
अनुप्रयोग
एनाटॉमी
कम्प्यूटेशनल एनाटॉमी मॉर्फोलॉजी (जीव विज्ञान) के विज़िबल अथवा ग्रॉस एनाटोमिकल स्केल पर शारीरिक संरचना और रूप का अध्ययन है। इसमें जैविक संरचनाओं के मॉडलिंग और अनुकरण के लिए कम्प्यूटेशनल मैथमेटिकल और डेटा-एनालिटिकल विधियों का विकास सम्मिलित है। यह मेडिकल इमेजिंग डिवाइसों के अतिरिक्त इमेज की जा रही शारीरिक संरचनाओं पर ध्यान केंद्रित करता है। मैग्नेटिक रेजोनेंस इमेजिंग जैसी टेक्नोलॉजीज के माध्यम से डेन्स 3डी मेज़रमेंट की उपलब्धता के कारण, कम्प्यूटेशनल एनाटॉमी 3डी में मोर्फेम स्केल पर एनाटोमिकल कोऑर्डिनेट सिस्टम निकालने के लिए मेडिकल इमेजिंग और बायोइंजीनियरिंग के उपक्षेत्र के रूप में प्रकट हुआ है।
कम्प्यूटेशनल एनाटॉमी का ओरिजिनल फार्मूलेशन रूपांतरणों के माध्यम से कार्य करने वाले उदाहरणों से आकार और रूप के जनरेटिव मॉडल के रूप में है।[10] डिफोमोर्फिज्म समूह का उपयोग में संरचनात्मक विन्यास से दूसरे में प्रवाह के लैग्रेंजियन और यूलेरियन वेगों के माध्यम से उत्पन्न समन्वय परिवर्तनों के माध्यम से विभिन्न समन्वय प्रणालियों का अध्ययन करने के लिए किया जाता है। यह आकृति सांख्यिकी और मॉर्फोमेट्रिक्स से संबंधित है, इस अंतर के साथ कि भिन्न-भिन्न समन्वय प्रणालियों को मैप करने के लिए इसका उपयोग किया जाता है, जिनके अध्ययन को डिफियोमोर्फोमेट्री के रूप में जाना जाता है।
डेटा और मॉडलिंग
गणितीय जीव विज्ञान जैविक प्रणालियों में संरचना, विकास और व्यवहार को नियंत्रित करने वाली प्रणालियों का परीक्षण करने के लिए जीवित जीवों के गणितीय मॉडल का उपयोग है। इसमें प्रायोगिक जीव विज्ञान के अधिक अनुभवजन्य दृष्टिकोण के अतिरिक्त समस्याओं के प्रति अधिक सैद्धांतिक दृष्टिकोण सम्मिलित है।[11] गणितीय जीव विज्ञान असतत गणित, टोपोलॉजी (कम्प्यूटेशनल मॉडलिंग के लिए भी उपयोगी), बायेसियन सांख्यिकी, रैखिक बीजगणित और बूलियन बीजगणित पर आधारित है।[12]
इन गणितीय दृष्टिकोणों ने बायोलॉजिकल डेटा को स्टोर करने, रिट्रीव करने और एनालाइज़ करने के लिए डेटाबेस और अन्य विधियों के निर्माण को सक्षम किया है, जिसे बायोइन्फार्मेटिक्स के रूप में जाना जाता है। सामान्यतः, इस प्रोसेस में जेनेटिक्स और जीन्स का एनालाइज़ेशन सम्मिलित होता है।
लार्ज डेटासेट को गेदर करने और उसका एनालाइज़ेशन करने से डेटा माइनिंग और कम्प्यूटेशनल बायोमॉडलिंग जैसे बढ़ते शोध क्षेत्रों के लिए स्थान बन गया है,[12] जो कंप्यूटर मॉडल और बायोलॉजिकल सिस्टम्स के विसुअल सिमुलेशन के निर्माण को संदर्भित करता है। यह शोधकर्ताओं को यह अनुमान लगाने की अनुमति देता है कि ऐसे सिस्टम्स डिफरेंट एनवायरनमेन्ट्स पर किस प्रकार रियेक्ट करते हैं, जो यह निर्धारित करने के लिए उपयोगी है कि क्या कोई सिस्टम एक्सटर्नल और इंटरनल पर्टरबेशन्स के विरुद्ध अपने स्टेट और फंक्शन्स को मेन्टेन रख सकता है।[13] कम्प्यूटेशनल बायोलॉजी ने प्रथम सिद्धांतों से एवोल्यूशनरी सिस्टम्स के पापुलेशन जेनेटिक्स मॉडल का निर्माण करके एवोल्यूशनरी बायोलॉजी की सहायता की है जिससे यह अनुमान लगाया जा सके कि क्या विकसित होने की संभावना है।
जीनोमिक्स
कम्प्यूटेशनल जीनोमिक्स कोशिका (जीव विज्ञान) और जीवों के जीनोम का अध्ययन है। ह्यूमन जीनोम प्रोजेक्ट कम्प्यूटेशनल जीनोमिक्स का उदाहरण है। यह प्रोजेक्ट सम्पूर्ण ह्यूमन जीनोम को डेटा के एक सेट में सीक्वेंस करना चाहता है। पूर्ण रूप से प्रस्तावित होने पर, यह डॉक्टरों को व्यक्तिगत रोगी के जीनोम को एनालाइज़ करने की अनुमति दे सकता है।[14] यह व्यक्तिगत चिकित्सा की संभावना को उन्मुक्त करता है, किसी व्यक्ति के प्री-एक्सिस्टिंग जेनेटिक पैटर्न के आधार पर उपचार निर्धारित करता है। शोधकर्ता जानवरों, पौधों, जीवाणुओं और अन्य सभी प्रकार के जीवन के जीनोम को सीक्वेंस करने का प्रयास कर रहे हैं।
जिन मुख्य प्रकारों में जीनोम की उपमा की जाती है उनमें से एक सीक्वेंस होमोलॉजी है। होमोलॉजी विभिन्न जीवों में जैविक संरचनाओं और न्यूक्लियोटाइड सीक्वेंसेस का अध्ययन है जो सामान्य पूर्वज से आते हैं। शोध से ज्ञात होता है कि न्यूली सीक्वेन्सड प्रोकैरियोट जीनोम में 80 से 90% जीनों की पहचान इस प्रकार से की जा सकती है।[15]
सीक्वेंस एलाइनमेंट बायोलॉजिकल सीक्वेंसेस या जीनों के मध्य समानता की अपेक्षा और ज्ञात करने की प्रक्रिया है। सीक्वेंस एलाइनमेंट कई बायोइन्फार्मेटिक्स अनुप्रयोगों में उपयोगी है, जैसे कि दो जीनों की सबसे लंबी सामान्य अनुगामी समस्या की गणना करना या कुछ रोगों के वेरिएंट की उपमा करना।
कम्प्यूटेशनल जीनोमिक्स में अनटच प्रोजेक्ट इंटरजेनिक रिजनों का एनालिसिस है, जिसमें ह्यूमन जीनोम का लगभग 97% भाग सम्मिलित है। शोधकर्ता कम्प्यूटेशनल और सांख्यिकीय विधियों के विकास और एनकोड तथा रोडमैप एपिजेनोमिक्स प्रोजेक्ट जैसे बड़े संघ प्रोजेक्ट्स के माध्यम से ह्यूमन जीनोम के नॉन-कोडिंग रिजनों के फंक्शन्स को समझने के लिए कार्य कर रहे हैं।
अणु, कोशिका (जीव विज्ञान), और जीव स्तर पर किसी जीव के जीव विज्ञान में व्यक्तिगत जीन किस प्रकार योगदान करते हैं, इसका अध्ययन करना जीन ओन्टोलॉजी के रूप में जाना जाता है। जीन ओन्टोलॉजी कंसोर्टियम का मिशन मॉलिक्यूलर लेवल से लेकर लार्जर पाथवेज़, सेलुलर और ऑर्गैनिस्म-लेवल सिस्टम्स तक बायोलॉजिकल सिस्टम्स का अप-टू-डेट, कम्प्रेहैन्सिव, कम्प्यूटेशनल मॉडल विकसित करना है। जीन ओन्टोलॉजी रिसोर्स मनुष्यों से लेकर जीवाणुओं तक, कई भिन्न-भिन्न जीवों से जीन के कार्यों (या जीन द्वारा उत्पादित प्रोटीन और नॉन-कोडिंग आरएनए अणुओं) के संबंध में करंट साइंटिफिक नॉलेज का कम्प्यूटेशनल रिप्रजेंटेशन प्रदान करता है।
3डी जीनोमिक्स कम्प्यूटेशनल बायोलॉजी में उपधारा है जो यूकेरियोटिक सेल के भीतर जीन के आर्गेनाईजेशन और इंटरेक्शन पर केंद्रित है। 3डी जीनोमिक डेटा गेदर करने के लिए उपयोग की जाने वाली विधि जीनोम आर्किटेक्चर मैपिंग (जीएएम) के माध्यम से है। जीएएम क्रायोसेक्शनिंग, डीएनए का परीक्षण करने के लिए नाभिक से स्ट्रिप प्राप्त करने की प्रक्रिया, लेजर माइक्रोडिसेक्शन के साथ संयोजन करके जीनोम में क्रोमेटिन और डीएनए की 3डी दूरी को मापता है। न्यूक्लेयर प्रोफ़ाइल केवल यह स्ट्रिप अथवा स्लाइस है जो नाभिक से ली गई है। प्रत्येक न्यूक्लेयर प्रोफ़ाइल में जीनोमिक विंडो होती हैं, जो डीएनए की आधार इकाई न्यूक्लियोटाइड के कुछ सीक्वेंस होते हैं। जीएएम पूर्ण सेल में काम्प्लेक्स, मल्टी एनहांसर क्रोमैटिन कॉन्टेक्ट्स के जीनोम नेटवर्क को कैप्चर करता है।
न्यूरोसाइंस
कम्प्यूटेशनल न्यूरोसाइंस नर्वस सिस्टम के इनफार्मेशन प्रोसेसिंग गुणों के संदर्भ में ब्रेन के कार्य का अध्ययन है। न्यूरोसाइंस का सबसेट, यह न्यूरोलॉजिकल सिस्टम के विशिष्ट दृश्यों का परीक्षण करने के लिए ब्रेन को मॉडल करता है।[16] ब्रेन के मॉडल में सम्मिलित हैं:
- रीयलिस्टिक ब्रेन मॉडल: ये मॉडल ब्रेन के प्रत्येक एस्पेक्ट का प्रतिनिधित्व करते हैं, जिसमें कोशीय स्तर पर जितना संभव हो उतना विवरण सम्मिलित होता है। रीयलिस्टिक मॉडल ब्रेन के संबंध में सबसे अधिक इनफार्मेशन प्रदान करते हैं, किन्तु एरर के लिए सबसे बड़ा मार्जिन भी होता है। ब्रेन मॉडल में अधिक वेरिएबल्स अधिक एरर होने की संभावना उत्पन्न करते हैं। ये मॉडल कोशीय संरचना के उन भागों के संबंध में नहीं बताते हैं जिनके संबंध में वैज्ञानिक नहीं जानते हैं। रीयलिस्टिक ब्रेन मॉडल कम्प्यूटेशनल रूप से सबसे हैवी और इम्प्लीमेंट करने के लिए अधिक बहुमूल्य हैं।[17]
- सिम्प्लिफायिंग ब्रेन मॉडल: ये मॉडल न्यूरोलॉजिकल सिस्टम की स्पेसिफिक फिजिकल प्रॉपर्टी का आकलन करने के लिए मॉडल के स्कोप को सीमित करने पर विचार करते हैं। यह इंटेंसिव कम्प्यूटेशनल प्रोब्लेम्स को सॉल्व करने की अनुमति देता है, और रीयलिस्टिक ब्रेन मॉडल से पोटेंशियल एरर के अमाउंट को कम करता है।[17] वर्तमान में ऐसी गणनाओं की गति में वृद्धि के लिए उपयोग किए जाने वाले एल्गोरिदम और डेटा स्ट्रक्चर्स में संशोधन करना कम्प्यूटेशनल न्यूरोसाइंटिस्ट का कार्य है।
कम्प्यूटेशनल न्यूरोसाइकिएट्री इमर्जिंग फील्ड है जो मानसिक विकारों में सम्मिलित ब्रेन मैकेनिज्म की मैथमेटिकल और कंप्यूटर-असिस्टेड मॉडलिंग का उपयोग करता है। कई उपक्रमों ने प्रदर्शित किया है कि कम्प्यूटेशनल मॉडलिंग न्यूरोनल सर्किट के अध्ययन में महत्वपूर्ण योगदान है जो मानसिक कार्यों और शिथिलताओं को उत्पन्न कर सकता है।[18][19][20]
फार्माकोलॉजी
कम्प्यूटेशनल फार्माकोलॉजी विशिष्ट जीनोटाइप और रोगों और फिर स्क्रीनिंग ड्रग डाटा के मध्य संबंध प्राप्त के लिए जीनोमिक डेटा के प्रभावों का अध्ययन है।[21] फार्मास्युटिकल उद्योग को ड्रग डेटा का एनालिसिस करने की विधियों में परिवर्तन करने की आवश्यकता है। फार्माकोलॉजिस्ट ड्रग की प्रभावशीलता से संबंधित रासायनिक और जीनोमिक डेटा की उपमा करने के लिए माइक्रोसॉफ्ट एक्सेल का उपयोग करने में सक्षम थे। यद्यपि, उद्योग उस स्थिति तक पहुँच गया है जिसे एक्सेल बैरिकेड कहा जाता है। यह स्प्रेडशीट पर एक्सेसिबल सेल्स की सीमित संख्या से उत्पन्न होता है। इस विकास ने कम्प्यूटेशनल फार्माकोलॉजी की आवश्यकता को उत्पन्न किया था। वैज्ञानिक और शोधकर्ता इन मैसिव डेटा सेट्स का एनालिसिस करने के लिए कम्प्यूटेशनल विधि विकसित करते हैं। यह नोटेबल डेटा पॉइंट्स के मध्य कुशल उपमा की अनुमति देता है और अधिक एक्यूरेट ड्रग्स को विकसित करने की अनुमति देता है।[22]
एनालिस्टों का अनुमान है कि यदि पेटेंट के कारण प्रमुख ड्रग्स विफल हो जाते हैं, तो मार्किट में उपस्थित ड्रगों को परिवर्तित करने के लिए कम्प्यूटेशनल बायोलॉजी की आवश्यकता होगी। कम्प्यूटेशनल बायोलॉजी में डॉक्टरेट छात्रों को डॉक्टरेट के पश्चात् के पदों को लेने के अतिरिक्त उद्योग में करियर बनाने के लिए प्रोत्साहित किया जा रहा है। यह नए ड्रग के उत्पादन के लिए आवश्यक बिग डेटा सेट के अधिक योग्य एनालिस्टों की आवश्यकता वाली प्रमुख ड्रग कंपनियों का प्रत्यक्ष परिणाम है।[22]
इसी प्रकार, कम्प्यूटेशनल ऑन्कोलॉजी का उद्देश्य एल्गोरिथम एप्रोच के माध्यम से कैंसर में भविष्य के उत्परिवर्तन को निर्धारित करना है। इस क्षेत्र में अनुसंधान ने उच्च-थ्रूपुट मापन का उपयोग करने के लिए प्रेरित किया है जो रोबोटिक्स और अन्य सेंसिंग डिवाइसों का उपयोग करते हुए लाखों डेटा पॉइंट्स हैं। यह डेटा डीएनए, आरएनए और अन्य जैविक संरचनाओं से एकत्र किया जाता है। फोकस के क्षेत्रों में ट्यूमर की विशेषताओं का निर्धारण करना, अणुओं का विश्लेषण करना जो कैंसर उत्पन्न करने में नियतात्मक हैं, और यह समझना कि ह्यूमन जीनोम ट्यूमर और कैंसर के कारण से कैसे संबंधित है।[23][24]
टेक्निक्स
कम्प्यूटेशनल जीवविज्ञानी अपने शोध करने के लिए सॉफ्टवेयर और एल्गोरिदम की विस्तृत श्रृंखला का उपयोग करते हैं।
अनसुपरवाइसड लर्निंग
अनसुपरवाइसड लर्निंग एल्गोरिदम है जो अनलेबेल्ड डेटा में पैटर्न फाइंड करता है। उदाहरण k-मीन्स क्लस्टरिंग है, जिसका उद्देश्य n डेटा पॉइंट्स को k समूहों में विभाजित करना है, जिसमें प्रत्येक डेटा पॉइंट निकटतम माध्य वाले क्लस्टर से संबंधित है। अन्य वर्जन k-मेडोइड्स एल्गोरिथम है, जो क्लस्टर सेण्टर या क्लस्टर सेंट्रोइड को सेलेक्ट करते समय, सेट में इसके डेटा पॉइंट्स में से एक को सेलेक्ट करेगा।
एल्गोरिथ्म इन चरणों का पालन करता है:
- यादृच्छिक रूप से k डिस्टिंक्ट डेटा पॉइंट्स को सेलेक्ट करें। ये इनिशियल क्लस्टर हैं।
- प्रत्येक पॉइंट और प्रत्येक 'k' क्लस्टर के मध्य की दूरी को मापें। (यह प्रत्येक पॉइंट k से पॉइंट्स की दूरी है)।
- प्रत्येक पॉइंट को निकटतम क्लस्टर में असाइन करें।
- प्रत्येक क्लस्टर (मेडॉइड) के सेंटर को फाइंड करें।
- तब तक रिपीट करें जब तक कि क्लस्टर नहीं चेंज नहीं होते हैं।
- प्रत्येक क्लस्टर के भीतर वेरिएशन ऐड करके क्लस्टरिंग की गुणवत्ता का आकलन करें।
- k की विभिन्न वैल्यूज के साथ प्रोसेस को रिपीट करें।
- उस प्लॉट में "एल्बो" फाइंड करके 'k' के लिए बेस्ट वैल्यू पिक करें, जिसकी k वैल्यू में सबसे कम वेरिएशन है।
जीव विज्ञान में इसका उदाहरण जीनोम की 3डी मैपिंग में प्रयोग किया जाता है। क्रोमोसोम 13 के माउस के हिस्ट1 रीजन की इनफार्मेशन जीन एक्सप्रेशन ओमनीबस से प्राप्त की जाती है।[25] इस इनफार्मेशन में डेटा सम्मिलित है जिस पर न्यूक्लेयर प्रोफाइल कुछ जीनोमिक रीजन में दिखाई देते हैं। इस इनफार्मेशन के साथ, सभी लोकी के मध्य सामान्यीकृत दूरी फाइंड करने के लिए जैकार्ड इंडेक्स का उपयोग किया जा सकता है।
ग्राफ एनालिटिक्स
ग्राफ़ एनालिटिक्स, या नेटवर्क थ्योरी, ग्राफ़ का अध्ययन है जो विभिन्न ऑब्जेक्ट्स के मध्य कनेक्शन का प्रतिनिधित्व करता है। जीव विज्ञान में ग्राफ़ सभी प्रकार के नेटवर्क का प्रतिनिधित्व कर सकते हैं जैसे प्रोटीन-प्रोटीन इंटरैक्शन या प्रोटीन-प्रोटीन इंटरैक्शन नेटवर्क, रेगुलेटरी नेटवर्क, मेटाबोलिक और बायोकैमिकल नेटवर्क इत्यादि। इन नेटवर्कों का एनालिसिस करने के कई प्रकार हैं। जिसमें ग्राफ़ में सेंट्रलिटी को देखना सम्मिलित है। ग्राफ़ में सेंट्रलिटी फाइंड करना नोड रैंकिंग को उनकी लोकप्रियता या ग्राफ़ में सेंट्रलिटी प्रदान करता है। यह ज्ञात करने में उपयोगी हो सकता है कि कौन से नोड सबसे महत्वपूर्ण हैं। जीव विज्ञान में यह कई प्रकार से अधिक उपयोगी हो सकता है। उदाहरण के लिए, यदि हमारे निकट निश्चित समय अवधि में जीन की गतिविधि पर डेटा होना था, तो हम यह देखने के लिए डिग्री सेंट्रलिटी का उपयोग कर सकते हैं कि कौन से जीन पूर्ण नेटवर्क में सबसे अधिक सक्रिय हैं, या कौन से जीन पूर्ण नेटवर्क में दूसरों के साथ सबसे अधिक इंटरैक्ट करते हैं। इससे हमें यह समझने में सहायता मिल सकती है कि कुछ जीन नेटवर्क में क्या भूमिका निभाते हैं।
ग्राफ़ में सेंट्रलिटी की गणना करने की कई विधियां हैं, जिनमें से सभी सेंट्रलिटी पर विभिन्न प्रकार की इनफार्मेशन दे सकती हैं। जीव विज्ञान में सेंट्रलिटी को फाइंड करना कई भिन्न-भिन्न परिस्थितियों में प्रारम्भ किया जा सकता है, जिनमें से कुछ जीन रेगुलेटरी, प्रोटीन इंटरैक्शन और मेटाबोलिक नेटवर्क हैं।[26]
सुपरवाइसड लर्निंग
सुपरवाइसड लर्निंग एल्गोरिदम है जो लेबेल्ड डेटा से लर्न करता है और यह लर्न करता है कि अनलेबेल्ड फ्यूचर डेटा को लेबल कैसे असाइन किया जाए। जीव विज्ञान में सुपरवाइसड लर्निंग तब सहायक हो सकती है जब हमारे निकट डेटा हो जिसे हम कैटेगोराइज़ करना जानते हों और हम अधिक डेटा को उन कैटेगोरिज़ में कैटेगोराइज़ करना चाहेंगे।
सामान्य सुपरवाइसड लर्निंग एल्गोरिथ्म रैंडम फारेस्ट है, जो डेटासेट को क्लास्सिफ़ाय करने के लिए मॉडल को ट्रेन करने के लिए कई डिसीज़न ट्रीज का उपयोग करता है। रैंडम फारेस्ट के आधार पर, डिसीज़न ट्री स्ट्रक्चर है जिसका उद्देश्य उस डेटा की कुछ ज्ञात विशेषताओं का उपयोग करके डेटा के कुछ सेट को क्लास्सिफ़ाय करना या लेबल करना है। इसका व्यावहारिक जैविक उदाहरण व्यक्ति के जेनेटिक डेटा को लेना और यह भविष्यवाणी करना होगा कि क्या वह व्यक्ति निश्चित रोग या कैंसर विकसित करने के लिए पूर्वनिर्धारित है या नहीं है। प्रत्येक इंटरनल नोड पर एल्गोरिद्म फीचर के लिए डेटासेट को चेक करता है, पूर्व उदाहरण में स्पेसिफिक जीन, और फिर परिणाम के आधार पर लेफ्ट अथवा राइट ब्रांचेज सम्मिलित हैं। फिर प्रत्येक लीफ नोड पर, डिसीजन ट्री डेटासेट को क्लास लेबल प्रदान करता है। इसलिए व्यवहार में, एल्गोरिथ्म डिसीजन ट्री के माध्यम से इनपुट डेटासेट के आधार पर स्पेसिफिक रूट-टू-लीफ पाथ पर चलता है, जिसके परिणामस्वरूप उस डेटासेट का क्लासिफिकेशन होता है। सामान्यतः, डिसीजन ट्री में टार्गेट वेरिएबल्स होते हैं जो डिस्क्रीट वैल्यूज लेते हैं, जैसे हां/नहीं, जिस स्थिति में इसे क्लासिफिकेशन ट्री के रूप में संदर्भित किया जाता है, किन्तु यदि टारगेट वेरिएबल कंटीन्यूअस है तो इसे रिग्रेशन ट्री कहा जाता है। डिसीजन ट्री का निर्माण करने के लिए, इसे प्रथम ट्रेनिंग सेट का उपयोग करके ट्रेन किया जाना चाहिए जिससे यह आइडेंटीफाई किया सके कि कौन से फीचर टारगेट वेरिएबल के बेस्ट प्रेडिक्टर्स हैं।
ओपन सोर्स सॉफ्टवेयर
ओपन सोर्स सॉफ्टवेयर कम्प्यूटेशनल बायोलॉजी के लिए प्लेटफार्म प्रदान करता है जहां कोई भी रीसर्च में डेवेलप्ड सॉफ्टवेयर एक्सेस कर सकता है और उससे लाभ प्राप्त कर सकता है। पीएलओएस ओपन सोर्स सॉफ़्टवेयर के उपयोग के चार मुख्य कारण बताता है:
- रेप्रोडूसिबिलिटी: यह शोधकर्ताओं को बायोलॉजिकल डेटा के मध्य संबंधों की गणना करने के लिए उपयोग की जाने वाली त्रुटिहीन विधियों का उपयोग करने की अनुमति देता है।
- फास्टर डेवलपमेंट: डेवलपर्स और शोधकर्ताओं को सामान्य कार्यों के लिए एक्सिस्टिंग कोड को पुनः बनाने की आवश्यकता नहीं है। इसके अतिरिक्त वे बड़े प्रोजेक्ट्स के डेवलपमेंट और इम्प्लीमेंटेशन पर टाइम सेव करने के लिए प्री-एक्सिस्टिंग प्रोग्राम्स का उपयोग कर सकते हैं।
- इंक्रीज़्ड क्वालिटी: समान विषय का अध्ययन करने वाले कई शोधकर्ताओं के इनपुट प्राप्त होने से अशौरेन्स की लेयर प्राप्त होती है कि कोड में एरर नहीं होंगे।
- लॉन्ग-टर्म अवेलेबिलिटी: ओपन सोर्स प्रोग्राम किसी व्यवसाय या पेटेंट से बंधे नहीं हैं। यह उन्हें कई वेब पेजेज पर पोस्ट करने की अनुमति देता है और यह सुनिश्चित करता है कि वे भविष्य में अवेलेबल हों।[27]
रिसर्च
कम्प्यूटेशनल बायोलॉजी से संबंधित कई बड़े कॉन्फरेन्सेस हैं। कुछ उल्लेखनीय उदाहरण, इंटेलिजेंट सिस्टम फॉर मॉलिक्यूलर बायोलॉजी, यूरोपियन कांफ्रेंस ऑन कम्प्यूटेशनल बायोलॉजी और रिसर्च इन कम्प्यूटेशनल मॉलिक्यूलर बायोलॉजी हैं।
कम्प्यूटेशनल बायोलॉजी के लिए समर्पित कई पत्रिकाएँ भी हैं। कुछ उल्लेखनीय उदाहरणों में कम्प्यूटेशनल बायोलॉजी के जर्नल और पीएलओएस कम्प्यूटेशनल बायोलॉजी, पीर-रिव्यू ओपन एक्सेस जर्नल जिसमें कम्प्यूटेशनल बायोलॉजी के क्षेत्र में कई नोटेबल रिसर्च प्रोजेक्ट्स सम्मिलित हैं। वे सॉफ़्टवेयर पर रिव्यू प्रदान करते हैं, ओपन सोर्स सॉफ्टवेयर के लिए ट्यूटोरियल और अपकमिंग कम्प्यूटेशनल बायोलॉजी कॉन्फरेन्सेस पर इनफार्मेशन डिस्प्ले करते हैं।
संबंधित क्षेत्र
कम्प्यूटेशनल बायोलॉजी, बायोइन्फार्मेटिक्स और गणितीय तथा सैद्धांतिक जीव विज्ञान, जीवन विज्ञान के लिए सभी इंटरडिसिप्लिनरी एप्प्रोच हैं जो गणित और इनफार्मेशन साइंस जैसे मात्रात्मक विषयों से आते हैं। नेशनल इंस्टीट्यूट ऑफ हेल्थ कम्प्यूटेशनल/मैथमेटिकल बायोलॉजी को जीव विज्ञान में सैद्धांतिक और प्रयोगात्मक प्रश्नों को संबोधित करने के लिए कम्प्यूटेशनल/मैथमेटिकल एप्प्रोच के उपयोग के रूप में वर्णित करता है, और इसके विपरीत, बायोइन्फार्मेटिक्स काम्प्लेक्स लाइफ-साइंस डेटा को समझने के लिए इनफार्मेशन साइंस के एप्लीकेशन के रूप में है।[1]
विशेष रूप से, एनआईएच परिभाषित करता है
कम्प्यूटेशनल बायोलॉजी: बायोलॉजिकल, बिहेवियरल, और सोशल सिस्टम्स के अध्ययन के लिए डेटा-एनालिटिकल और सैद्धांतिक विधियों, गणितीय मॉडलिंग और कम्प्यूटेशनल सिमुलेशन टेक्निक्स का डेवलपमेंट और एप्लीकेशन है।[1]
बायोइन्फार्मेटिक्स: बायोलॉजिकल, मेडिकल, बिहेवियरल और हेल्थ डेटा के उपयोग में वृद्धि के लिए कम्प्यूटेशनल टूल और एप्प्रोच की रिसर्च, डेवलपमेंट, या एप्लीकेशन, जिसमें ऐसे डेटा को एक्वायर करना, स्टोर करना, ऑर्गनाइज़ करना, एनालाइज करना या विसुअलाइज करना सम्मिलित है।.[1]
जबकि प्रत्येक क्षेत्र भिन्न है, उनके इंटरफ़ेस पर महत्वपूर्ण ओवरलैप हो सकता है,[1] इतना अधिक कि कई लोगों के लिए, बायोइन्फार्मेटिक्स और कम्प्यूटेशनल बायोलॉजी ऐसे शब्द हैं जिनका परस्पर विनिमय किया जाता है।
कम्प्यूटेशनल बायोलॉजी और एवोलुशनरी कम्प्यूटेशन शब्दों का समान नाम है, किन्तु उन्हें भ्रमित नहीं होना चाहिए। कम्प्यूटेशनल बायोलॉजी के विपरीत, एवोलुशनरी कम्प्यूटेशन बायोलॉजिकल डेटा के मॉडलिंग और एनालिसिस से संबंधित नहीं है। इसके अतिरिक्त यह विभिन्न प्रजातियों में विकास के विचारों के आधार पर एल्गोरिदम बनाता है। कभी-कभी जेनेटिक एल्गोरिद्म के रूप में संदर्भित, इस क्षेत्र का शोध कम्प्यूटेशनल बायोलॉजी पर प्रयुक्त किया जा सकता है। जबकि एवोलुशनरी कम्प्यूटेशन स्वाभाविक रूप से कम्प्यूटेशनल बायोलॉजी का अंश नहीं है, कम्प्यूटेशनल एवोलुशनरी बायोलॉजी इसका उपक्षेत्र है।[28]
यह भी देखें
- आर्टिफिशियल लाइफ
- बायोइन्फार्मेटिक्स
- बायोलॉजिकल कंप्यूटिंग
- बायोलॉजिकल सिमुलेशन
- बायोसिमुलेशन
- बायोस्टेटिस्टिक्स
- कम्प्यूटेशनल ऑडियोलॉजी
- कम्प्यूटेशनल केमिस्ट्री
- कम्प्यूटेशनल साइंस
- कम्प्यूटेशनल हिस्ट्री
- डीएनए सिक्वेंसिंग
- फंक्शनल जीनोमिक्स
- इंटरनेशनल सोसाइटी फॉर कम्प्यूटेशनल बायोलॉजी
- बायोइन्फार्मेटिक्स संस्थानों की लिस्ट
- बायोलॉजिकल डेटाबेसेस की लिस्ट
- मैथमेटिकल बायोलॉजी
- मोंटे कार्लो मेथड
- मॉलिक्यूलर मॉडलिंग
- नेटवर्क बायोलॉजी
- फाइलोंजेनेटिक्स
- प्रोटिओमिक्स
- स्ट्रक्चरल जीनोमिक्स
- सिंथेटिक बायोलॉजी
- सिस्टम्स बायोलॉजी
संदर्भ
- ↑ 1.0 1.1 1.2 1.3 1.4 "NIH working definition of bioinformatics and computational biology" (PDF). Biomedical Information Science and Technology Initiative. 17 July 2000. Archived from the original (PDF) on 5 September 2012. Retrieved 18 August 2012.
- ↑ "About the CCMB". Center for Computational Molecular Biology. Retrieved 18 August 2012.
- ↑ 3.0 3.1 3.2 Hogeweg, Paulien (7 March 2011). "The Roots of Bioinformatics in Theoretical Biology". PLOS Computational Biology. 3. 7 (3): e1002021. Bibcode:2011PLSCB...7E2021H. doi:10.1371/journal.pcbi.1002021. PMC 3068925. PMID 21483479.
- ↑ "The Human Genome Project". The Human Genome Project. 22 December 2020. Retrieved 13 April 2022.
- ↑ "Human Genome Project FAQ". Genome.gov (in English). Retrieved 2022-04-20.
- ↑ "T2T-CHM13v1.1 - Genome - Assembly - NCBI". www.ncbi.nlm.nih.gov. Retrieved 2022-04-20.
- ↑ "Genome List - Genome - NCBI". www.ncbi.nlm.nih.gov. Retrieved 2022-04-20.
- ↑ Bourne, Philip (2012). "Rise and Demise of Bioinformatics? Promise and Progress". PLOS Computational Biology. 8 (4): e1002487. Bibcode:2012PLSCB...8E2487O. doi:10.1371/journal.pcbi.1002487. PMC 3343106. PMID 22570600.
- ↑ "COSI Information". www.iscb.org. Retrieved 2022-04-21.
- ↑ Grenander, Ulf; Miller, Michael I. (1998-12-01). "Computational Anatomy: An Emerging Discipline". Q. Appl. Math. 56 (4): 617–694. doi:10.1090/qam/1668732.
- ↑ "Mathematical Biology | Faculty of Science". www.ualberta.ca. Retrieved 2022-04-18.
- ↑ 12.0 12.1 "The Sub-fields of Computational Biology". Ninh Laboratory of Computational Biology (in English). 2013-02-18. Retrieved 2022-04-18.
- ↑ Kitano, Hiroaki (14 November 2002). "Computational systems biology". Nature. 420 (6912): 206–10. Bibcode:2002Natur.420..206K. doi:10.1038/nature01254. PMID 12432404. S2CID 4401115. ProQuest 204483859.
- ↑ "Genome Sequencing to the Rest of Us". Scientific American.
- ↑ Koonin, Eugene (6 March 2001). "Computational Genomics". Curr. Biol. 11 (5): 155–158. doi:10.1016/S0960-9822(01)00081-1. PMID 11267880. S2CID 17202180.
- ↑ "Computational Neuroscience | Neuroscience". www.bu.edu.
- ↑ 17.0 17.1 Sejnowski, Terrence; Christof Koch; Patricia S. Churchland (9 September 1988). "Computational Neuroscience". Science. 4871. 241 (4871): 1299–306. Bibcode:1988Sci...241.1299S. doi:10.1126/science.3045969. PMID 3045969.
- ↑ Dauvermann, Maria R.; Whalley, Heather C.; Schmidt, Andrã©; Lee, Graham L.; Romaniuk, Liana; Roberts, Neil; Johnstone, Eve C.; Lawrie, Stephen M.; Moorhead, Thomas W. J. (2014). "Computational Neuropsychiatry – Schizophrenia as a Cognitive Brain Network Disorder". Frontiers in Psychiatry. 5: 30. doi:10.3389/fpsyt.2014.00030. PMC 3971172. PMID 24723894.
- ↑ Tretter, F.; Albus, M. (December 2007). "'Computational Neuropsychiatry' of Working Memory Disorders in Schizophrenia: The Network Connectivity in Prefrontal Cortex - Data and Models". Pharmacopsychiatry. 40 (S 1): S2–S16. doi:10.1055/S-2007-993139. S2CID 18574327.
- ↑ Marin-Sanguino, A.; Mendoza, E. (2008). "Hybrid Modeling in Computational Neuropsychiatry". Pharmacopsychiatry. 41: S85–S88. doi:10.1055/s-2008-1081464. PMID 18756425.
- ↑ Price, Michael (2012-04-13). "Computational Biologists: The Next Pharma Scientists?".
- ↑ 22.0 22.1 Jessen, Walter (2012-04-15). "Pharma's shifting strategy means more jobs for computational biologists".
- ↑ Barbolosi, Dominique; Ciccolini, Joseph; Lacarelle, Bruno; Barlesi, Fabrice; Andre, Nicolas (2016). "Computational oncology--mathematical modelling of drug regimens for precision medicine". Nature Reviews Clinical Oncology. 13 (4): 242–254. doi:10.1038/nrclinonc.2015.204. PMID 26598946. S2CID 22492353.
- ↑ Yakhini, Zohar (2011). "Cancer Computational Biology". BMC Bioinformatics. 12: 120. doi:10.1186/1471-2105-12-120. PMC 3111371. PMID 21521513.
- ↑ [1]
- ↑ Koschützki, Dirk; Schreiber, Falk (2008-05-15). "Centrality Analysis Methods for Biological Networks and Their Application to Gene Regulatory Networks". Gene Regulation and Systems Biology. 2: 193–201. doi:10.4137/grsb.s702. ISSN 1177-6250. PMC 2733090. PMID 19787083.
- ↑ Prlić, Andreas; Lapp, Hilmar (2012). "The PLOS Computational Biology Software Section". PLOS Computational Biology. 8 (11): e1002799. Bibcode:2012PLSCB...8E2799P. doi:10.1371/journal.pcbi.1002799. PMC 3510099.
- ↑ Foster, James (June 2001). "Evolutionary Computation". Nature Reviews Genetics. 2 (6): 428–436. doi:10.1038/35076523. PMID 11389459. S2CID 205017006.