इंटरप्रो: Difference between revisions
No edit summary |
No edit summary |
||
| Line 52: | Line 52: | ||
;सुपरफैमिली: सुपरफैमिली प्रोफाइल छिपे हुए मार्कोव प्ररूप की लाइब्रेरी है जो ज्ञात संरचना के सभी प्रोटीन का प्रतिनिधित्व करती है। लाइब्रेरी प्रोटीन के संरचनात्मक वर्गीकरण डेटाबेस प्रोटीन के वर्गीकरण पर आधारित है: प्रत्येक प्ररूप एससीओपी डोमेन के समान है एवं इसका उद्देश्य पूरे एससीओपी [[प्रोटीन सुपरफैमिली]] का प्रतिनिधित्व करना है जो डोमेन से संबंधित है। सुपरफ़ैमिली का उपयोग सभी पूर्णतः अनुक्रमित जीनोमों में संरचनात्मक कार्य करने के लिए किया गया है। | ;सुपरफैमिली: सुपरफैमिली प्रोफाइल छिपे हुए मार्कोव प्ररूप की लाइब्रेरी है जो ज्ञात संरचना के सभी प्रोटीन का प्रतिनिधित्व करती है। लाइब्रेरी प्रोटीन के संरचनात्मक वर्गीकरण डेटाबेस प्रोटीन के वर्गीकरण पर आधारित है: प्रत्येक प्ररूप एससीओपी डोमेन के समान है एवं इसका उद्देश्य पूरे एससीओपी [[प्रोटीन सुपरफैमिली]] का प्रतिनिधित्व करना है जो डोमेन से संबंधित है। सुपरफ़ैमिली का उपयोग सभी पूर्णतः अनुक्रमित जीनोमों में संरचनात्मक कार्य करने के लिए किया गया है। | ||
;एसएफएलडी: एंजाइमों का श्रेणीबद्ध वर्गीकरण जो विशिष्ट अनुक्रम-संरचना विशेषताओं को विशिष्ट रासायनिक क्षमताओं से जोड़ता है। | ;एसएफएलडी: एंजाइमों का श्रेणीबद्ध वर्गीकरण जो विशिष्ट अनुक्रम-संरचना विशेषताओं को विशिष्ट रासायनिक क्षमताओं से जोड़ता है। | ||
;[[TIGRFAMs]]: TIGRFAMs प्रोटीन सदस्यों का संग्रह है, जिसमें क्यूरेटेड मल्टीपल अनुक्रम संरेखण, छिपे हुए मार्कोव प्ररूप (HMM) एवं एनोटेशन सम्मिलित हैं, जो अनुक्रम होमोलॉजी के आधार पर कार्यात्मक रूप से संबंधित प्रोटीन की पहचान करने के लिए | ;[[TIGRFAMs]]: TIGRFAMs प्रोटीन सदस्यों का संग्रह है, जिसमें क्यूरेटेड मल्टीपल अनुक्रम संरेखण, छिपे हुए मार्कोव प्ररूप (HMM) एवं एनोटेशन सम्मिलित हैं, जो अनुक्रम होमोलॉजी के आधार पर कार्यात्मक रूप से संबंधित प्रोटीन की पहचान करने के लिए उपकरण प्रदान करता है। वे प्रविष्टियाँ जो समतुल्य समूह हैं, समजात प्रोटीन हैं जो कार्य के संबंध में संरक्षित हैं। | ||
=== डेटा प्रकार === | === डेटा प्रकार === | ||
| Line 93: | Line 93: | ||
==== इंटरप्रो प्रविष्टि प्रकार ==== | ==== इंटरप्रो प्रविष्टि प्रकार ==== | ||
इंटरप्रो प्रविष्टियों को | इंटरप्रो प्रविष्टियों को पाँच प्रकारों में विभाजित किया जा सकता है: | ||
* | * समजात सुपरफैमिली: प्रोटीन का समूह जो समान विकासवादी उत्पत्ति साझा करता है जैसा कि उनकी संरचनात्मक समानता में देखा जाता है, अपितु उनके अनुक्रम अत्यधिक समान न हों। ये प्रविष्टियाँ विशेष रूप से केवल दो सदस्य डेटाबेस : CATH-Gene3D एवं सुपरफ़ैमिली द्वारा प्रदान की जाती हैं। | ||
* सदस्य: प्रोटीन का समूह जिसकी सामान्य विकासवादी उत्पत्ति संरचनात्मक समानता, संबंधित कार्यों या [[अनुक्रम समरूपता]] के माध्यम से निर्धारित होती है। | * सदस्य: प्रोटीन का समूह जिसकी सामान्य विकासवादी उत्पत्ति संरचनात्मक समानता, संबंधित कार्यों या [[अनुक्रम समरूपता]] के माध्यम से निर्धारित होती है। | ||
* डोमेन: किसी विशेष कार्य, संरचना या अनुक्रम के साथ प्रोटीन में | * डोमेन: किसी विशेष कार्य, संरचना या अनुक्रम के साथ प्रोटीन में विशिष्ट इकाई है। | ||
*दोहराएँ: अमीनो एसिड का क्रम, सामान्यतः 50 अमीनो एसिड से अधिक नहीं, जो प्रोटीन में कई बार दोहराया जाता है। | *दोहराएँ: अमीनो एसिड का क्रम, सामान्यतः 50 अमीनो एसिड से अधिक नहीं, जो प्रोटीन में कई बार दोहराया जाता है। | ||
* साइट: अमीनो एसिड का | * साइट: अमीनो एसिड का छोटा अनुक्रम जहां कम से कम अमीनो एसिड संरक्षित होता है। इनमें [[अनुवाद के बाद का संशोधन|अनुवाद के पश्चात की संशोधन]] साइटें, संरक्षित साइटें, [[ बाध्यकारी साइट | बाध्यकारी साइटें]] एवं [[सक्रिय साइट|सक्रिय साइटें]] सम्मिलित हैं। | ||
== पहुँच == | == पहुँच == | ||
Revision as of 10:11, 15 July 2023
| File:InterPro logo.png | |
| Content | |
|---|---|
| Description | InterPro functionally analyzes protein sequences and classifies them into protein families while predicting the presence of domains and functional sites. |
| Contact | |
| Research center | EMBL |
| Laboratory | European Bioinformatics Institute |
| Primary citation | The InterPro protein families and domains database: 20 years on[1] |
| Release date | 1999 |
| Access | |
| Website | www |
| Download URL | ftp.ebi.ac.uk/pub/databases/interpro/ |
| Miscellaneous | |
| Data release frequency | 8-weekly |
| Version | 91.0 (13 October 2022) |
इंटरप्रो प्रोटीन सदस्य, प्रोटीन डोमेन एवं कार्यात्मक साइटों का डेटाबेस है जिसमें ज्ञात प्रोटीन में पाई जाने वाली पहचान योग्य विशेषताओं को कार्यात्मक रूप से चित्रित करने के लिए नए प्रोटीन अनुक्रमों पर प्रस्तावित किया जा सकता है।[2] [3][4]इंटरप्रो की सामग्री में डायग्नोस्टिक हस्ताक्षर एवं प्रोटीन सम्मिलित हैं जो महत्वपूर्ण रूप से समान होते हैं। हस्ताक्षरों में प्ररूप (सरल प्रकार, जैसे नियमित अभिव्यक्ति या अधिक कठिन, जैसे हिडन मार्कोव प्ररूप) सम्मिलित होते हैं जो प्रोटीन सदस्यों, डोमेन या साइटों का वर्णन करते हैं। प्ररूप ज्ञात सदस्यों या डोमेन के अमीनो एसिड अनुक्रमों से बनाए जाते हैं एवं पश्चात में उन्हें वर्गीकृत करने के लिए अज्ञात अनुक्रमों (जैसे कि उपन्यास जीनोम अनुक्रमण से उत्पन्न होने वाले) की शोध करने के लिए उपयोग किया जाता है। इंटरप्रो का प्रत्येक सदस्य डेटाबेस बहुत उच्च-स्तरीय, संरचना-आधारित वर्गीकरण ((सुपरफैमिली एवं CATH-Gene3D) से लेकर अधिक विशिष्ट उप-सदस्य वर्गीकरण (प्रिंट एवं पैंथर) तक, भिन्न क्षेत्र में योगदान देता है।
इंटरप्रो का इरादा प्रोटीन वर्गीकरण के लिए वन-स्टॉप-शॉप प्रदान करना है, जहां विभिन्न सदस्य डेटाबेस द्वारा उत्पादित सभी हस्ताक्षर इंटरप्रो डेटाबेस के अंदर प्रविष्टियों में रखे जाते हैं। समकक्ष डोमेन, साइटों या सदस्यों का प्रतिनिधित्व करने वाले हस्ताक्षरों को ही प्रविष्टि में रखा जाता है एवं प्रविष्टियाँ दूसरे से संबंधित भी हो सकती हैं। जहां संभव हो, अतिरिक्त जानकारी जैसे विवरण, सुसंगत नाम एवं जीन ओण्टोलॉजी (जीओ) शब्द प्रत्येक प्रविष्टि के साथ जुड़े हुए हैं।
इंटरप्रो में निहित डेटा
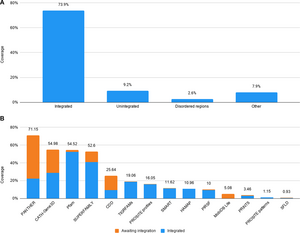
इंटरप्रो में तीन मुख्य इकाइयाँ : प्रोटीन, हस्ताक्षर (जिन्हें विधियाँ या प्ररूप भी कहा जाता है) एवं प्रविष्टियाँ सम्मिलित हैं। UniProtKB में प्रोटीन इंटरप्रो में केंद्रीय प्रोटीन इकाइयाँ भी हैं। कौन से हस्ताक्षर इन प्रोटीनों से महत्वपूर्ण रूप से समान होते हैं, इसकी जानकारी की गणना UniProtKB द्वारा अनुक्रम प्रस्तावित किए जाने पर की जाती है एवं ये परिणाम जनता के लिए उपलब्ध कराए जाते हैं (नीचे देखें)। प्रोटीन के साथ हस्ताक्षरों का मिलान यह निर्धारित करता है कि इंटरप्रो प्रविष्टियों में हस्ताक्षरों को साथ कैसे एकीकृत किया जाता है: मिलान किए गए प्रोटीन सेटों का अपेक्षात्मक ओवरलैप एवं अनुक्रमों पर हस्ताक्षरों के मिलान का स्थान संबंधितता के संकेतक के रूप में उपयोग किया जाता है। केवल पर्याप्त गुणवत्ता वाले हस्ताक्षर ही इंटरप्रो में एकीकृत किए जाते हैं। संस्करण 81.0 (21 अगस्त 2020 को प्रस्तावित) के अनुसार, इंटरप्रो प्रविष्टियों ने UniProtKB में पाए गए 73.9% अवशेषों को एनोटेट किया, अन्य 9.2% को हस्ताक्षरों द्वारा एनोटेट किया गया जो एकीकरण के लिए लंबित हैं।[5]
इंटरप्रो में स्प्लिस वेरिएंट एवं यूनीपार्क एवं यूनीएमईएस डेटाबेस में उपस्थित प्रोटीन का डेटा भी सम्मिलित है।
इंटरप्रो कंसोर्टियम सदस्य डेटाबेस
इंटरप्रो के हस्ताक्षर 13 सदस्य डेटाबेस से आते हैं, जो नीचे सूचीबद्ध हैं।
- CATH-Gene3D
- संपूर्ण जीनोम में प्रोटीन सदस्यों एवं डोमेन आर्किटेक्चर का वर्णन करता है। प्रोटीन सदस्य मार्कोव क्लस्टरिंग एल्गोरिदम का उपयोग करके बनाए जाते हैं, जिसके पश्चात अनुक्रम पहचान के अनुसार मल्टी-लिंकेज क्लस्टरिंग होती है। पूर्वानुमानित संरचना एवं अनुक्रम डोमेन का मानचित्रण CATH एवं Pfam डोमेन का प्रतिनिधित्व करने वाले छिपे हुए मार्कोव प्ररूप पुस्तकालयों का उपयोग करके किया जाता है। कई संसाधनों से प्रोटीन को कार्यात्मक एनोटेशन प्रदान किया जाता है। डोमेन आर्किटेक्चर की कार्यात्मक भविष्यवाणी एवं विश्लेषण Gene3D वेबसाइट पर उपलब्ध है।
- सीडीडी
- संरक्षित डोमेन डेटाबेस प्रोटीन एनोटेशन संसाधन है जिसमें प्राचीन डोमेन एवं पूर्ण-लंबाई प्रोटीन के लिए एनोटेटेड एकाधिक अनुक्रम संरेखण प्ररूप का संग्रह सम्मिलित है। ये आरपीएस-ब्लास्ट के माध्यम से प्रोटीन अनुक्रमों में संरक्षित डोमेन की तीव्रता से पहचान के लिए स्थिति-विशिष्ट स्कोर मैट्रिक्स (पीएसएसएम) के रूप में उपलब्ध हैं।
- HAMAP
- माइक्रोबियल प्रोटीन के उच्च गुणवत्ता वाले स्वचालित एवं मैन्युअल एनोटेशन के लिए है। HAMAP प्रोफ़ाइल विशेषज्ञ क्यूरेटर द्वारा मैन्युअल रूप से बनाई जाती हैं, वे उन प्रोटीनों की पहचान करते हैं जो संरक्षित बैक्टीरिया, आर्कियल एवं प्लास्टिड-एनकोडेड (यानी क्लोरोप्लास्ट, साइनेल, एपिकोप्लास्ट, गैर-प्रकाश संश्लेषक प्लास्टिड) प्रोटीन सदस्यों या उपसदस्यों का भाग हैं।
- MobiDB
- MobiDB प्रोटीन में आंतरिक विकार की व्याख्या करने वाला डेटाबेस है।
- पैंथर
- पैंथर प्रोटीन सदस्यों का बड़ा संग्रह है जिसे मानव विशेषज्ञता का उपयोग करके कार्यात्मक रूप से संबंधित उप-सदस्यों में विभाजित किया गया है। ये उपसदस्य प्रोटीन सदस्यों के अंदर विशिष्ट कार्यों के विचलन को प्ररूप करते हैं, जिससे फ़ंक्शन (मानव-क्यूरेटेड आणविक फ़ंक्शन एवं जैविक प्रक्रिया वर्गीकरण एवं मार्ग आरेख) के साथ अधिक त्रुटिहीन जुड़ाव की अनुमति मिलती है, साथ ही कार्यात्मक विशिष्टता के लिए महत्वपूर्ण अमीनो एसिड का अनुमान भी लगाया जा सकता है। अतिरिक्त प्रोटीन अनुक्रमों को वर्गीकृत करने के लिए प्रत्येक सदस्य एवं उपसदस्य के लिए हिडन मार्कोव प्ररूप (एचएमएम) बनाए गए हैं।
- Pfam
- कई अनुक्रम संरेखण और छिपे हुए मार्कोव मॉडल का बड़ा संग्रह है जो कई सामान्य प्रोटीन डोमेन और परिवारों को कवर करता है। पीआईआरएसएफ प्रोटीन वर्गीकरण प्रणाली सुपरफैमिली से उपफैमिली तक अनुक्रम विविधता के कई स्तरों वाला नेटवर्क है जो पूर्ण-लंबाई प्रोटीन एवं डोमेन के विकासवादी संबंध को दर्शाता है। प्राथमिक पीआईआरएसएफ वर्गीकरण इकाई होमोमोर्फिक सदस्य है, जिसके सदस्य समजात ( सामान्य पूर्वज से विकसित) एवं होमोमोर्फिक (पूर्ण लंबाई अनुक्रम समानता एवं सामान्य डोमेन वास्तुकला साझा करने वाले) दोनों हैं।
- प्रिंट्स
- प्रिंट्स प्रोटीन फ़िंगरप्रिंट्स का संग्रह है। फ़िंगरप्रिंट संरक्षित रूपांकनों का समूह है जिसका उपयोग प्रोटीन सदस्य को चित्रित करने के लिए किया जाता है; इसकी नैदानिक बल को UniProt की पुनरावृत्तीय स्कैनिंग द्वारा परिष्कृत किया जाता है। सामान्यतः रूपांकन ओवरलैप नहीं होते हैं, बल्कि अनुक्रम के साथ भिन्न हो जाते हैं, हालांकि वे 3डी-स्पेस में सन्निहित हो सकते हैं। फ़िंगरप्रिंट ल रूपांकनों की अपेक्षा में प्रोटीन सिलवटों एवं कार्यात्मकताओं को अधिक लचीले एवं बलशाली ढंग से एनकोड कर सकते हैं, उनकी पूर्ण नैदानिक क्षमता रूपांकन पड़ोसियों द्वारा प्रदान किए गए पारस्परिक संदर्भ से प्राप्त होती है।
- प्रोसाइट
- प्रोसाइट प्रोटीन सदस्यों एवं डोमेन का डेटाबेस है। इसमें जैविक रूप से महत्वपूर्ण साइटें, पैटर्न एवं प्रोफाइल सम्मिलित हैं जो विश्वसनीय रूप से यह पहचानने में सहायता करते हैं कि नया अनुक्रम किस ज्ञात प्रोटीन सदस्य से संबंधित है।
- स्मार्ट
- सरल मॉड्यूलर वास्तुकला अनुसंधान उपकरण आनुवंशिक रूप से मोबाइल डोमेन की पहचान एवं एनोटेशन एवं डोमेन आर्किटेक्चर के विश्लेषण की अनुमति देता है। सिग्नलिंग, बाह्यकोशिकीय एवं क्रोमैटिन से जुड़े प्रोटीन में पाए जाने वाले 800 से अधिक डोमेन सदस्य ज्ञात करने योग्य हैं। इन डोमेन को फ़ाइलेटिक वितरण, कार्यात्मक वर्ग, तृतीयक संरचनाओं एवं कार्यात्मक रूप से महत्वपूर्ण अवशेषों के संबंध में बड़े स्तर पर एनोटेट किया गया है।
- सुपरफैमिली
- सुपरफैमिली प्रोफाइल छिपे हुए मार्कोव प्ररूप की लाइब्रेरी है जो ज्ञात संरचना के सभी प्रोटीन का प्रतिनिधित्व करती है। लाइब्रेरी प्रोटीन के संरचनात्मक वर्गीकरण डेटाबेस प्रोटीन के वर्गीकरण पर आधारित है: प्रत्येक प्ररूप एससीओपी डोमेन के समान है एवं इसका उद्देश्य पूरे एससीओपी प्रोटीन सुपरफैमिली का प्रतिनिधित्व करना है जो डोमेन से संबंधित है। सुपरफ़ैमिली का उपयोग सभी पूर्णतः अनुक्रमित जीनोमों में संरचनात्मक कार्य करने के लिए किया गया है।
- एसएफएलडी
- एंजाइमों का श्रेणीबद्ध वर्गीकरण जो विशिष्ट अनुक्रम-संरचना विशेषताओं को विशिष्ट रासायनिक क्षमताओं से जोड़ता है।
- TIGRFAMs
- TIGRFAMs प्रोटीन सदस्यों का संग्रह है, जिसमें क्यूरेटेड मल्टीपल अनुक्रम संरेखण, छिपे हुए मार्कोव प्ररूप (HMM) एवं एनोटेशन सम्मिलित हैं, जो अनुक्रम होमोलॉजी के आधार पर कार्यात्मक रूप से संबंधित प्रोटीन की पहचान करने के लिए उपकरण प्रदान करता है। वे प्रविष्टियाँ जो समतुल्य समूह हैं, समजात प्रोटीन हैं जो कार्य के संबंध में संरक्षित हैं।
डेटा प्रकार
इंटरप्रो में कंसोर्टियम के विभिन्न सदस्यों द्वारा प्रदान किए गए सात प्रकार के डेटा सम्मिलित हैं:
| Data Type | Description | Contributing Databases |
|---|---|---|
| InterPro Entries | Structural and/or functional domains of proteins predicted using one or more signatures | All 13 member databases |
| Member Database signatures | Signatures from member databases. These include signatures that are integrated into InterPro, and those that are not | All 13 member databases |
| Protein | Protein sequences | UniProtKB (Swiss-Prot and TrEMBL) |
| Proteome | Collection of proteins that belong to a single organism | UniProtKB |
| Structure | 3-dimensional structures of proteins | PDBe |
| Taxonomy | Protein taxonomic information | UniProtKB |
| Set | Groups of evolutionary related families | Pfam, CDD |

इंटरप्रो प्रविष्टि प्रकार
इंटरप्रो प्रविष्टियों को पाँच प्रकारों में विभाजित किया जा सकता है:
- समजात सुपरफैमिली: प्रोटीन का समूह जो समान विकासवादी उत्पत्ति साझा करता है जैसा कि उनकी संरचनात्मक समानता में देखा जाता है, अपितु उनके अनुक्रम अत्यधिक समान न हों। ये प्रविष्टियाँ विशेष रूप से केवल दो सदस्य डेटाबेस : CATH-Gene3D एवं सुपरफ़ैमिली द्वारा प्रदान की जाती हैं।
- सदस्य: प्रोटीन का समूह जिसकी सामान्य विकासवादी उत्पत्ति संरचनात्मक समानता, संबंधित कार्यों या अनुक्रम समरूपता के माध्यम से निर्धारित होती है।
- डोमेन: किसी विशेष कार्य, संरचना या अनुक्रम के साथ प्रोटीन में विशिष्ट इकाई है।
- दोहराएँ: अमीनो एसिड का क्रम, सामान्यतः 50 अमीनो एसिड से अधिक नहीं, जो प्रोटीन में कई बार दोहराया जाता है।
- साइट: अमीनो एसिड का छोटा अनुक्रम जहां कम से कम अमीनो एसिड संरक्षित होता है। इनमें अनुवाद के पश्चात की संशोधन साइटें, संरक्षित साइटें, बाध्यकारी साइटें एवं सक्रिय साइटें सम्मिलित हैं।
पहुँच
डेटाबेस वेबसर्वर के माध्यम से पाठ एवं अनुक्रम-आधारित शोधों के लिए एवं अनाम एफ़टीपी के माध्यम से डाउनलोड के लिए उपलब्ध है। अन्य यूरोपीय जैव सूचना विज्ञान संस्थान डेटाबेस की तरह, यह सार्वजनिक डोमेन में है, क्योंकि इसकी सामग्री का उपयोग कोई भी व्यक्ति एवं किसी भी उद्देश्य के लिए कर सकता है।[8] इंटरप्रो का लक्ष्य हर 8 सप्ताह में जनता के लिए डेटा प्रस्तावित करना है, सामान्यतः समान प्रोटीन के UniProtKB रिलीज के दिन के अंदर।
इंटरप्रो एप्लिकेशन प्रोग्रामिंग इंटरफ़ेस (एपीआई)
इंटरप्रो JSON प्रारूप में सभी इंटरप्रो प्रविष्टियों एवं उनकी संबंधित प्रविष्टियों तक प्रोग्रामेटिक पहुंच के लिए एपीआई प्रदान करता है।[9] विभिन्न इंटरप्रो डेटा प्रकारों के अनुरूप एपीआई के लिए छह मुख्य समापन बिंदु हैं: प्रविष्टि, प्रोटीन, संरचना, वर्गीकरण, प्रोटिओम एवं सेट।
इंटरप्रोस्कैन
इंटरप्रोस्कैन सॉफ्टवेयर पैकेज है जो उपयोगकर्ताओं को सदस्य डेटाबेस हस्ताक्षरों के विरुद्ध अनुक्रमों को स्कैन करने की अनुमति देता है। उपयोगकर्ता इस हस्ताक्षर स्कैनिंग सॉफ़्टवेयर का उपयोग नवीन न्यूक्लियोटाइड या प्रोटीन अनुक्रमों को कार्यात्मक रूप से चिह्नित करने के लिए कर सकते हैं।[10] रुचि के जीनोम का प्रथम-पास लक्षण वर्णन प्राप्त करने के लिए जीनोम परियोजनाओं में अक्सर इंटरप्रोस्कैन का उपयोग किया जाता है।[11][12] As of December 2020[update], इंटरप्रोस्कैन (v5.x) का सार्वजनिक संस्करण जावा (प्रोग्रामिंग भाषा)|जावा-आधारित आर्किटेक्चर का उपयोग करता है।[13] सॉफ़्टवेयर पैकेज वर्तमान में केवल 64-बिट लिनक्स ऑपरेटिंग सिस्टम पर समर्थित है।
इंटरप्रोस्कैन, कई अन्य ईएमबीएल-ईबीआई जैव सूचना विज्ञान उपकरणों के साथ, प्रतिनिधित्ववादी स्थिति में स्थानांतरण एवं एसओएपी वेब सर्विसेज एपीआई का उपयोग करके प्रोग्रामेटिक रूप से भी ्सेस किया जा सकता है।[14]
यह भी देखें
- प्रोटीन सदस्य
- अज्ञात फ़ंक्शन का डोमेन
- अनुक्रम आकृति
संदर्भ
- ↑ Blum M, Chang HY, Chuguransky S, Grego T, Kandasaamy S, Mitchell A, et al. (November 2020). "The InterPro protein families and domains database: 20 years on". Nucleic Acids Research. 49 (D1): D344–D354. doi:10.1093/nar/gkaa977. PMC 7778928. PMID 33156333.
- ↑ Hunter S, Jones P, Mitchell A, Apweiler R, Attwood TK, Bateman A, et al. (January 2012). "InterPro in 2011: new developments in the family and domain prediction database". Nucleic Acids Research. 40 (Database issue): D306-12. doi:10.1093/nar/gkr948. PMC 3245097. PMID 22096229.
- ↑ Apweiler R, Attwood TK, Bairoch A, Bateman A, Birney E, Biswas M, et al. (January 2001). "इंटरप्रो डेटाबेस, प्रोटीन परिवारों, डोमेन और कार्यात्मक साइटों के लिए एक एकीकृत दस्तावेज़ीकरण संसाधन". Nucleic Acids Research. 29 (1): 37–40. doi:10.1093/nar/29.1.37. PMC 29841. PMID 11125043.
- ↑ Apweiler R, Attwood TK, Bairoch A, Bateman A, Birney E, Biswas M, et al. (December 2000). "इंटरप्रो--प्रोटीन परिवारों, डोमेन और कार्यात्मक साइटों के लिए एक एकीकृत दस्तावेज़ीकरण संसाधन". Bioinformatics. 16 (12): 1145–50. doi:10.1093/bioinformatics/16.12.1145. PMID 11159333.
- ↑ 5.0 5.1 Blum, Matthias; Chang, Hsin-Yu; Chuguransky, Sara; Grego, Tiago; Kandasaamy, Swaathi; Mitchell, Alex; Nuka, Gift; Paysan-Lafosse, Typhaine; Qureshi, Matloob; Raj, Shriya; Richardson, Lorna (2020-11-06). "The InterPro protein families and domains database: 20 years on". Nucleic Acids Research (in English). 49 (D1): D344–D354. doi:10.1093/nar/gkaa977. ISSN 0305-1048. PMC 7778928. PMID 33156333.
- ↑ EMBL-EBI. "Where does the data come from? | InterPro" (in English). Retrieved 2020-12-04.
- ↑ EMBL-EBI. "InterPro entry types | InterPro" (in English). Retrieved 2020-12-04.
- ↑ "Terms of Use for EMBL-EBI Services | European Bioinformatics Institute".
- ↑ "How to download InterPro data? — InterPro Documentation". interpro-documentation.readthedocs.io. Retrieved 2020-12-04.
- ↑ Quevillon E, Silventoinen V, Pillai S, Harte N, Mulder N, Apweiler R, Lopez R (July 2005). "InterProScan: protein domains identifier" (Free full text). Nucleic Acids Research. 33 (Web Server issue): W116-20. doi:10.1093/nar/gki442. PMC 1160203. PMID 15980438.
- ↑ Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (February 2001). "प्रारंभिक अनुक्रमण और मानव जीनोम का विश्लेषण" (PDF). Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ↑ Holt RA, Subramanian GM, Halpern A, Sutton GG, Charlab R, Nusskern DR, et al. (October 2002). "मलेरिया मच्छर एनोफ़ेलीज़ गैम्बिया का जीनोम अनुक्रम". Science. 298 (5591): 129–49. Bibcode:2002Sci...298..129H. CiteSeerX 10.1.1.149.9058. doi:10.1126/science.1076181. PMID 12364791. S2CID 4512225.
- ↑ Jones P, Binns D, Chang HY, Fraser M, Li W, McAnulla C, et al. (May 2014). "InterProScan 5: genome-scale protein function classification". Bioinformatics. 30 (9): 1236–40. doi:10.1093/bioinformatics/btu031. PMC 3998142. PMID 24451626.
- ↑ Madeira F, Park YM, Lee J, Buso N, Gur T, Madhusoodanan N, et al. (July 2019). "The EMBL-EBI search and sequence analysis tools APIs in 2019". Nucleic Acids Research. 47 (W1): W636–W641. doi:10.1093/nar/gkz268. PMC 6602479. PMID 30976793.
बाहरी संबंध
- Official website — webserver