अनुक्रम संयोजन: Difference between revisions
No edit summary |
No edit summary |
||
| Line 45: | Line 45: | ||
1) प्री-असेंबली: यह चरण डाउनलाइन विश्लेषण जैसे वेरिएंट कॉलिंग या अंतिम मचान अनुक्रम की अखंडता सुनिश्चित करने के लिए आवश्यक है। इस चरण में दो कालानुक्रमिक वर्कफ़्लो सम्मिलित हैं: | 1) प्री-असेंबली: यह चरण डाउनलाइन विश्लेषण जैसे वेरिएंट कॉलिंग या अंतिम मचान अनुक्रम की अखंडता सुनिश्चित करने के लिए आवश्यक है। इस चरण में दो कालानुक्रमिक वर्कफ़्लो सम्मिलित हैं: | ||
ए) गुणवत्ता जांच: अनुक्रमण विधि के प्रकार के आधार पर, विभिन्न त्रुटियां उत्पन्न हो सकती हैं जो गलत [[बेस कॉलिंग]] का कारण बन सकती हैं। उदाहरण के लिए, NAAAAAAAAAAAAN और | ए) गुणवत्ता जांच: अनुक्रमण विधि के प्रकार के आधार पर, विभिन्न त्रुटियां उत्पन्न हो सकती हैं जो गलत [[बेस कॉलिंग]] का कारण बन सकती हैं। उदाहरण के लिए, "NAAAAAAAAAAAAN" और "NAAAAAAAAAAAN" का अनुक्रम जिसमें 12 एडेनिन सम्मिलित है, को गलत तरीके से इसके अतिरिक्त 11 एडेनिन कहा जा सकता है। लक्ष्य डीएनए/आरएनए के अत्यधिक दोहराव वाले खंड को अनुक्रमित करने के परिणामस्वरूप छोटी या अधिक आधार वाली कॉल हो सकती है। पढ़ने की गुणवत्ता सामान्यतः Phred गुणवत्ता स्कोर द्वारा मापी जाती है जो कि पढ़ने के अनुक्रम के अंदर प्रत्येक न्यूक्लियोटाइड गुणवत्ता का एन्कोडेड स्कोर है। कुछ अनुक्रमण विधि जैसे [[ पचबियो |पचबियो]] में उनके अनुक्रमित पढ़ने के लिए कोई स्कोरिंग विधि नहीं है। इस चरण में उपयोग किया जाने वाला सामान्य उपकरण FastQC है।<ref>{{Cite web |title=बब्राहम जैव सूचना विज्ञान - फास्टक्यूसी उच्च थ्रूपुट अनुक्रम डेटा के लिए एक गुणवत्ता नियंत्रण उपकरण|url=https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ |access-date=2022-05-09 |website=www.bioinformatics.babraham.ac.uk}}</ref> बी) रीड्स को फ़िल्टर करना: जो रीड्स गुणवत्ता जांच पास करने में विफल रहे, उन्हें सर्वोत्तम असेंबली कॉन्टिग्स प्राप्त करने के लिए फास्टक्यू फ़ाइल से हटा दिया जाना चाहिए। | ||
2) असेंबली: इस चरण के समय, प्रत्येक रीड को संभावित स्थान पर मैप करने के लिए विभिन्न मानदंडों के साथ रीड अलाइनमेंट का उपयोग किया जाएगा। किसी पाठ की अनुमानित स्थिति या तब इस पर आधारित होती है कि उसका अनुक्रम कितना अन्य पाठों या किसी संदर्भ के साथ संरेखित होता है। विभिन्न अनुक्रमण प्रौद्योगिकियों से पढ़ने के लिए विभिन्न संरेखण एल्गोरिदम का उपयोग किया जाता है। असेंबली में सामान्यतः उपयोग किए जाने वाले कुछ दृष्टिकोण डी ब्रुइज़न अनुक्रम ग्राफ और ओवरलैपिंग हैं। लंबाई, [[कवरेज (आनुवांशिकी)]], गुणवत्ता और उपयोग की गई अनुक्रमण विधि पढ़ें डीएनए अनुक्रमण के स्थितियों में सर्वोत्तम संरेखण एल्गोरिदम चुनने में प्रमुख भूमिका निभाती है।<ref>{{Cite journal |last1=Ruffalo |first1=M. |last2=LaFramboise |first2=T. |last3=Koyuturk |first3=M. |date=2011-10-15 |title=अगली पीढ़ी के अनुक्रमण रीड अलाइनमेंट के लिए एल्गोरिदम का तुलनात्मक विश्लेषण|journal=Bioinformatics |language=en |volume=27 |issue=20 |pages=2790–2796 |doi=10.1093/bioinformatics/btr477 |pmid=21856737 |issn=1367-4803|doi-access=free }}</ref> दूसरी ओर, तीसरी पीढ़ी के अनुक्रमण को संरेखित करने वाले एल्गोरिदम को उनके साथ जुड़ी उच्च त्रुटि दर को ध्यान में रखते हुए अग्रिम दृष्टिकोण की आवश्यकता होती है। | 2) असेंबली: इस चरण के समय, प्रत्येक रीड को संभावित स्थान पर मैप करने के लिए विभिन्न मानदंडों के साथ रीड अलाइनमेंट का उपयोग किया जाएगा। किसी पाठ की अनुमानित स्थिति या तब इस पर आधारित होती है कि उसका अनुक्रम कितना अन्य पाठों या किसी संदर्भ के साथ संरेखित होता है। विभिन्न अनुक्रमण प्रौद्योगिकियों से पढ़ने के लिए विभिन्न संरेखण एल्गोरिदम का उपयोग किया जाता है। असेंबली में सामान्यतः उपयोग किए जाने वाले कुछ दृष्टिकोण डी ब्रुइज़न अनुक्रम ग्राफ और ओवरलैपिंग हैं। लंबाई, [[कवरेज (आनुवांशिकी)]], गुणवत्ता और उपयोग की गई अनुक्रमण विधि पढ़ें डीएनए अनुक्रमण के स्थितियों में सर्वोत्तम संरेखण एल्गोरिदम चुनने में प्रमुख भूमिका निभाती है।<ref>{{Cite journal |last1=Ruffalo |first1=M. |last2=LaFramboise |first2=T. |last3=Koyuturk |first3=M. |date=2011-10-15 |title=अगली पीढ़ी के अनुक्रमण रीड अलाइनमेंट के लिए एल्गोरिदम का तुलनात्मक विश्लेषण|journal=Bioinformatics |language=en |volume=27 |issue=20 |pages=2790–2796 |doi=10.1093/bioinformatics/btr477 |pmid=21856737 |issn=1367-4803|doi-access=free }}</ref> दूसरी ओर, तीसरी पीढ़ी के अनुक्रमण को संरेखित करने वाले एल्गोरिदम को उनके साथ जुड़ी उच्च त्रुटि दर को ध्यान में रखते हुए अग्रिम दृष्टिकोण की आवश्यकता होती है। | ||
| Line 94: | Line 94: | ||
!सॉफ़्टवेयर | !सॉफ़्टवेयर | ||
!पढ़ें प्रकार | !पढ़ें प्रकार | ||
! | !टूल वेब पेज | ||
! | !टिप्पणियाँ | ||
|- | |- | ||
|फास्टक्यूसी | |फास्टक्यूसी | ||
Revision as of 13:22, 18 July 2023
जैव सूचना विज्ञान में, अनुक्रम असेंबली का तात्पर्य मूल अनुक्रम के पुनर्निर्माण के लिए अनुक्रम संरेखण और लंबे डीएनए अनुक्रम से टुकड़ों को संरेखित करना और विलय करना है। इसकी आवश्यकता है क्योंकि डीएनए अनुक्रमण विधि बार में पूरे जीनोम को 'पढ़ने' में सक्षम नहीं हो सकती है, किंतु उपयोग की गई विधि के आधार पर 20 से 30,000 आधारों के मध्य के छोटे टुकड़ों को पढ़ती है। सामान्यतः, छोटे टुकड़े (रीड्स) शॉटगन अनुक्रमण जीनोम डीएनए, या प्रतिलेखन (आनुवांशिकी) (व्यक्त अनुक्रम टैग) से उत्पन्न होते हैं।
अनुक्रम संयोजन की समस्या की तुलना किसी पुस्तक की कई प्रतियां लेने, उनमें से प्रत्येक को अलग कटर के साथ श्रेडर से गुजारने और कटे हुए टुकड़ों को देखकर पुस्तक के पाठ को वापस साथ जोड़ने से की जा सकती है। इस कार्य की स्पष्ट कठिनाई के अतिरिक्त, कुछ अतिरिक्त व्यावहारिक मुद्दे भी हैं: मूल में कई दोहराए गए पैराग्राफ हो सकते हैं, और टाइपो त्रुटियों के लिए श्रेडिंग के समय कुछ टुकड़ों को संशोधित किया जा सकता है। किसी अन्य पुस्तक के अंश भी इसमें जोड़े जा सकते हैं और कुछ अंश पूरी तरह से पहचानने योग्य नहीं हो सकते हैं।
जीनोम असेंबलर
पहला अनुक्रम असेंबलर 1980 के दशक के अंत और 1990 के दशक की शुरुआत में डीएनए सीक्वेंसर कहे जाने वाले स्वचालित अनुक्रमण उपकरणों द्वारा उत्पन्न बड़ी मात्रा में टुकड़ों को साथ जोड़ने के लिए सरल अनुक्रम संरेखण कार्यक्रमों के वेरिएंट के रूप में दिखाई देने लगा। जैसे-जैसे अनुक्रमित जीवों का आकार और जटिलता बढ़ती गई (प्लाज्मिड्स पर छोटे वायरस से लेकर जीवाणु और अंततः यूकैर्योसाइटों तक), इन जीनोम परियोजनाओं में उपयोग किए जाने वाले असेंबली कार्यक्रमों को संभालने के लिए तेजी से परिष्कृत रणनीतियों की आवश्यकता थी:
- अनुक्रमण डेटा के टेराबाइट्स जिन्हें क्लस्टर कंप्यूटिंग पर प्रसंस्करण की आवश्यकता होती है;
- समान और लगभग समान अनुक्रम (दोहराव के रूप में जाना जाता है) जो, सबसे खराब स्थिति में, एल्गोरिदम की समय और स्थान जटिलता को चतुष्कोणीय रूप से बढ़ा सकता है;
- डीएनए अनुक्रमण उपकरणों से टुकड़ों में त्रुटियों को पढ़ता है, जो असेंबली को भ्रमित कर सकता है।
पहले बड़े यूकेरियोटिक जीनोम - 2000 में फल मक्खी ड्रोसोफिला मेलानोगास्टर और ठीक साल पश्चात् मानव जीनोम - को असेंबल करने की चुनौती का सामना करते हुए, वैज्ञानिकों ने सेलेरा असेंबलर जैसे असेंबलर विकसित किए[1] और अर्चन[2] 130 मिलियन (उदाहरण के लिए, फल मक्खी डी. मेलानोगास्टर) से 3 बिलियन (उदाहरण के लिए, मानव जीनोम) आधार जोड़े के जीनोम को संभालने में सक्षम। इन प्रयासों के पश्चात्, कई अन्य समूहों ने, अधिकतर प्रमुख जीनोम अनुक्रमण केंद्रों पर, बड़े पैमाने पर असेंबलर बनाए, और खुला स्रोत प्रयास जिसे एएमओएस के नाम से जाना जाता है[3] खुला स्रोत सॉफ्टवेयर ढांचे के अनुसार जीनोम असेंबली विधि में सभी नवाचारों को साथ लाने के लिए लॉन्च किया गया था।
ईएसटी असेंबलर
व्यक्त अनुक्रम टैग या ईएसटी असेंबली प्रारंभिक रणनीति थी, जो 1990 के दशक के मध्य से लेकर 2000 के दशक के मध्य तक, पूरे जीनोम के अतिरिक्त व्यक्तिगत जीन को इकट्ठा करने के लिए थी। समस्या कई मायनों में जीनोम असेंबली से भिन्न है। ईएसटी असेंबली के लिए इनपुट अनुक्रम कोशिका के लिखित मैसेंजर आरएनए के टुकड़े हैं और पूरे जीनोम के केवल उपसमूह का प्रतिनिधित्व करते हैं। जीनोम और ईएसटी असेंबली के मध्य कई एल्गोरिथम संबंधी समस्याएं भिन्न होती हैं। उदाहरण के लिए, जीनोम में अधिकांशतः बड़ी मात्रा में दोहराव वाले अनुक्रम होते हैं, जो इंटरजेनिक क्षेत्रों में केंद्रित होते हैं। प्रतिलेखित जीन में बहुत कम दोहराव होते हैं, जिससे संयोजन कुछ सीमा तक आसान हो जाता है। दूसरी ओर, कुछ जीनों को बहुत अधिक संख्या में व्यक्त (प्रतिलेखित) किया जाता है (उदाहरण के लिए, हाउसकीपिंग जीन), जिसका अर्थ है कि पूरे जीनोम शॉटगन अनुक्रमण के विपरीत, रीड्स को पूरे जीनोम में समान रूप से नमूना नहीं किया जाता है।
ईएसटी असेंबली को (सीआईएस-) वैकल्पिक स्प्लिसिंग, ट्रांस स्प्लिसिंग , एकल न्यूकलोटाइड बहुरूपता और पोस्ट-ट्रांसक्रिप्शनल संशोधन जैसी सुविधाओं द्वारा और अधिक जटिल बना दिया गया है। 2008 की शुरुआत में जब आरएनए-सेक का आविष्कार किया गया था, ईएसटी अनुक्रमण को इस कहीं अधिक कुशल विधि से बदल दिया गया था, जिसे नई ट्रांस्क्रिप्टोम असेंबली से के अनुसार वर्णित किया गया था।
अनुक्रम संयोजन के प्रकार
अनुक्रमण डेटा को इकट्ठा करने के तीन दृष्टिकोण हैं:
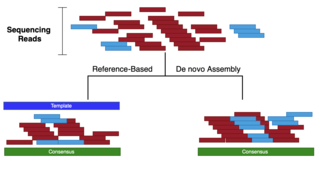
- डी-नोवो: टेम्प्लेट का उपयोग किए बिना, अनुक्रमण को असेंबल करने से पूर्ण-लंबाई (कभी-कभी उपन्यास) अनुक्रम तैयार होते हैं (डे नोवो अनुक्रम असेंबलर, डे नोवो ट्रांस्क्रिप्टोम असेंबली देखें)
- मानचित्रण/संरेखित करना: रीड्स को टेम्प्लेट (AKA संदर्भ) के विरुद्ध संरेखित करके असेंबल करना। एकत्रित सर्वसम्मति टेम्पलेट के समान नहीं हो सकती है।
- संदर्भ-निर्देशित: संदर्भ के अंदर सबसे समान क्षेत्र की समानता के आधार पर पठन का समूहन (चरणवार मानचित्रण)। फिर प्रत्येक समूह में पढ़े गए पाठों को लघु पाठ्य गुणवत्ता की नकल करने के लिए छोटा कर दिया जाता है। ऐसा करने की विशिष्ट विधि के-अधिक दृष्टिकोण है। तीसरी पीढ़ी की अनुक्रमण|लांग-रीड्स का उपयोग करके संदर्भ-निर्देशित असेंबली सबसे उपयोगी है।
संदर्भित-निर्देशित असेंबली अन्य प्रकारों का संयोजन है। इस प्रकार को बड़े पैमाने पर समानांतर अनुक्रमण लाभ (अर्थात कॉल गुणवत्ता) की नकल करने के लिए लंबे समय तक पढ़ी जाने वाली अनुक्रमणिका पर प्रयुक्त किया जाता है। इसके पीछे तर्क संदर्भ के अंदर छोटी विंडो द्वारा रीड्स को समूहीकृत करना है। उच्चतम गुणवत्ता और सबसे संभावित सन्निहित (कॉन्टिग) का चयन करने के लिए के-मात्र दृष्टिकोण का उपयोग करके प्रत्येक समूह में रीड्स को आकार में कम किया जाएगा। फिर मचान बनाने के लिए कॉन्टिग्स को साथ जोड़ा जाएगा। अंतिम सहमति मचान में किसी भी अंतराल को बंद करके बनाई जाती है।
डी-नोवो बनाम मानचित्रण असेंबली
जटिलता और समय की आवश्यकताओं के संदर्भ में, डे-नोवो असेंबली मानचित्रण असेंबली की तुलना में धीमी और अधिक मेमोरी गहन होती हैं। यह अधिकतर इस तथ्य के कारण है कि असेंबली एल्गोरिदम को प्रत्येक रीड की तुलना हर दूसरे रीड के साथ करने की आवश्यकता होती है (एक ऑपरेशन जिसमें O(n की अनुभवहीन समय जटिलता होती है)2)). वर्तमान डे-नोवो जीनोम असेंबलर विभिन्न प्रकार के ग्राफ़-आधारित एल्गोरिदम का उपयोग कर सकते हैं, जैसे:
- ओवरलैप/लेआउट/आम सहमति (ओएलसी) दृष्टिकोण, जो सेंगर-डेटा असेंबलरों के लिए विशिष्ट था और ओवरलैप ग्राफ़ पर निर्भर करता है।
- डी ब्रुइज़न ग्राफ़ (डीबीजी) दृष्टिकोण, जो सोलेक्सा और एसओएलआईडी प्लेटफार्मों से लघु रीड्स के लिए सबसे व्यापक रूप से प्रयुक्त होता है। यह के-मेर ग्राफ़ पर निर्भर करता है, जो बड़ी मात्रा में लघु पठन के साथ अच्छा प्रदर्शन करता है।
- लालची ग्राफ़-आधारित दृष्टिकोण, जो OLC या DBG दृष्टिकोणों में से किसी का भी उपयोग कर सकता है। लालची ग्राफ-आधारित एल्गोरिदम के साथ, contigs लालची विस्तार से आगे बढ़ें, सदैव उच्चतम स्कोरिंग ओवरलैप का पालन करके जो पढ़ा जाता है उसे लेते रहें।[4]
परिचय में कटी हुई किताबों से की गई तुलना का जिक्र करते हुए: जबकि असेंबली मानचित्रण के लिए टेम्पलेट के रूप में बहुत ही समान पुस्तक होगी (संभवतः मुख्य पात्रों के नाम और कुछ स्थानों को बदल दिया गया है), डे-नोवो असेंबली और अधिक चुनौतीपूर्ण प्रस्तुत करती है इसमें चुनौती यह है कि किसी को पहले से पता नहीं चलेगा कि यह विज्ञान पुस्तक, उपन्यास, कैटलॉग या यहां तक कि कई किताबें बन जाएंगी। साथ ही, प्रत्येक टुकड़े की तुलना हर दूसरे टुकड़े से की जाएगी।
डे-नोवो असेंबली में दोहराव को संभालने के लिए निकटतम दोहराव का प्रतिनिधित्व करने वाले ग्राफ़ सिद्धांत के निर्माण की आवश्यकता होती है। इस तरह की जानकारी पूर्ण या शॉटगन_सीक्वेंसिंग#पेयर्ड-एंड_सीक्वेंसिंग में दोहराव को कवर करने वाले लंबे टुकड़े को पढ़ने से प्राप्त की जा सकती है। दूसरी ओर, मानचित्रण असेंबली में, कई या बिना मिलान वाले भागों को सामान्यतः किसी अन्य संयोजन विधि पर गौर करने के लिए छोड़ दिया जाता है।[5]
अनुक्रम असेंबली पाइपलाइन (जैव सूचना विज्ञान)
सामान्यतः, अनुक्रमण को मचान में इकट्ठा करने में तीन चरण होते हैं:
1) प्री-असेंबली: यह चरण डाउनलाइन विश्लेषण जैसे वेरिएंट कॉलिंग या अंतिम मचान अनुक्रम की अखंडता सुनिश्चित करने के लिए आवश्यक है। इस चरण में दो कालानुक्रमिक वर्कफ़्लो सम्मिलित हैं:
ए) गुणवत्ता जांच: अनुक्रमण विधि के प्रकार के आधार पर, विभिन्न त्रुटियां उत्पन्न हो सकती हैं जो गलत बेस कॉलिंग का कारण बन सकती हैं। उदाहरण के लिए, "NAAAAAAAAAAAAN" और "NAAAAAAAAAAAN" का अनुक्रम जिसमें 12 एडेनिन सम्मिलित है, को गलत तरीके से इसके अतिरिक्त 11 एडेनिन कहा जा सकता है। लक्ष्य डीएनए/आरएनए के अत्यधिक दोहराव वाले खंड को अनुक्रमित करने के परिणामस्वरूप छोटी या अधिक आधार वाली कॉल हो सकती है। पढ़ने की गुणवत्ता सामान्यतः Phred गुणवत्ता स्कोर द्वारा मापी जाती है जो कि पढ़ने के अनुक्रम के अंदर प्रत्येक न्यूक्लियोटाइड गुणवत्ता का एन्कोडेड स्कोर है। कुछ अनुक्रमण विधि जैसे पचबियो में उनके अनुक्रमित पढ़ने के लिए कोई स्कोरिंग विधि नहीं है। इस चरण में उपयोग किया जाने वाला सामान्य उपकरण FastQC है।[6] बी) रीड्स को फ़िल्टर करना: जो रीड्स गुणवत्ता जांच पास करने में विफल रहे, उन्हें सर्वोत्तम असेंबली कॉन्टिग्स प्राप्त करने के लिए फास्टक्यू फ़ाइल से हटा दिया जाना चाहिए।
2) असेंबली: इस चरण के समय, प्रत्येक रीड को संभावित स्थान पर मैप करने के लिए विभिन्न मानदंडों के साथ रीड अलाइनमेंट का उपयोग किया जाएगा। किसी पाठ की अनुमानित स्थिति या तब इस पर आधारित होती है कि उसका अनुक्रम कितना अन्य पाठों या किसी संदर्भ के साथ संरेखित होता है। विभिन्न अनुक्रमण प्रौद्योगिकियों से पढ़ने के लिए विभिन्न संरेखण एल्गोरिदम का उपयोग किया जाता है। असेंबली में सामान्यतः उपयोग किए जाने वाले कुछ दृष्टिकोण डी ब्रुइज़न अनुक्रम ग्राफ और ओवरलैपिंग हैं। लंबाई, कवरेज (आनुवांशिकी), गुणवत्ता और उपयोग की गई अनुक्रमण विधि पढ़ें डीएनए अनुक्रमण के स्थितियों में सर्वोत्तम संरेखण एल्गोरिदम चुनने में प्रमुख भूमिका निभाती है।[7] दूसरी ओर, तीसरी पीढ़ी के अनुक्रमण को संरेखित करने वाले एल्गोरिदम को उनके साथ जुड़ी उच्च त्रुटि दर को ध्यान में रखते हुए अग्रिम दृष्टिकोण की आवश्यकता होती है।
3) पोस्ट असेंबली: यह चरण असेंबल किए गए अनुक्रम से बहुमूल्य जानकारी निकालने पर केंद्रित है। तुलनात्मक जीनोमिक्स, और जनसंख्या विश्लेषण ऐसे उदाहरण हैं जो संयोजन के पश्चात् के विश्लेषण में जाते हैं।
विधि परिवर्तनों का प्रभाव
अनुक्रम संयोजन की जटिलता दो प्रमुख कारकों से प्रेरित होती है: टुकड़ों की संख्या और उनकी लंबाई। जबकि अधिक और लंबे टुकड़े अनुक्रम ओवरलैप की उत्तम पहचान की अनुमति देते हैं, वह समस्याएं भी उत्पन्न करते हैं क्योंकि अंतर्निहित एल्गोरिदम टुकड़ों की संख्या और उनकी लंबाई दोनों के लिए द्विघात या यहां तक कि घातीय जटिलता व्यवहार दिखाते हैं। और जबकि छोटे अनुक्रमों को संरेखित करना तेज़ होता है, वह असेंबली के लेआउट चरण को भी जटिल बनाते हैं क्योंकि छोटे रीड्स को दोहराव या समान दोहराव के साथ उपयोग करना अधिक कठिन होता है।
डीएनए अनुक्रमण के प्रारंभिक दिनों में, प्रयोगशालाओं में हफ्तबं के काम के पश्चात् वैज्ञानिक केवल छोटी लंबाई (कुछ दर्जन आधार) के कुछ अनुक्रम ही प्राप्त कर सके। इसलिए, इन अनुक्रमों को हाथ से कुछ ही मिनटों में संरेखित किया जा सकता है।
1975 में, चेन टर्मिनेशन मेथड मेथड (AKA माइक्रोफ्लुइडिक सेंगर सीक्वेंसिंग) का आविष्कार किया गया था और 2000 के कुछ समय पश्चात् तक, विधि को उस बिंदु तक सुधार दिया गया था जहां पूरी तरह से स्वचालित मशीनें 24 घंटे प्रतिदिन अत्यधिक समानांतर मोड में अनुक्रमों का मंथन कर सकती थीं। विश्व भर के बड़े जीनोम केंद्रों में इन अनुक्रमण मशीनों के पूर्ण फार्म स्थित थे, जिसके परिणामस्वरूप संपूर्ण-जीनोम शॉटगन अनुक्रमण परियोजनाओं से अनुक्रमों के लिए असेंबलरों को अनुकूलित करने की आवश्यकता हुई, जहां रीड्स
- लगभग 800-900 आधार लंबे हैं
- अनुक्रमण और क्लोनिंग वैक्टर जैसी अनुक्रमण कलाकृतियाँ सम्मिलित हैं
- त्रुटि दर 0.5 और 10% के मध्य है
सेंगर विधि के साथ, 20,000 से 200,000 रीड वाले बैक्टीरियल प्रोजेक्ट आसानी से कंप्यूटर पर इकट्ठे किए जा सकते हैं। लगभग 35 मिलियन रीड्स वाले मानव जीनोम जैसी बड़ी परियोजनाओं के लिए बड़े कंप्यूटिंग फार्म और वितरित कंप्यूटिंग की आवश्यकता होती है।
2004/2005 तक, 454 लाइफ साइंसेज द्वारा pyrosequencing को व्यावसायिक व्यवहार्यता में लाया गया था। यह नई अनुक्रमण विधि उत्पन्न सेंगर अनुक्रमण की तुलना में बहुत कम पढ़ती है: शुरुआत में लगभग 100 आधार, अब 400-500 आधार। इसकी बहुत अधिक थ्रूपुट और कम व्यय (सेंगर अनुक्रमण की तुलना में) ने जीनोम केंद्रों द्वारा इस विधि को अपनाने को प्रेरित किया, जिसके परिणामस्वरूप अनुक्रम असेंबलरों का विकास हुआ जो रीड समूह को कुशलतापूर्वक संभाल सकते थे। रीड्स में प्रौद्योगिकी-विशिष्ट त्रुटि पैटर्न के साथ जुड़े डेटा की विशाल मात्रा ने असेंबलरों के विकास में देरी की; 2004 की शुरुआत में 454 से केवल नवसिखुआ असेंबलर उपलब्ध था। 2007 के मध्य में रिलीज़ हुई,[8] शेवरेक्स एट अल द्वारा MIRA असेंबलर का हाइब्रिड संस्करण। पहला स्वतंत्र रूप से उपलब्ध असेंबलर था जो 454 रीड्स के साथ-साथ 454 रीड्स और सेंगर रीड्स के मिश्रण को भी असेंबल कर सकता था। विभिन्न अनुक्रमण प्रौद्योगिकियों से अनुक्रमों को इकट्ठा करके पश्चात् में हाइब्रिड जीनोम असेंबली तैयार की गई।
2006 से, इलुमिना (कंपनी) (पहले सोलेक्सा) विधि उपलब्ध है और अनुक्रमण मशीन पर प्रति रन लगभग 100 मिलियन रीड्स उत्पन्न कर सकती है। इसकी तुलना मानव जीनोम परियोजना के 35 मिलियन रीड्स से करें, जिसे सैकड़ों अनुक्रमण मशीनों पर तैयार करने में कई वर्षों की आवश्यकता थी। इलुमिना प्रारंभ में केवल 36 आधारों की लंबाई तक सीमित थी, जिससे यह डे नोवो असेंबली (जैसे कि डे नोवो ट्रांस्क्रिप्टोम असेंबली) के लिए कम उपयुक्त थी, किन्तु प्रौद्योगिकी के नए पुनरावृत्तियों ने 3-400बीपी क्लोन के दोनों सिरों से 100 बेस से ऊपर पढ़ने की लंबाई प्राप्त की। . 2007 के अंत में SHARCGS असेंबलर की घोषणा की गई[9] डोहम एट अल द्वारा। पहला प्रकाशित असेंबलर था जिसका उपयोग सोलेक्सा रीड्स के साथ असेंबली के लिए किया गया था। इसका तुरंत कई अन्य लोगों ने अनुसरण किया।
पश्चात् में, एप्लाइड बायोसिस्टम्स से एबीआई ठोस अनुक्रमण, आयन टोरेंट और एसएमआरटी सीक्वेंसिंग जैसी नई प्रौद्योगिकियां जारी की गईं और नई प्रौद्योगिकियां (जैसे नैनोपोर अनुक्रमण ) उभरती रहीं। इन प्रौद्योगिकियों की उच्च त्रुटि दर के अतिरिक्त वह असेंबली के लिए महत्वपूर्ण हैं क्योंकि उनकी लंबी पढ़ने की लंबाई दोहराव की समस्या का समाधान करने में सहायता करती है। पूर्ण दोहराव के माध्यम से इकट्ठा करना असंभव है जो अधिकतम पढ़ी गई लंबाई से अधिक लंबा है; यद्यपि, जैसे-जैसे पढ़ा जाना लंबा होता जाता है, पूर्ण दोहराव की संभावना उतनी ही बड़ी होती जाती है। इससे लंबे समय एसएमआरटी अनुक्रमण पढ़ने से दोहराव को इकट्ठा करने में लाभ मिलता है, यदि उनकी त्रुटिहीनता कम हो (~ 85%)।
असेंबली एल्गोरिदम
विभिन्न जीवों के जीनोम के अंदर उच्च जटिलता का विशिष्ट क्षेत्र होता है। इसलिए, विभिन्न कम्प्यूटेशनल दृष्टिकोण की आवश्यकता है। सामान्यतः उपयोग किए जाने वाले कुछ एल्गोरिदम हैं:
- ग्राफ असेंबली: कंप्यूटर विज्ञान में ग्राफ सिद्धांत पर आधारित है। डी ब्रुइज़न ग्राफ़ इस दृष्टिकोण का उदाहरण है और रीड्स से सन्निहित को इकट्ठा करने के लिए के-मर्स का उपयोग करता है।
- लालची ग्राफ़ असेंबली: यह दृष्टिकोण असेंबली में प्रत्येक जोड़े गए रीड को स्कोर करता है और ओवरलैपिंग क्षेत्र से उच्चतम संभव स्कोर का चयन करता है।
अनुक्रम खंडों के समूह को देखते हुए, वस्तु को लंबा अनुक्रम ढूंढना है जिसमें सभी टुकड़े सम्मिलित हों (अनुक्रम असेंबली के प्रकार के अनुसार चित्र देखें):
- सभी टुकड़ों के जोड़ीवार संरेखण की गणना करें।
- सबसे बड़े ओवरलैप वाले दो टुकड़े चुनें।
- चुने हुए अंशों को मर्ज करें.
- चरण 2 और 3 को तब तक दोहराएँ जब तक कि केवल टुकड़ा न रह जाए।
परिणाम समस्या का इष्टतम समाधान नहीं हो सकता है।
कार्यक्रम
डे-नोवो असेम्बलर्स की सूची के लिए, डे-नोवो अनुक्रम असेम्बलर्स देखें। मानचित्रण एलाइनर्स की सूची के लिए, List_of_sequence_alignment_software#Short-read_sequence_alignment|अनुक्रम संरेखण सॉफ्टवेयर की सूची § लघु-पढ़ें अनुक्रम संरेखण देखें।
विभिन्न असेंबली चरणों में उपयोग किए जाने वाले कुछ सामान्य उपकरण निम्नलिखित तालिका में सूचीबद्ध हैं:
| सॉफ़्टवेयर | पढ़ें प्रकार | टूल वेब पेज | टिप्पणियाँ |
|---|---|---|---|
| फास्टक्यूसी | विभिन्न | https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ | यह सामान्य उपकरण है जिसका उपयोग इलुमिना, 454 और PacBio जैसी विभिन्न अनुक्रमण प्रौद्योगिकियों से रीड्स की गुणवत्ता की जांच करने के लिए किया जाता है। |
| बीडब्ल्यूए | लघु एवं दीर्घ पाठ्य | https://sourceforge.net/projects/bio-bwa/files/ | यह कमांड लाइन टूल है. अधिकतर हल्के रन और त्रुटिहीन अनुक्रम संरेखण के लिए जाना जाता है। |
| मिनीमैप2 | लंबे समय तक पढ़ता है | https://github.com/lh3/minimap2 | यह कमांड लाइन टूल PacBio और ऑक्सफोर्ड नैनोपोर को संभालने के लिए डिज़ाइन किया गया है और 15% त्रुटि दर के साथ पढ़ता है। |
| LoReTTA | लंबे समय तक पढ़ता है | https://github.com/salvocamiolo/LoReTTA/releases/tag/v0.1 | यह उपकरण PacBio CCS रीड्स का उपयोग करके अधिक त्रुटिहीनता से वायरल जीनोम को इकट्ठा करने (संदर्भ-निर्देशित) के लिए डिज़ाइन किया गया है। |
| SPAdes | लघु एवं दीर्घ पाठ्य | http://cab.spbu.ru/software/spades/ | यह असेंबली टूल है जो कमांड लाइन पर चलता है। |
| Samtools | संरेखण विश्लेषण | https://samtools.github.io | यह असेंबली के पश्चात् उपयोगी है. यह विभिन्न आँकड़े उत्पन्न कर सकता है और संरेखण फ़ाइल में कई फ़िल्टरिंग चरण निष्पादित कर सकता है। |
यह भी देखें
- डे नोवो अनुक्रम असेंबलर
- अनुक्रम संरेखण
- डे नोवो ट्रांस्क्रिप्टोम असेंबली
- समूह कवर समस्या
- अनुक्रमित पशु जीनोम की सूची
संदर्भ
- ↑ Myers, E. W.; Sutton, GG; Delcher, AL; Dew, IM; Fasulo, DP; Flanigan, MJ; Kravitz, SA; Mobarry, CM; et al. (March 2000). "ड्रोसोफिला की एक संपूर्ण-जीनोम असेंबली". Science. 287 (5461): 2196–204. Bibcode:2000Sci...287.2196M. CiteSeerX 10.1.1.79.9822. doi:10.1126/science.287.5461.2196. PMID 10731133. S2CID 6049420.
- ↑ Batzoglou, S.; Jaffe, DB; Stanley, K; Butler, J; Gnerre, S; Mauceli, E; Berger, B; Mesirov, JP; Lander, ES (January 2002). "ARACHNE: a whole-genome shotgun assembler". Genome Research. 12 (1): 177–89. doi:10.1101/gr.208902. PMC 155255. PMID 11779843.
- ↑ "हमें विकी पसंद है". amos.sourceforge.net. Retrieved 2023-01-02.
- ↑ Miller, Jason R.; Koren, Sergey; Sutton, Granger (2010-03-06). "अगली पीढ़ी के अनुक्रमण डेटा के लिए असेंबली एल्गोरिदम". Genomics (in English). 95 (6): 315–327. doi:10.1016/j.ygeno.2010.03.001. PMC 2874646. PMID 20211242.
- ↑ Wolf, Beat. "डे नोवो जीनोम असेंबली बनाम एक संदर्भ जीनोम की मैपिंग" (PDF). University of Applied Sciences Western Switzerland. Retrieved 6 April 2019.
- ↑ "बब्राहम जैव सूचना विज्ञान - फास्टक्यूसी उच्च थ्रूपुट अनुक्रम डेटा के लिए एक गुणवत्ता नियंत्रण उपकरण". www.bioinformatics.babraham.ac.uk. Retrieved 2022-05-09.
- ↑ Ruffalo, M.; LaFramboise, T.; Koyuturk, M. (2011-10-15). "अगली पीढ़ी के अनुक्रमण रीड अलाइनमेंट के लिए एल्गोरिदम का तुलनात्मक विश्लेषण". Bioinformatics (in English). 27 (20): 2790–2796. doi:10.1093/bioinformatics/btr477. ISSN 1367-4803. PMID 21856737.
- ↑ "MIRA 2.9.8 for 454 and 454 / Sanger hybrid assembly". groups.google.com. Retrieved 2023-01-02.
- ↑ Dohm, J. C.; Lottaz, C.; Borodina, T.; Himmelbauer, H. (November 2007). "SHARCGS, डे नोवो जीनोमिक अनुक्रमण के लिए एक तेज़ और अत्यधिक सटीक शॉर्ट-रीड असेंबली एल्गोरिदम". Genome Research. 17 (11): 1697–706. doi:10.1101/gr.6435207. PMC 2045152. PMID 17908823.