साइटोक्रोम सी ऑक्सीडेज
| साइटोक्रोम सी ऑक्सीडेज | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| Identifiers | |||||||||
| EC no. | 1.9.3.1 | ||||||||
| CAS no. | 9001-16-5 | ||||||||
| Databases | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| PDB structures | RCSB PDB PDBe PDBsum | ||||||||
| Gene Ontology | AmiGO / QuickGO | ||||||||
| |||||||||
| Cytochrome c oxidase | |
|---|---|
 | |
| Identifiers | |
| Symbol | Cytochrome c oxidase |
| OPM superfamily | 4 |
| OPM protein | 2dyr |
| Membranome | 257 |
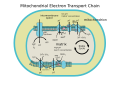
एंजाइम साइटोक्रोम सी ऑक्सीडेज या कॉम्प्लेक्स IV, ( EC 1.9.3.1 था जिसे अब एक ट्रांसलोकेस EC 7.1.1.9 के रूप में पुनर्वर्गीकृत) एक बड़ी ट्रांसमेम्ब्रेन प्रोटीन है जो यूकैर्योसाइटों के जीवाणु, आर्किया और माइटोकॉन्ड्रिया में जटिल पाया जाता है।[1]
यह झिल्ली में स्थित कोशिकाओं की श्वसन इलेक्ट्रॉन परिवहन श्रृंखला का अंतिम एंजाइम है। यह चार साइटोक्रोम सी अणुओं में से प्रत्येक से एक इलेक्ट्रॉन प्राप्त करता है और उन्हें एक ऑक्सीजन अणु और चार प्रोटॉन में स्थानांतरित करता है, जिससे पानी के दो अणु बनते हैं। आंतरिक जलीय चरण से चार प्रोटॉन को बांधने के अलावा, यह झिल्ली के पार एक प्रोटॉन को स्थानांतरित करता है, प्रोटॉन इलेक्ट्रोकेमिकल क्षमता के ट्रांसमेम्ब्रेन अंतर को बढ़ाता है, जिसे एटीपी सिंथेज़ तब एटीपी को संश्लेषित करने के लिए उपयोग करता है।
संरचना
परिसर
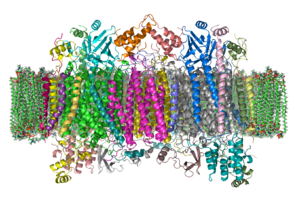
कॉम्प्लेक्स एक बड़ा अभिन्न झिल्ली प्रोटीन है जो कई कॉफ़ेक्टर (बायोकेमिस्ट्री) # मेटल आयनों और 14 से बना है [2] स्तनधारियों में प्रोटीन सबयूनिट्स। स्तनधारियों में, ग्यारह सबयूनिट मूल रूप से परमाणु होते हैं, और तीन माइटोकॉन्ड्रिया में संश्लेषित होते हैं। कॉम्प्लेक्स में दो वो मुझे होते हैं, एक साइटोक्रोम ए और साइटोक्रोम ए | साइटोक्रोम ए3, और दो तांबे के केंद्र, CuA और क्यूB केंद्र।[3] वास्तव में, साइटोक्रोम ए3 और क्यूB एक द्विनाभिक केंद्र बनाते हैं जो ऑक्सीजन की कमी का स्थल है। साइटोक्रोम सी, जो श्वसन श्रृंखला (साइटोक्रोम बीसी1 कॉम्प्लेक्स, कॉम्प्लेक्स III) के पूर्ववर्ती घटक द्वारा कम किया जाता है, क्यू के पास डॉक करता हैA बाइन्यूक्लियर सेंटर और इसमें एक इलेक्ट्रॉन पास करता है, Fe युक्त साइटोक्रोम c में वापस ऑक्सीकृत हो जाता है3+. घटा हुआ घनA बाइन्यूक्लियर सेंटर अब एक इलेक्ट्रॉन को साइटोक्रोम ए पर भेजता है, जो बदले में एक इलेक्ट्रॉन को साइटोक्रोम ए पर भेजता है3>-साथB द्विपरमाणु केंद्र। इस द्विनाभिक केंद्र में दो धातु आयन 4.5 Å अलग हैं और पूरी तरह से ऑक्सीकृत अवस्था में एक हाइड्रोक्साइड आयन का समन्वय करते हैं।
साइटोक्रोम सी ऑक्सीडेज की एक्स - रे क्रिस्टलोग्राफी एक असामान्य पोस्ट-ट्रांसलेशनल मॉडिफिकेशन दिखाती है, जो टीयर (244) के सी6 और हिस (240) के ε-एन (गोजातीय एंजाइम नंबरिंग) को जोड़ती है। यह साइटोक्रोम ए को सक्षम करने में महत्वपूर्ण भूमिका निभाता है3- साथB आणविक ऑक्सीजन को कम करने में चार इलेक्ट्रॉनों और पानी में चार प्रोटॉन को स्वीकार करने के लिए द्विनाभिकीय केंद्र। कमी के तंत्र को पूर्व में एक पेरोक्साइड मध्यवर्ती शामिल करने के लिए सोचा गया था, जिसके बारे में माना जाता था कि इससे सुपरऑक्साइड का उत्पादन होता है। हालांकि, वर्तमान में स्वीकृत तंत्र में तेजी से चार-इलेक्ट्रॉन कमी शामिल है, जिसमें तत्काल ऑक्सीजन-ऑक्सीजन बंधन दरार शामिल है, जिससे सुपरऑक्साइड बनाने की किसी भी मध्यवर्ती संभावना से बचा जा सकता है।[4]: 865–866
संरक्षित सबयूनिट्स
| क्रम सं | सबयूनिट नाम | मानव प्रोटीन | यूनीप्रोट से प्रोटीन विवरण | मानव प्रोटीन के साथ Pfam परिवार |
|---|---|---|---|---|
| 1 | Cox1 | COX1_HUMAN | साइटोक्रोम सी ऑक्सीडेज सबयूनिट 1 | Pfam PF00115 |
| 2 | Cox2 | COX2_HUMAN | साइटोक्रोम सी ऑक्सीडेज सबयूनिट 2 | Pfam PF02790, Pfam PF00116 |
| 3 | Cox3 | COX3_HUMAN | साइटोक्रोम सी ऑक्सीडेज सबयूनिट 3 | Pfam PF00510 |
| 4 | Cox4i1 | COX41_HUMAN | साइटोक्रोम सी ऑक्सीडेज सबयूनिट 4 आइसोफॉर्म 1, माइटोकॉन्ड्रियल | Pfam PF02936 |
| 5 | Cox4a2 | COX42_HUMAN | Cytochrome c oxidase subunit 4 isoform 2, mitochondrial | Pfam PF02936 |
| 6 | Cox5a | COX5A_HUMAN | Cytochrome c oxidase subunit 5A, mitochondrial | Pfam PF02284 |
| 7 | Cox5b | COX5B_HUMAN | Cytochrome c oxidase subunit 5B, mitochondrial | Pfam PF01215 |
| 8 | Cox6a1 | CX6A1_HUMAN | Cytochrome c oxidase subunit 6A1, mitochondrial | Pfam PF02046 |
| 9 | Cox6a2 | CX6A2_HUMAN | Cytochrome c oxidase subunit 6A2, mitochondrial | Pfam PF02046 |
| 10 | Cox6b1 | CX6B1_HUMAN | Cytochrome c oxidase subunit 6B1 | Pfam PF02297 |
| 11 | Cox6b2 | CX6B2_HUMAN | Cytochrome c oxidase subunit 6B2 | Pfam PF02297 |
| 12 | Cox6c | COX6C_HUMAN | Cytochrome c oxidase subunit 6C | Pfam PF02937 |
| 13 | Cox7a1 | CX7A1_HUMAN | Cytochrome c oxidase subunit 7A1, mitochondrial | Pfam PF02238 |
| 14 | Cox7a2 | CX7A2_HUMAN | Cytochrome c oxidase subunit 7A2, mitochondrial | Pfam PF02238 |
| 15 | Cox7a3 | COX7S_HUMAN | Putative cytochrome c oxidase subunit 7A3, mitochondrial | Pfam PF02238 |
| 16 | Cox7b | COX7B_HUMAN | Cytochrome c oxidase subunit 7B, mitochondrial | Pfam PF05392 |
| 17 | Cox7c | COX7C_HUMAN | Cytochrome c oxidase subunit 7C, mitochondrial | Pfam PF02935 |
| 18 | Cox7r | COX7R_HUMAN | Cytochrome c oxidase subunit 7A-related protein, mitochondrial | Pfam PF02238 |
| 19 | Cox8a | COX8A_HUMAN | Cytochrome c oxidase subunit 8A, mitochondrial P | Pfam PF02285 |
| 20 | Cox8c | COX8C_HUMAN | Cytochrome c oxidase subunit 8C, mitochondrial | Pfam PF02285 |
| Assembly subunits[7][8][9] | ||||
| 1 | Coa1 | COA1_HUMAN | Cytochrome c oxidase assembly factor 1 homolog | Pfam PF08695 |
| 2 | Coa3 | COA3_HUMAN | Cytochrome c oxidase assembly factor 3 homolog, mitochondrial | Pfam PF09813 |
| 3 | Coa4 | COA4_HUMAN | Cytochrome c oxidase assembly factor 4 homolog, mitochondrial | Pfam PF06747 |
| 4 | Coa5 | COA5_HUMAN | Cytochrome c oxidase assembly factor 5 | Pfam PF10203 |
| 5 | Coa6 | COA6_HUMAN | Cytochrome c oxidase assembly factor 6 homolog | Pfam PF02297 |
| 6 | Coa7 | COA7_HUMAN | Cytochrome c oxidase assembly factor 7, | Pfam PF08238 |
| 7 | Cox11 | COX11_HUMAN | Cytochrome c oxidase assembly protein COX11 mitochondrial | Pfam PF04442 |
| 8 | Cox14 | COX14_HUMAN | Cytochrome c oxidase assembly protein | Pfam PF14880 |
| 9 | Cox15 | COX15_HUMAN | Cytochrome c oxidase assembly protein COX15 homolog | Pfam PF02628 |
| 10 | Cox16 | COX16_HUMAN | Cytochrome c oxidase assembly protein COX16 homolog mitochondrial | Pfam PF14138 |
| 11 | Cox17 | COX17_HUMAN | Cytochrome c oxidase copper chaperone | Pfam PF05051 |
| 12 | Cox18[10] | COX18_HUMAN | Mitochondrial inner membrane protein (Cytochrome c oxidase assembly protein 18) | Pfam PF02096 |
| 13 | Cox19 | COX19_HUMAN | Cytochrome c oxidase assembly protein | Pfam PF06747 |
| 14 | Cox20 | COX20_HUMAN | Cytochrome c oxidase protein 20 homolog | Pfam PF12597 |
विधानसभा
एस. सेरेविसिया में COX असेंबली एक जटिल प्रक्रिया है जिसे हाइड्रोफोबिक सबयूनिट्स के तीव्र और अपरिवर्तनीय एकत्रीकरण के कारण पूरी तरह से समझा नहीं जा सका है जो होलोनीजाइम कॉम्प्लेक्स बनाते हैं, साथ ही उजागर हाइड्रोफोबिक पैच के साथ म्यूटेंट सबयूनिट्स का एकत्रीकरण।[11]COX उपइकाइयां परमाणु और माइटोकॉन्ड्रियल जीनोम दोनों में एन्कोडेड हैं। COX कैटेलिटिक कोर बनाने वाली तीन सबयूनिट माइटोकॉन्ड्रियल जीनोम में एन्कोडेड हैं।
हेम्स और कॉफ़ेक्टर्स को सब यूनिट I और II में डाला जाता है। दो हीम अणु सबयूनिट I में रहते हैं, सबयूनिट II में परिवहन में मदद करते हैं जहां दो तांबे के अणु इलेक्ट्रॉनों के निरंतर हस्तांतरण में सहायता करते हैं।[12] सबयूनिट I और IV असेंबली आरंभ करते हैं। अलग-अलग सबयूनिट्स सब-कॉम्प्लेक्स इंटरमीडिएट बनाने के लिए संबद्ध हो सकते हैं जो बाद में COX कॉम्प्लेक्स बनाने के लिए अन्य सबयूनिट्स से जुड़ जाते हैं।[11]असेंबली के बाद के संशोधनों में, COX एक होमोडीमर बनाएगा। यह गतिविधि के लिए आवश्यक है। डिमर्स एक कार्डियोलिपिन अणु से जुड़े होते हैं,[11][13][14] जो होलोनीजाइम कॉम्प्लेक्स के स्थिरीकरण में महत्वपूर्ण भूमिका निभाते पाए गए हैं। सबयूनिट्स VIIa और III के पृथक्करण के साथ-साथ कार्डियोलिपिन को हटाने से एंजाइम गतिविधि का कुल नुकसान होता है।[14]परमाणु जीनोम में एन्कोडेड सबयूनिट्स को एंजाइम डिमराइजेशन और स्थिरता में भूमिका निभाने के लिए जाना जाता है। इन सबयूनिट्स के उत्परिवर्तन COX फ़ंक्शन को समाप्त कर देते हैं।[11]
असेंबली को कम से कम तीन अलग-अलग दर-निर्धारण चरणों में जाना जाता है। इन चरणों के उत्पाद पाए गए हैं, हालांकि विशिष्ट सबयूनिट संयोजन निर्धारित नहीं किए गए हैं।[11]
COX सबयूनिट्स I, II, और III के संश्लेषण और असेंबली को ट्रांसलेशनल एक्टिविस्ट्स द्वारा सुगम बनाया जाता है, जो माइटोकॉन्ड्रियल mRNA ट्रांसक्रिप्ट के 5' अनट्रांसलेटेड क्षेत्रों के साथ इंटरैक्ट करते हैं। ट्रांसलेशनल एक्टिवेटर्स न्यूक्लियस में एन्कोडेड हैं। वे अनुवाद मशीनरी के अन्य घटकों के साथ प्रत्यक्ष या अप्रत्यक्ष बातचीत के माध्यम से काम कर सकते हैं, लेकिन इन-विट्रो में अनुवाद मशीनरी को संश्लेषित करने से जुड़ी कठिनाइयों के कारण सटीक आणविक तंत्र स्पष्ट नहीं हैं।[15][16] हालांकि माइटोकॉन्ड्रियल जीनोम के भीतर एन्कोड किए गए सबयूनिट I, II और III के बीच की बातचीत, बिगेनोमिक सबयूनिट्स के बीच की बातचीत की तुलना में एंजाइम स्थिरता में कम योगदान देती है, ये सबयूनिट्स अधिक संरक्षित हैं, जो एंजाइम गतिविधि के लिए संभावित अस्पष्टीकृत भूमिकाओं का संकेत देते हैं।[17]
जैव रसायन
This section is missing information about names of the six traditional intermediate states (APFOER); 2021 Cyro-EM result proposing an RPFOE mechanism with reversed assignment of red-ox phases (doi:10.1038/s41467-021-27174-y |
समग्र प्रतिक्रिया है
- 4 फे2+ - साइटोक्रोम सी + 4 एच+ + ओ2 → 4 फ़े3+ - साइटोक्रोम सी + 2 एच2डीfGo' = - 218 केजे/मोल
Cu के माध्यम से दो साइटोक्रोम c's से दो इलेक्ट्रॉन पारित किए जाते हैंA और साइटोक्रोम ए साइटों को साइटोक्रोम ए3-साथB द्विनाभिकीय केंद्र, धातुओं को Fe तक कम करता है2+ फार्म और क्यू+. हाइड्रॉक्साइड लिगैंड प्रोटोनेट होता है और पानी के रूप में खो जाता है, धातुओं के बीच एक शून्य बनाता है जो ओ द्वारा भरा जाता है2. Fe से आने वाले दो इलेक्ट्रॉनों के साथ, ऑक्सीजन तेजी से कम हो जाती है2+-साइटोक्रोम ए3, जिसे फेरिल ऑक्सो फॉर्म (Fe4+= ओ)। क्यू के करीब ऑक्सीजन परमाणुB Cu से एक इलेक्ट्रॉन लेता है+, और एक दूसरा इलेक्ट्रॉन और टायर (244) के हाइड्रॉकसिल से एक प्रोटॉन, जो टायरोसिल रेडिकल बन जाता है। दूसरा ऑक्सीजन दो इलेक्ट्रॉनों और एक प्रोटॉन को लेकर एक हाइड्रॉक्साइड आयन में परिवर्तित हो जाता है। दूसरे साइटोक्रोम c से एक तीसरा इलेक्ट्रॉन पहले दो इलेक्ट्रॉन वाहकों के माध्यम से साइटोक्रोम a में जाता है3-साथB बाइन्यूक्लियर सेंटर, और यह इलेक्ट्रॉन और दो प्रोटॉन टायरोसिल रेडिकल को वापस टायर में बदल देते हैं, और हाइड्रॉक्साइड Cu से बंध जाता हैB2+ एक पानी के अणु को। दूसरे साइटोक्रोम c से चौथा इलेक्ट्रॉन Cu से होकर बहता हैA और साइटोक्रोम ए से साइटोक्रोम ए3-साथB द्विनाभिक केंद्र, Fe को कम करना4+= फ़े3+, ऑक्सीजन परमाणु एक साथ एक प्रोटॉन उठा रहा है, इस ऑक्सीजन को हाइड्रॉक्साइड आयन के रूप में पुन: उत्पन्न कर रहा है जो साइटोक्रोम ए के मध्य में समन्वित है3-साथB केंद्र के रूप में यह इस चक्र की शुरुआत में था। कुल मिलाकर, चार कम साइटोक्रोम सी ऑक्सीकृत होते हैं जबकि ओ2 और चार प्रोटॉन दो पानी के अणुओं में कम हो जाते हैं।Cite error: Closing </ref> missing for <ref> tag
साइनाइड, अब्द और कार्बन मोनोआक्साइड[18] सभी साइटोक्रोम सी ऑक्सीडेज से बंधते हैं, प्रोटीन को कार्य करने से रोकते हैं और कोशिकाओं के रासायनिक श्वासावरोध की ओर ले जाते हैं। अवरोधक सांद्रता में वृद्धि के लिए क्षतिपूर्ति करने के लिए आणविक ऑक्सीजन की उच्च सांद्रता की आवश्यकता होती है, जिससे अवरोधक की उपस्थिति में सेल में चयापचय गतिविधि में समग्र कमी आती है। अन्य लिगेंड, जैसे नाइट्रिक ऑक्साइड और हाइड्रोजन सल्फाइड, एंजाइम पर नियामक साइटों को बाध्य करके सीओएक्स को रोक सकते हैं, सेलुलर श्वसन की दर को कम कर सकते हैं।[19]
साइनाइड COX के लिए एक गैर-प्रतिस्पर्धी अवरोधक है,[20][21] एंजाइम की आंशिक रूप से कम अवस्था के लिए उच्च आत्मीयता के साथ बंधन और एंजाइम की और कमी में बाधा। स्पंदित अवस्था में साइनाइड धीरे-धीरे बंधता है, लेकिन उच्च आत्मीयता के साथ। लिगैंड को इलेक्ट्रोस्टैटिक रूप से दोनों धातुओं को एक ही बार में उनके बीच स्थित करके स्थिर करने के लिए प्रस्तुत किया जाता है। एक उच्च नाइट्रिक ऑक्साइड सांद्रता, जैसे कि एंजाइम में बहिर्जात रूप से जोड़ा गया, COX के साइनाइड निषेध को उलट देता है।[22]
नाइट्रिक ऑक्साइड उलटा हो सकता है[23] नाइट्राइट में ऑक्सीकृत होने के लिए द्विनाभिकीय केंद्र में किसी भी धातु आयन से बांधें। सं और सीएन− कोशिकीय श्वसन की दर को कम करते हुए साइट पर बाँधने के लिए ऑक्सीजन के साथ प्रतिस्पर्धा करेगा। अंतर्जात NO, हालांकि, जो निचले स्तर पर उत्पादित होता है, CN को बढ़ाता है− निषेध। NO का उच्च स्तर, जो कम अवस्था में अधिक एंजाइम के अस्तित्व के साथ संबंध रखता है, साइनाइड के अधिक निषेध का कारण बनता है।[24]इन बेसल सांद्रता पर, कॉम्प्लेक्स IV के सं अवरोध को लाभकारी प्रभाव के लिए जाना जाता है, जैसे कि रक्त वाहिका के ऊतकों में ऑक्सीजन का स्तर बढ़ाना। पानी में ऑक्सीजन को कम करने के लिए एंजाइम की अक्षमता के परिणामस्वरूप ऑक्सीजन का निर्माण होता है, जो आसपास के ऊतकों में गहराई तक फैल सकता है।[23]कॉम्प्लेक्स IV के NO निषेध का कम ऑक्सीजन सांद्रता पर बड़ा प्रभाव पड़ता है, जिससे ज़रूरत के ऊतकों में वैसोडिलेटर के रूप में इसकी उपयोगिता बढ़ जाती है।[23]
हाइड्रोजन सल्फाइड कार्बन मोनोऑक्साइड के समान एंजाइम पर एक नियामक साइट पर एक गैर-प्रतिस्पर्धी फैशन में सीओएक्स को बांध देगा। सल्फाइड में एंजाइम के स्पंदित या आंशिक रूप से कम होने वाले राज्यों के लिए उच्चतम संबंध है, और हीम ए पर एंजाइम को आंशिक रूप से कम करने में सक्षम है3 केंद्र। यह स्पष्ट नहीं है कि अंतर्जात एच2एस स्तर एंजाइम को बाधित करने के लिए पर्याप्त हैं। हाइड्रोजन सल्फाइड और सीओएक्स की पूरी तरह से कम संरचना के बीच कोई बातचीत नहीं है।[19]
विकृत अल्कोहल में मेथनॉल चींटी का तेजाब में परिवर्तित हो जाता है, जो उसी ऑक्सीडेज सिस्टम को भी रोकता है। एटीपी के उच्च स्तर माइटोकॉन्ड्रियल मैट्रिक्स के भीतर से बाध्यकारी, साइटोक्रोम सी ऑक्सीडेज को बाधित कर सकते हैं।[25]
एक्स्ट्रामाइटोकोंड्रियल और उपकोशिकीय स्थानीयकरण

साइटोक्रोम सी ऑक्सीडेज में 3 सबयूनिट्स हैं जो माइटोकॉन्ड्रियल डीएनए (साइटोक्रोम सी ऑक्सीडेज साइटोक्रोम सी ऑक्सीडेज सबयूनिट आई, साइटोक्रोम सी ऑक्सीडेज सबयूनिट II, और साइटोक्रोम सी ऑक्सीडेज सबयूनिट III) द्वारा एन्कोडेड हैं। माइटोकॉन्ड्रियल डीएनए द्वारा एन्कोड किए गए इन 3 सबयूनिट्स में से दो की पहचान एक्स्ट्रामाइटोकॉन्ड्रियल स्थानों में की गई है। अग्नाशयी संगोष्ठी ऊतक में, ये सबयूनिट्स zymogen कणिकाओं में पाए गए। इसके अतिरिक्त, पूर्वकाल पिट्यूटरी में, इन सबयूनिट्स की अपेक्षाकृत उच्च मात्रा वृद्धि हार्मोन स्रावी कणिकाओं में पाई गई।[26] इन साइटोक्रोम सी ऑक्सीडेज सबयूनिट्स के एक्स्ट्रामाइटोकॉन्ड्रियल फ़ंक्शन को अभी तक विशेषता नहीं दी गई है। साइटोक्रोम सी ऑक्सीडेज सबयूनिट्स के अलावा, अन्य माइटोकॉन्ड्रियल प्रोटीनों की बड़ी संख्या के लिए एक्स्ट्रामाइटोकॉन्ड्रियल स्थानीयकरण भी देखा गया है।[27][28] यह माइटोकॉन्ड्रिया से अन्य सेलुलर गंतव्यों तक प्रोटीन स्थानांतरण के लिए अभी तक अज्ञात विशिष्ट तंत्र के अस्तित्व की संभावना को बढ़ाता है।[26][28][29]
आनुवंशिक दोष और विकार
साइटोक्रोम सी ऑक्सीडेज (सीओएक्स) की कार्यक्षमता या संरचना को बदलने वाले आनुवंशिक उत्परिवर्तनों से जुड़े दोषों के परिणामस्वरूप गंभीर, अक्सर घातक चयापचय संबंधी विकार हो सकते हैं। इस तरह के विकार आमतौर पर बचपन में प्रकट होते हैं और मुख्य रूप से उच्च ऊर्जा की मांग वाले ऊतकों (मस्तिष्क, हृदय, मांसपेशियों) को प्रभावित करते हैं। कई वर्गीकृत माइटोकॉन्ड्रियल बीमारियों में से, निष्क्रिय सीओएक्स असेंबली को शामिल करने वालों को सबसे गंभीर माना जाता है।[30] सीओएक्स विकारों के विशाल बहुमत परमाणु-एन्कोडेड प्रोटीन में उत्परिवर्तन से जुड़े होते हैं जिन्हें असेंबली कारक या असेंबली प्रोटीन कहा जाता है। ये असेंबली कारक COX संरचना और कार्यक्षमता में योगदान करते हैं, और कई आवश्यक प्रक्रियाओं में शामिल होते हैं, जिनमें माइटोकॉन्ड्रियन-एन्कोडेड सबयूनिट्स का ट्रांसक्रिप्शन और अनुवाद, प्रीप्रोटीन का प्रसंस्करण और झिल्ली सम्मिलन, और कॉफ़ेक्टर बायोसिंथेसिस और निगमन शामिल हैं।[31] वर्तमान में, सात COX असेंबली कारकों में उत्परिवर्तन की पहचान की गई है: SURF1, SCO1, SCO2, COX10, COX15, COX20, COA5 और LRPPRC। इन प्रोटीनों में उत्परिवर्तन के परिणामस्वरूप सब-कॉम्प्लेक्स असेंबली, कॉपर ट्रांसपोर्ट या ट्रांसलेशनल रेगुलेशन की कार्यक्षमता बदल सकती है। प्रत्येक जीन उत्परिवर्तन एक विशिष्ट बीमारी के एटियलजि से जुड़ा होता है, जिसमें कुछ का कई विकारों में प्रभाव होता है। जीन म्यूटेशन के माध्यम से डिसफंक्शनल COX असेंबली से जुड़े विकारों में लेह सिंड्रोम, कार्डियोमायोपैथी, ल्यूकोडिस्ट्रॉफी, रक्ताल्पता और सेंसरिनुरल बहरापन शामिल हैं।
हिस्टोकेमिस्ट्री
ऊर्जा के लिए ऑक्सीडेटिव फास्फारिलीकरण पर न्यूरॉन्स की बढ़ती निर्भरता[32] जानवरों में क्षेत्रीय मस्तिष्क चयापचय की मैपिंग में COX हिस्टोकेमिस्ट्री के उपयोग की सुविधा देता है, क्योंकि यह एंजाइम गतिविधि और न्यूरोनल गतिविधि के बीच प्रत्यक्ष और सकारात्मक संबंध स्थापित करता है।[33] यह COX एंजाइम राशि और गतिविधि के बीच संबंध में देखा जा सकता है, जो जीन अभिव्यक्ति के स्तर पर COX के नियमन को इंगित करता है। COX वितरण पशु मस्तिष्क के विभिन्न क्षेत्रों में असंगत है, लेकिन इसके वितरण का पैटर्न जानवरों के अनुरूप है। यह पैटर्न बंदर, चूहे और बछड़े के मस्तिष्क में देखा गया है। मस्तिष्क के हिस्टोकेमिकल विश्लेषण में COX के एक आइसोजाइम का लगातार पता लगाया गया है।[34] इस तरह के ब्रेन मैपिंग को अनुमस्तिष्क रोग जैसे रीलर के साथ सहज उत्परिवर्ती चूहों में पूरा किया गया है[35] और अल्जाइमर रोग का एक ट्रांसजेनिक मॉडल।[36] इस तकनीक का उपयोग पशु मस्तिष्क में सीखने की गतिविधि को मैप करने के लिए भी किया गया है।[37]
अतिरिक्त छवियां
यह भी देखें
- साइटोक्रोम सी ऑक्सीडेज सबयूनिट आई
- साइटोक्रोम सी ऑक्सीडेज सबयूनिट II
- साइटोक्रोम सी ऑक्सीडेज सबयूनिट III
- हेम ए
संदर्भ
- ↑ Castresana J, Lübben M, Saraste M, Higgins DG (June 1994). "Evolution of cytochrome oxidase, an enzyme older than atmospheric oxygen". The EMBO Journal (in English). 13 (11): 2516–2525. doi:10.1002/j.1460-2075.1994.tb06541.x. PMC 395125. PMID 8013452.
- ↑ Balsa E, Marco R, Perales-Clemente E, Szklarczyk R, Calvo E, Landázuri MO, Enríquez JA (September 2012). "NDUFA4 is a subunit of complex IV of the mammalian electron transport chain". Cell Metabolism. 16 (3): 378–86. doi:10.1016/j.cmet.2012.07.015. PMID 22902835.
- ↑ Tsukihara T, Aoyama H, Yamashita E, Tomizaki T, Yamaguchi H, Shinzawa-Itoh K, Nakashima R, Yaono R, Yoshikawa S (August 1995). "Structures of metal sites of oxidized bovine heart cytochrome c oxidase at 2.8 A". Science. 269 (5227): 1069–74. Bibcode:1995Sci...269.1069T. doi:10.1126/science.7652554. PMID 7652554. S2CID 27210776.
- ↑ Cite error: Invalid
<ref>tag; no text was provided for refs namedVoest_2011 - ↑ Zhang Z, Huang L, Shulmeister VM, Chi YI, Kim KK, Hung LW, Crofts AR, Berry EA, Kim SH (April 1998). "Electron transfer by domain movement in cytochrome bc1". Nature. 392 (6677): 677–84. Bibcode:1998Natur.392..677Z. doi:10.1038/33612. PMID 9565029. S2CID 4380033.
- ↑ Kaila VR, Oksanen E, Goldman A, Bloch DA, Verkhovsky MI, Sundholm D, Wikström M (July 2011). "A combined quantum chemical and crystallographic study on the oxidized binuclear center of cytochrome c oxidase". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1807 (7): 769–78. doi:10.1016/j.bbabio.2010.12.016. PMID 21211513.
- ↑ Szklarczyk R, Wanschers BF, Cuypers TD, Esseling JJ, Riemersma M, van den Brand MA, Gloerich J, Lasonder E, van den Heuvel LP, Nijtmans LG, Huynen MA (February 2012). "Iterative orthology prediction uncovers new mitochondrial proteins and identifies C12orf62 as the human ortholog of COX14, a protein involved in the assembly of cytochrome c oxidase". Genome Biology. 13 (2): R12. doi:10.1186/gb-2012-13-2-r12. PMC 3334569. PMID 22356826.
- ↑ Mick DU, Dennerlein S, Wiese H, Reinhold R, Pacheu-Grau D, Lorenzi I, Sasarman F, Weraarpachai W, Shoubridge EA, Warscheid B, Rehling P (December 2012). "MITRAC links mitochondrial protein translocation to respiratory-chain assembly and translational regulation". Cell. 151 (7): 1528–41. doi:10.1016/j.cell.2012.11.053. PMID 23260140.
- ↑ Kozjak-Pavlovic V, Prell F, Thiede B, Götz M, Wosiek D, Ott C, Rudel T (February 2014). "C1orf163/RESA1 is a novel mitochondrial intermembrane space protein connected to respiratory chain assembly". Journal of Molecular Biology. 426 (4): 908–20. doi:10.1016/j.jmb.2013.12.001. PMID 24333015.
- ↑ Gaisne M, Bonnefoy N (September 2006). "The COX18 gene, involved in mitochondrial biogenesis, is functionally conserved and tightly regulated in humans and fission yeast". FEMS Yeast Research. 6 (6): 869–82. doi:10.1111/j.1567-1364.2006.00083.x. PMID 16911509.
- ↑ 11.0 11.1 11.2 11.3 11.4 Fontanesi F, Soto IC, Horn D, Barrientos A (December 2006). "Assembly of mitochondrial cytochrome c-oxidase, a complicated and highly regulated cellular process". American Journal of Physiology. Cell Physiology. 291 (6): C1129-47. doi:10.1152/ajpcell.00233.2006. PMID 16760263.
- ↑ Crofts A (1996). "Cytochrome oxidase: Complex IV". University of Illinois at Urbana-Champaign. Archived from the original on 2018-01-23. Retrieved 2018-01-28.
- ↑ Khalimonchuk O, Rödel G (December 2005). "Biogenesis of cytochrome c oxidase". Mitochondrion. 5 (6): 363–88. doi:10.1016/j.mito.2005.08.002. PMID 16199211.
- ↑ 14.0 14.1 Sedlák E, Robinson NC (September 2015). "Destabilization of the Quaternary Structure of Bovine Heart Cytochrome c Oxidase upon Removal of Tightly Bound Cardiolipin". Biochemistry. 54 (36): 5569–77. doi:10.1021/acs.biochem.5b00540. PMID 26284624.
- ↑ Herrmann JM, Woellhaf MW, Bonnefoy N (February 2013). "Control of protein synthesis in yeast mitochondria: the concept of translational activators". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1833 (2): 286–94. doi:10.1016/j.bbamcr.2012.03.007. PMID 22450032.
- ↑ Soto IC, Fontanesi F, Liu J, Barrientos A (June 2012). "Biogenesis and assembly of eukaryotic cytochrome c oxidase catalytic core". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1817 (6): 883–97. doi:10.1016/j.bbabio.2011.09.005. PMC 3262112. PMID 21958598.
- ↑ Aledo JC, Valverde H, Ruíz-Camacho M, Morilla I, López FD (October 2014). "Protein-protein interfaces from cytochrome c oxidase I evolve faster than nonbinding surfaces, yet negative selection is the driving force". Genome Biology and Evolution. 6 (11): 3064–76. doi:10.1093/gbe/evu240. PMC 4255772. PMID 25359921.
- ↑ Alonso JR, Cardellach F, López S, Casademont J, Miró O (September 2003). "Carbon monoxide specifically inhibits cytochrome c oxidase of human mitochondrial respiratory chain". Pharmacology & Toxicology. 93 (3): 142–6. doi:10.1034/j.1600-0773.2003.930306.x. PMID 12969439.
- ↑ 19.0 19.1 Nicholls P, Marshall DC, Cooper CE, Wilson MT (October 2013). "Sulfide inhibition of and metabolism by cytochrome c oxidase". Biochemical Society Transactions. 41 (5): 1312–6. doi:10.1042/BST20130070. PMID 24059525. S2CID 11554252.
- ↑ Roberts M, Reiss MJ, Monger G (2000). Advanced Biology (in English). Nelson Thornes. ISBN 9780174387329. Archived from the original on 2022-02-24. Retrieved 2020-10-25.
- ↑ Roberts MB (1986). Biology: A Functional Approach (in English). Nelson Thornes. ISBN 9780174480198. Archived from the original on 2022-02-24. Retrieved 2020-10-25.
- ↑ Jensen P, Wilson MT, Aasa R, Malmström BG (December 1984). "Cyanide inhibition of cytochrome c oxidase. A rapid-freeze e.p.r. investigation". The Biochemical Journal. 224 (3): 829–37. doi:10.1042/bj2240829. PMC 1144519. PMID 6098268.
- ↑ 23.0 23.1 23.2 Gladwin MT, Shiva S (May 2009). "The ligand binding battle at cytochrome c oxidase: how NO regulates oxygen gradients in tissue". Circulation Research. 104 (10): 1136–8. doi:10.1161/CIRCRESAHA.109.198911. PMID 19461104.
- ↑ Cite error: Invalid
<ref>tag; no text was provided for refs namedpmid17906319 - ↑ Arnold S, Kadenbach B (October 1997). "Cell respiration s controlled by ATP, an allosteric inhibitor of cytochrome-c oxidase". Eur J Biochem. 249 (1): 350–354. doi:10.1111/j.1432-1033.1997.t01-1-00350.x. PMID 9363790.
- ↑ 26.0 26.1 Sadacharan SK, Singh B, Bowes T, Gupta RS (November 2005). "Localization of mitochondrial DNA encoded cytochrome c oxidase subunits I and II in rat pancreatic zymogen granules and pituitary growth hormone granules". Histochemistry and Cell Biology. 124 (5): 409–21. doi:10.1007/s00418-005-0056-2. PMID 16133117. S2CID 24440427.
- ↑ Gupta RS, Ramachandra NB, Bowes T, Singh B (2008). "Unusual cellular disposition of the mitochondrial molecular chaperones Hsp60, Hsp70 and Hsp10". Novartis Foundation Symposium. Novartis Foundation Symposia. 291: 59–68, discussion 69–73, 137–40. doi:10.1002/9780470754030.ch5. ISBN 9780470754030. PMID 18575266.
- ↑ 28.0 28.1 Soltys BJ, Gupta RS (1999). "Mitochondrial proteins at unexpected cellular locations: export of proteins from mitochondria from an evolutionary perspective". International Review of Cytology. 194: 133–96. doi:10.1016/S0074-7696(08)62396-7. ISBN 9780123645982. PMID 10494626.
- ↑ Soltys BJ, Gupta RS (May 1999). "Mitochondrial-matrix proteins at unexpected locations: are they exported?". Trends in Biochemical Sciences. 24 (5): 174–7. doi:10.1016/s0968-0004(99)01390-0. PMID 10322429.
- ↑ Pecina P, Houstková H, Hansíková H, Zeman J, Houstek J (2004). "Genetic defects of cytochrome c oxidase assembly" (PDF). Physiological Research. 53 Suppl 1: S213-23. PMID 15119951. Archived (PDF) from the original on 2011-07-18. Retrieved 2010-11-17.
- ↑ Zee JM, Glerum DM (December 2006). "Defects in cytochrome oxidase assembly in humans: lessons from yeast". Biochemistry and Cell Biology. 84 (6): 859–69. doi:10.1139/o06-201. PMID 17215873.
- ↑ Johar K, Priya A, Dhar S, Liu Q, Wong-Riley MT (November 2013). "Neuron-specific specificity protein 4 bigenomically regulates the transcription of all mitochondria- and nucleus-encoded cytochrome c oxidase subunit genes in neurons". Journal of Neurochemistry. 127 (4): 496–508. doi:10.1111/jnc.12433. PMC 3820366. PMID 24032355.
- ↑ Wong-Riley MT (March 1989). "Cytochrome oxidase: an endogenous metabolic marker for neuronal activity". Trends in Neurosciences. 12 (3): 94–101. doi:10.1016/0166-2236(89)90165-3. PMID 2469224. S2CID 42996304.
- ↑ Hevner RF, Wong-Riley MT (November 1989). "Brain cytochrome oxidase: purification, antibody production, and immunohistochemical/histochemical correlations in the CNS". The Journal of Neuroscience. 9 (11): 3884–98. doi:10.1523/jneurosci.09-11-03884.1989. PMC 6569932. PMID 2555458.
- ↑ Strazielle C, Hayzoun K, Derer M, Mariani J, Lalonde R (April 2006). "Regional brain variations of cytochrome oxidase activity in Relnrl-orl mutant mice". Journal of Neuroscience Research. 83 (5): 821–31. doi:10.1002/jnr.20772. PMID 16511878. S2CID 45787322.
- ↑ Strazielle C, Sturchler-Pierrat C, Staufenbiel M, Lalonde R (2003). "Regional brain cytochrome oxidase activity in beta-amyloid precursor protein transgenic mice with the Swedish mutation". Neuroscience. 118 (4): 1151–63. doi:10.1016/S0306-4522(03)00037-X. PMID 12732258. S2CID 9366458.
- ↑ Conejo NM, González-Pardo H, Gonzalez-Lima F, Arias JL (March 2010). "Spatial learning of the water maze: progression of brain circuits mapped with cytochrome oxidase histochemistry". Neurobiology of Learning and Memory. 93 (3): 362–71. doi:10.1016/j.nlm.2009.12.002. PMID 19969098. S2CID 24271956.
बाहरी संबंध
- The Cytochrome Oxidase home page at Rice University
- Interactive Molecular model of cytochrome c oxidase (Requires MDL Chime)
- UMich Orientation of Proteins in Membranes families/superfamily-4
- Cytochrome-c+Oxidase at the US National Library of Medicine Medical Subject Headings (MeSH)