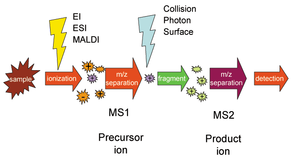
सिलेक्टेड रिएक्शन मॉनिटरिंग
सिलेक्टेड रिएक्शन मॉनिटरिंग (SRM), जिसे मल्टीपल रिएक्शन मॉनिटरिंग (MRM) भी कहा जाता है, एक अग्रानुक्रम मास स्पेक्ट्रोमेट्री में उपयोग की जाने वाली एक विधि है जिसमें एक विशेष द्रव्यमान के एक आयन को टेंडेम मास स्पेक्ट्रोमीटर के पहले चरण में और एक आयन उत्पाद का चयन किया जाता है। पता लगाने के लिए दूसरे मास स्पेक्ट्रोमीटर चरण में प्रीकर्सर (रसायन विज्ञान) की विखंडन प्रतिक्रिया का चयन किया जाता है।[1]
वेरिएंट
एसआरएम के एक सामान्य मामले का प्रतिनिधित्व किसके द्वारा किया जा सकता है
जहां अग्रगामी आयन ABCD+ मास स्पेक्ट्रोमेट्री (MS1) के पहले चरण द्वारा चुना जाता है, अणु AB और उत्पाद आयन CD में वियोजित हो जाता है+, और बाद वाले को मास स्पेक्ट्रोमेट्री (MS2) के दूसरे चरण द्वारा चुना जाता है और पता लगाया जाता है। अग्रदूत और उत्पाद आयन जोड़ी को एसआरएम संक्रमण कहा जाता है।[2] क्रमिक प्रतिक्रिया निगरानी (सीआरएम) एसआरएम के लिए मास स्पेक्ट्रोमेट्री के तीन या अधिक चरणों का क्रमिक अनुप्रयोग है, जिसे एक साधारण मामले में दर्शाया गया है
जहां एबीसीडी+ MS1 द्वारा चुना जाता है, अणु AB और आयन CD में वियोजित हो जाता है+.[3] आयन को दूसरे मास स्पेक्ट्रोमेट्री चरण MS2 में चुना जाता है, फिर आयन डी बनाने के लिए आगे विखंडन से गुजरता है+ जिसे तीसरे मास स्पेक्ट्रोमेट्री चरण MS3 में चुना गया और पता लगाया गया।
मल्टीपल रिएक्शन मॉनिटरिंग (MRM) एक या अधिक अग्रदूत आयनों से कई उत्पाद आयनों के लिए चयनित रिएक्शन मॉनिटरिंग का अनुप्रयोग है,[3][4] उदाहरण के लिए
जहां एबीसीडी+ MS1 द्वारा चुना जाता है और दो रास्तों से अलग हो जाता है, जिससे या तो AB बन जाता है+ या सीडी+. आयनों को MS2 द्वारा क्रमिक रूप से चुना जाता है और पता लगाया जाता है। समानांतर प्रतिक्रिया निगरानी (पीआरएम) एक उच्च रिज़ॉल्यूशन मास स्पेक्ट्रोमीटर का उपयोग करके एकल विश्लेषण में सभी संक्रमणों के समानांतर पता लगाने के साथ एसआरएम का अनुप्रयोग है।[5]
प्रोटिओमिक्स
मास स्पेक्ट्रोमेट्री द्वारा लक्षित मात्रात्मक प्रोटिओमिक्स के लिए एसआरएम का उपयोग किया जा सकता है।[6] आयनीकरण के बाद, उदाहरण के लिए, एक इलेक्ट्रोस्प्रे आयनीकरण आयन स्रोत, एक पेप्टाइड अग्रदूत को सबसे पहले इच्छित प्रजातियों की पर्याप्त आयन आबादी प्राप्त करने के लिए अलग किया जाता है। यह आबादी तब उत्पाद आयनों को उत्पन्न करने के लिए विखंडन (रसायन विज्ञान) है जिसका संकेत बहुतायत नमूने में पेप्टाइड की प्रचुरता का संकेत है। यह प्रयोग ट्रिपल क्वाड्रुपोल मास स्पेक्ट्रोमीटर पर किया जा सकता है, जहां द्रव्यमान-समाधान Q1 अग्रदूत को अलग करता है, क्यू2 एक टक्कर सेल के रूप में कार्य करता है, और द्रव्यमान-समाधान Q3 उत्पाद आयनों के माध्यम से चक्रित किया जाता है जो एक इलेक्ट्रॉन गुणक द्वारा अंतिम चतुष्कोणीय द्रव्यमान विश्लेषक से बाहर निकलने पर पता लगाया जाता है। एक पूर्ववर्ती/उत्पाद जोड़ी को अक्सर संक्रमण के रूप में संदर्भित किया जाता है। यह सुनिश्चित करने के लिए बहुत काम किया जाता है कि अधिकतम विशिष्टता वाले संक्रमणों का चयन किया जाए।
भारी-लेबल वाले समस्थानिक लेबलिंग का उपयोग करना (जैसे, ड्यूटेरियम, कार्बन-13|13सी, या नाइट्रोजन-15|15N) पेप्टाइड्स को मानक समाधान के रूप में एक जटिल मैट्रिक्स के लिए, SRM का उपयोग एक अंशांकन वक्र के निर्माण के लिए किया जा सकता है जो देशी, प्रकाश पेप्टाइड, और पूर्ण परिमाणीकरण (यानी, प्रतिलिपि संख्या प्रति कोशिका (जीव विज्ञान)) प्रदान कर सकता है। विस्तार से, इसका मूल प्रोटीन।[2]
एसआरएम का उपयोग जंगली-प्रकार और उत्परिवर्ती जीन (उत्परिवर्ती प्रोटीन) द्वारा एन्कोड किए गए प्रोटीन की पहचान करने और ट्यूमर और जैविक तरल पदार्थ में उनकी पूर्ण प्रतिलिपि संख्या को मापने के लिए किया गया है, इस प्रकार एक कोशिका में प्रोटीन की पूर्ण प्रतिलिपि संख्या के बारे में मूल प्रश्नों का उत्तर दिया जाता है, जो स्तनधारी कोशिकाओं और मानव शरीर के डिजिटल मॉडलिंग और ट्यूमर में आनुवंशिक रूप से असामान्य प्रोटीन के सापेक्ष स्तर और नैदानिक अनुप्रयोगों के लिए उपयोगी साबित होने में आवश्यक होगा।[7][8] SRM का उपयोग पेप्टाइड्स के पूर्ण उत्पाद आयन स्कैन को ट्रिगर करने की एक विधि के रूप में भी किया गया है a) SRM संक्रमण की विशिष्टता की पुष्टि करें, या b) विशिष्ट पोस्ट-ट्रांसलेशनल संशोधनों का पता लगाएं जो मानक MS विश्लेषणों की पहचान की सीमा से नीचे हैं।[9] 2017 में, SRM को एक अत्यधिक संवेदनशील और प्रतिलिपि प्रस्तुत करने योग्य द्रव्यमान स्पेक्ट्रोमेट्री-आधारित प्रोटीन लक्षित पहचान मंच (SAFE-SRM के हकदार) के रूप में विकसित किया गया है, और यह प्रदर्शित किया गया है कि SRM-आधारित नई पाइपलाइन के नैदानिक प्रोटिओमिक्स अनुप्रयोगों में पारंपरिक पर प्रमुख लाभ हैं। एसआरएम पाइपलाइन, और इसने एलिसा जैसे एंटीबॉडी-आधारित प्रोटीन बायोमार्कर डायग्नोस्टिक विधियों से नाटकीय रूप से बेहतर नैदानिक प्रदर्शन का प्रदर्शन किया है।[10]
यह भी देखें
- मात्रात्मक प्रोटिओमिक्स
- प्रोटीन मास स्पेक्ट्रोमेट्री
संदर्भ
- ↑ E. de Hoffmann (1996). "Tandem Mass Spectrometry: a Primer" (PDF). Journal of Mass Spectrometry. 31 (2): 129–137. Bibcode:1996JMSp...31..129D. doi:10.1002/(SICI)1096-9888(199602)31:2<129::AID-JMS305>3.0.CO;2-T.
- ↑ 2.0 2.1 Lange, Vinzenz; Picotti, Paola; Domon, Bruno; Aebersold, Ruedi (2008). "Selected reaction monitoring for quantitative proteomics: a tutorial". Molecular Systems Biology. 4: 222. doi:10.1038/msb.2008.61. ISSN 1744-4292. PMC 2583086. PMID 18854821.
- ↑ 3.0 3.1 Murray, Kermit K.; Boyd, Robert K.; Eberlin, Marcos N.; Langley, G. John; Li, Liang; Naito, Yasuhide (2013). "Definitions of terms relating to mass spectrometry (IUPAC Recommendations 2013)". Pure and Applied Chemistry. 85 (7): 1515–1609. doi:10.1351/PAC-REC-06-04-06. ISSN 1365-3075.
- ↑ Kondrat, R. W.; McClusky, G. A.; Cooks, R. G. (1978). "Multiple reaction monitoring in mass spectrometry/mass spectrometry for direct analysis of complex mixtures". Analytical Chemistry. 50 (14): 2017–2021. doi:10.1021/ac50036a020. ISSN 0003-2700.
- ↑ Peterson, A. C.; Russell, J. D.; Bailey, D. J.; Westphall, M. S.; Coon, J. J. (2012). "उच्च संकल्प और उच्च द्रव्यमान सटीकता मात्रात्मक, लक्षित प्रोटिओमिक्स के लिए समानांतर प्रतिक्रिया निगरानी". Molecular & Cellular Proteomics. 11 (11): 1475–1488. doi:10.1074/mcp.O112.020131. ISSN 1535-9476. PMC 3494192. PMID 22865924.
- ↑ Picotti, Paola; Aebersold, Ruedi (2012). "Selected reaction monitoring–based proteomics: workflows, potential, pitfalls and future directions". Nature Methods. 9 (6): 555–566. doi:10.1038/nmeth.2015. ISSN 1548-7091. PMID 22669653. S2CID 205420574.
- ↑ Wang Q, Chaerkady R, Wu J, et al. (February 2011). "उत्परिवर्ती प्रोटीन कैंसर-विशिष्ट बायोमार्कर के रूप में।". Proc. Natl. Acad. Sci. U.S.A. 108 (6): 2444–9. Bibcode:2011PNAS..108.2444W. doi:10.1073/pnas.1019203108. PMC 3038743. PMID 21248225.
- ↑ "निरपेक्ष प्रोटीन परिमाणीकरण के लिए चयनित रिएक्शन मॉनिटरिंग मास स्पेक्ट्रोमेट्री". Journal of Visualized Experiments.
- ↑ Unwin RD, Griffiths JG, et al. (August 2005). "उच्च संवेदनशीलता वाले प्रोटीन फास्फोराइलेशन की साइटों की पहचान करने के लिए एकाधिक प्रतिक्रिया निगरानी।". Molecular & Cellular Proteomics. 4 (8): 1134–44. doi:10.1074/mcp.M500113-MCP200. PMID 15923565.
- ↑ Wang Q, Zhang M, Tomita T, et al. (December 2017). "पेप्टाइड बायोमार्कर को मान्य करने के लिए चयनित प्रतिक्रिया निगरानी दृष्टिकोण।". Proc. Natl. Acad. Sci. U.S.A. 114 (51): 13519–13524. doi:10.1073/pnas.1712731114. PMC 5754789. PMID 29203663.
बाहरी संबंध
- SRMatlas; quantify proteins in complex proteome digests by mass spectrometry