डीएनए बेमेल विरोहण (डीएनए बेमेल रिपेयर)
This article includes a list of general references, but it lacks sufficient corresponding inline citations. (May 2018) (Learn how and when to remove this template message) |
| Part of a series on |
| Genetics |
|---|
 |

डीएनए मिसमैच रिपेयर (MMR) डीएनए प्रतिकृति और आनुवंशिक पुनर्संयोजन के दौरान उत्पन्न होने वाले गलत सम्मिलन, विलोपन और गलत समावेशन को पहचानने और मरम्मत करने के साथ-साथ डीएनए क्षति के कुछ रूपों की मरम्मत के लिए एक प्रणाली है।[1][2]
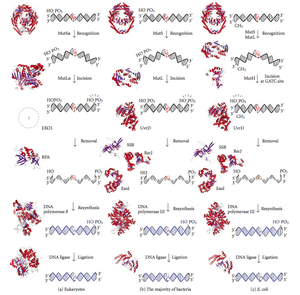
बेमेल मरम्मत कतरा-विशिष्ट है। डीएनए संश्लेषण के दौरान नए संश्लेषित (बेटी) स्ट्रैंड में आमतौर पर त्रुटियां शामिल होंगी। मरम्मत शुरू करने के लिए, बेमेल मरम्मत मशीनरी नए संश्लेषित स्ट्रैंड को टेम्पलेट (पैतृक) से अलग करती है। ग्राम-नकारात्मक जीवाणुओं में, क्षणिक हेमीमिथाइलेशन स्ट्रैंड्स को अलग करता है (माता-पिता मिथाइलेटेड है और बेटी नहीं है)। हालांकि, अन्य प्रोकैरियोट्स और यूकेरियोट्स में, सटीक तंत्र स्पष्ट नहीं है। यह संदेह है कि, यूकेरियोट्स में, नव संश्लेषित लैगिंग-स्ट्रैंड डीएनए में क्षणिक रूप से निक्स (डीएनए) होते हैं (डीएनए लिगेज द्वारा सील किए जाने से पहले) होता है और एक संकेत प्रदान करता है जो बेमेल प्रूफरीडिंग सिस्टम को उपयुक्त स्ट्रैंड को निर्देशित करता है। इसका तात्पर्य यह है कि ये निक अग्रणी स्ट्रैंड में मौजूद होने चाहिए, और इसके प्रमाण हाल ही में मिले हैं।[3]
हाल के काम[4] में दिखाया गया है कि निक्स आरएफसी-आश्रित लोडिंग के लिए प्रतिकृति स्लाइडिंग क्लैंप, प्रोलिफायरिंग सेल परमाणु एंटीजन (पीसीएनए) के लिए साइट हैं, एक अभिविन्यास-विशिष्ट तरीके से जैसे कि डोनट-आकार प्रोटीन का एक चेहरा 3'-OH अंत निक पर जुड़ा होता है। लोडेड PCNA तब एक बेमेल और MutSalpha या MutSbeta की उपस्थिति में MutLalpha endonuclease [5] की कार्रवाई को बेटी स्ट्रैंड पर निर्देशित करता है।
कोई भी उत्परिवर्ती घटना जो डीएनए की सुपरहिकल संरचना को बाधित करती है, उसके साथ कोशिका की आनुवंशिक स्थिरता से समझौता करने की क्षमता होती है। तथ्य यह है कि क्षति का पता लगाने और मरम्मत प्रणाली उतनी ही जटिल है जितनी प्रतिकृति मशीनरी स्वयं डीएनए निष्ठा से जुड़े महत्व के विकास पर प्रकाश डालती है।
बेमेल आधारों के उदाहरणों में G/T या A/C पेयरिंग शामिल है (डीएनए मरम्मत देखें)। बेमेल आमतौर पर डीएनए प्रतिकृति के दौरान आधारों के टॉटोमेराइज़ेशन के कारण होते हैं। बेमेल के कारण होने वाली विकृति की पहचान करके, टेम्पलेट और गैर-टेम्प्लेट स्ट्रैंड का निर्धारण करके और गलत तरीके से शामिल किए गए आधार को हटाकर सही न्यूक्लियोटाइड के साथ बदलकर क्षति की मरम्मत की जाती है। निष्कासन प्रक्रिया में केवल बेमेल न्यूक्लियोटाइड से अधिक शामिल है। नए संश्लेषित डीएनए स्ट्रैंड के कुछ या हजारों बेस जोड़े को हटाया जा सकता है।
बेमेल मरम्मत प्रोटीन
| DNA mismatch repair protein, C-terminal domain | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 hpms2-atpgs | |||||||||
| Identifiers | |||||||||
| Symbol | DNA_mis_repair | ||||||||
| Pfam | PF01119 | ||||||||
| Pfam clan | CL0329 | ||||||||
| InterPro | IPR013507 | ||||||||
| PROSITE | PDOC00057 | ||||||||
| SCOP2 | 1bkn / SCOPe / SUPFAM | ||||||||
| |||||||||
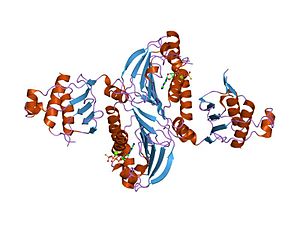
मिसमैच रिपेयर प्रोकैरियोट्स से यूकेरियोट्स तक एक अत्यधिक संरक्षित प्रक्रिया है। बेमेल मरम्मत के लिए पहला सबूत एस निमोनिया (हेक्सा और हेक्सबी जीन) से प्राप्त किया गया था। ई. कोलाई पर बाद के काम ने कई जीनों की पहचान की है, जो उत्परिवर्तनीय रूप से निष्क्रिय होने पर अतिपरिवर्तनीय तनाव पैदा करते हैं। इसलिए, जीन उत्पादों को "मट" प्रोटीन कहा जाता है, और बेमेल मरम्मत प्रणाली के प्रमुख सक्रिय घटक हैं। बेमेल का पता लगाने और उसकी मरम्मत करने वाली मशीनरी को निर्देशित करने के लिए इनमें से तीन प्रोटीन आवश्यक हैं: MutS-1, MutH और MutL (MutS, HexA का एक होमोलॉग है और HexB का MutL है)।
MutS एक डिमर (MutS2) बनाता है जो बेटी स्ट्रैंड पर बेमेल आधार को पहचानता है और उत्परिवर्तित डीएनए को बांधता है। MutH बेटी डीएनए के साथ हेमीमेथिलेटेड साइटों पर बांधता है, लेकिन इसकी क्रिया अव्यक्त होती है, केवल एक MutL डिमर (MutL2) के संपर्क में आने पर सक्रिय होती है) जो MutS-DNA कॉम्प्लेक्स को बांधता है और MutS2 के बीच मध्यस्थ के रूप में कार्य करता है और MutH बाद वाले को सक्रिय कर रहा है। बेमेल के निकटतम d(GATC) मेथिलिकरण स्थल की खोज के लिए डीएनए को लूप आउट किया जाता है, जो 1 kb तक दूर हो सकता है। MutS-DNA कॉम्प्लेक्स द्वारा सक्रिय होने पर, MutH हेमीमेथिलेटेड साइट के पास बेटी को फंसाता है। MutL एक विशिष्ट 3' से 5' ध्रुवीयता के साथ दो स्ट्रैंड को अलग करने के लिए UvrD हेलिकेज़ (डीएनए हेलिकेज़ II) की भर्ती करता है। संपूर्ण MutSHL कॉम्प्लेक्स तब बेमेल की दिशा में डीएनए के साथ स्लाइड करता है, जिससे किनारा जाने के लिए मुक्त हो जाता है। एक एक्सोन्यूक्लिज़ कॉम्प्लेक्स का पता लगाता है और ss-DNA tail को पचाता है। भर्ती किया गया एक्सोन्यूक्लिज़ इस बात पर निर्भर करता है कि बेमेल के किस तरफ MutH स्ट्रैंड - 5' या 3' को उकसाता है। यदि MutH द्वारा बनाया गया निक बेमेल के 5' छोर पर है, तो या तो RecJ या ExoVII (दोनों 5' से 3' एक्सोन्यूक्लाइजेस) का उपयोग किया जाता है। यदि, हालांकि, निक बेमेल के 3' छोर पर है, तो ExoI (3' से 5' एंजाइम) का उपयोग किया जाता है।
पूरी प्रक्रिया बेमेल साइट के बाद समाप्त होती है - यानी, साइट और उसके आस-पास के न्यूक्लियोटाइड दोनों ही पूरी तरह से उत्सर्जित होते हैं। एक्सोन्यूक्लिज़ द्वारा बनाए गए सिंगल-स्ट्रैंड गैप को डीएनए पोलीमरेज़ III (सिंगल-स्ट्रैंड-बाइंडिंग प्रोटीन द्वारा सहायता प्राप्त) द्वारा रिपेयर किया जा सकता है, जो दूसरे स्ट्रैंड को टेम्प्लेट के रूप में उपयोग करता है, और अंत में डीएनए लिगेज द्वारा सील कर दिया जाता है। डीएनए मिथाइलेज़ तब बेटी स्ट्रैंड को तेजी से मिथाइलेट करता है।
MutS होमोलॉग्स
बाध्य होने पर, MutS2 डिमर डीएनए हेलिक्स को मोड़ देता है और लगभग 20 बेस पेयर को ढाल देता है। इसकी ATPase गतिविधि कमजोर है, और एडेनोसाइन ट्रायफ़ोस्फेट ATP के बंधन से अणु की सतह पर तृतीयक संरचनाओं का निर्माण होता है। MutS की क्रिस्टल संरचना से पता चलता है कि यह असाधारण रूप से असममित है, और, जबकि इसकी सक्रिय रचना एक मंदक है, दो हिस्सों में से केवल एक बेमेल साइट के साथ संपर्क करता है।
यूकेरियोट्स में, MutS homologs समरूपता दो प्रमुख विधर्मी बनाते हैं: Msh2/Msh6 (MutSα) और Msh2/Msh3 (MutSβ)। MutSα मार्ग मुख्य रूप से आधार प्रतिस्थापन और छोटे-पाश बेमेल मरम्मत में शामिल है। MutSβ पाथवे लार्ज-लूप (~10 न्यूक्लियोटाइड लूप्स) रिपेयर के अलावा स्मॉल-लूप रिपेयर में भी शामिल है। हालाँकि, MutSβ आधार प्रतिस्थापन की मरम्मत नहीं करता है।
MutL होमोलॉग्स
MutL में कमजोर ATPase गतिविधि भी है (यह आंदोलन के प्रयोजनों के लिए ATP का उपयोग करती है)। यह MutS और MutH के साथ एक सम्मिश्र बनाता है, डीएनए पर MutS पदचिह्न को बढ़ाता है।
हालांकि, UvrD की प्रक्रियात्मकता (वह दूरी जो एंजाइम डीएनए के साथ आगे बढ़ सकती है) केवल ~ 40-50 बीपी है। क्योंकि MutH द्वारा बनाए गए निक और बेमेल के बीच की दूरी ~ 600 bp औसत हो सकती है, अगर कोई अन्य UvrD भारित नहीं किया गया है, तो इसके पूरक स्ट्रैंड को फिर से जोड़ने के लिए स्वतंत्र है, जिससे प्रक्रिया फिर से शुरू हो जाती है। हालाँकि, जब MutL द्वारा सहायता प्रदान की जाती है, तो UvrD लोडिंग की दर बहुत बढ़ जाती है। जबकि व्यक्तिगत यूवीआरडी अणुओं की प्रक्रियात्मकता (और एटीपी उपयोग) समान रहती है, डीएनए पर कुल प्रभाव काफी बढ़ जाता है; डीएनए के पास फिर से एनील करने का कोई मौका नहीं है, क्योंकि प्रत्येक यूवीआरडी डीएनए के 40-50 BP को खोल देता है, अलग हो जाता है और फिर प्रक्रिया को दोहराते हुए तुरंत दूसरे यूवीआरडी द्वारा बदल दिया जाता है। यह डीएनए के बड़े हिस्से को एक्सोन्यूक्लिएज पाचन के लिए उजागर करता है, जिससे गलत डीएनए के त्वरित छांटने (और बाद में प्रतिस्थापन) की अनुमति मिलती है।
यूकेरियोट्स में MLH1, MLH2, MLH3, PMS1, और PMS2 के रूप में नामित पांच MutL होमोलॉग हैं। वे हेटेरोडिमर बनाते हैं जो ई. कोलाई में MutL की नकल करते हैं। प्रोकैरियोटिक म्यूटल के मानव समरूप तीन परिसरों का निर्माण करते हैं जिन्हें MutLα, MutLβ और MutLγ कहा जाता है। MutLα कॉम्प्लेक्स MLH1 और PMS2 सबयूनिट्स से बना है, MutLβ हेटेरोडिमर MLH1 और PMS1 से बना है, जबकि MutLγ MLH1 और MLH3 से बना है। MutLα एक एंडोन्यूक्लिज़ के रूप में कार्य करता है जो बेमेल और अन्य आवश्यक प्रोटीन, MutSα और PCNA द्वारा सक्रियण पर बेटी स्ट्रैंड में स्ट्रैंड ब्रेक का परिचय देता है। ये स्ट्रैंड रुकावट एक एक्सोन्यूक्लिज़ गतिविधि के लिए प्रवेश बिंदु के रूप में काम करती है जो बेमेल डीएनए को हटा देती है। बेमेल मरम्मत में MutLβ और MutLγ द्वारा निभाई गई भूमिकाएं कम समझी जाती हैं।
=== MutH: ई. कोलाई और साल्मोनेला === में मौजूद एक एंडोन्यूक्लिएज
MutH एक बहुत ही कमजोर एंडोन्यूक्लिएज है जो एक बार MutL (जो खुद MutS के लिए बाध्य है) से बंध जाने पर सक्रिय हो जाता है। यह अनमेथिलेटेड डीएनए और हेमीमेथिलेटेड डीएनए के अनमेथिलेटेड स्ट्रैंड को हटा देता है लेकिन पूरी तरह से मिथाइलेटेड डीएनए को नहीं निकालता है। प्रयोगों से पता चला है कि यदि किसी भी स्ट्रैंड में मिथाइलेट नहीं किया जाता है तो बेमेल मरम्मत यादृच्छिक होती है।[citation needed] इन व्यवहारों ने प्रस्ताव को जन्म दिया कि MutH यह निर्धारित करता है कि किस स्ट्रैंड में बेमेल है।
MutH का कोई यूकेरियोटिक समरूपता नहीं है। इसका एंडोन्यूक्लिज़ कार्य MutL होमोलॉग्स द्वारा किया जाता है, जिसमें कुछ विशेष 5'-3' एक्सोन्यूक्लिज़ गतिविधि होती है। यूकेरियोट्स में नए संश्लेषित बेटी स्ट्रैंड से बेमेल को हटाने के लिए स्ट्रैंड पूर्वाग्रह प्रतिकृति के दौरान बनाए गए नए स्ट्रैंड में ओकाजाकी टुकड़ों के मुक्त 3' सिरों द्वारा प्रदान किया जा सकता है।
पीसीएनए β-स्लाइडिंग क्लैंप
PCNA और β-स्लाइडिंग क्लैंप क्रमशः MutSα/β और MutS के साथ जुड़ते हैं। हालाँकि प्रारंभिक रिपोर्टों ने सुझाव दिया था कि PCNA-MutSα कॉम्प्लेक्स बेमेल पहचान को बढ़ा सकता है,[5] यह हाल ही में प्रदर्शित किया गया है[6] PCNA की उपस्थिति या अनुपस्थिति में बेमेल के लिए MutSα की आत्मीयता में कोई स्पष्ट परिवर्तन नहीं हुआ है। इसके अलावा, MutSα के म्यूटेंट जो इन विट्रो में पीसीएनए के साथ बातचीत करने में असमर्थ हैं, बेमेल प्रकार के स्तरों के पास बेमेल मान्यता और बेमेल छांटने की क्षमता प्रदर्शित करते हैं। इस तरह के म्यूटेंट 5' स्ट्रैंड ब्रेक द्वारा निर्देशित मरम्मत प्रतिक्रिया में दोषपूर्ण हैं, जो प्रतिक्रिया के बाद के चरण में पहली बार MutSα फ़ंक्शन का सुझाव देते हैं।
नैदानिक महत्व
बेमेल मरम्मत में वंशानुगत दोष
म्यूट प्रोटीन के मानव समरूपों में उत्परिवर्तन जीनोमिक स्थिरता को प्रभावित करते हैं, जिसके परिणामस्वरूप कुछ मानव कैंसर में माइक्रोसेटेलाइट अस्थिरता (MSI) हो सकती है। विशेष रूप से, वंशानुगत नॉनपोलिपोसिस कोलोरेक्टल कैंसर (HNPCC या लिंच सिंड्रोम) को क्रमशः MutS और MutL होमोलॉग्स MSH2 और MLH1 को एन्कोडिंग करने वाले जीन में हानिकारक जर्मलाइन वेरिएंट के लिए जिम्मेदार ठहराया जाता है, जिन्हें इस प्रकार ट्यूमर सप्रेसर जीन के रूप में वर्गीकृत किया जाता है। एचएनपीसीसी का एक उपप्रकार, मुइर-टोरे सिंड्रोम (एमटीएस), त्वचा के ट्यूमर से जुड़ा है। यदि एमएमआर जीन की विरासत में मिली दोनों प्रतियाँ (एलील) आनुवंशिक रूपांतरों को नुकसान पहुंचाती हैं, तो इसका परिणाम बहुत ही दुर्लभ और गंभीर स्थिति में होता है: बेमेल मरम्मत कैंसर सिंड्रोम (या संवैधानिक बेमेल मरम्मत की कमी, सीएमएमआर-डी), ट्यूमर की कई घटनाओं के रूप में प्रकट होता है। कम उम्र, अक्सर कोलन और मस्तिष्क का ट्यूमर ।[7]
=== बेमेल मरम्मत जीन === की एपिजेनेटिक साइलेंसिंग
डीएनए की मरम्मत की कमी वाले विकीर्ण कैंसर में शायद ही कभी डीएनए की मरम्मत करने वाले जीन में उत्परिवर्तन होता है, लेकिन इसके बजाय वे प्रमोटर मेथिलिकरण जैसे एपिजेनेटिक्स परिवर्तन होते हैं जो डीएनए की मरम्मत जीन अभिव्यक्ति को रोकते हैं।[8] आमतौर पर MLH1 (9.8%), या कभी-कभी MSH2, MSH6 या PMS2 (सभी ≤1.5%) की हानि के कारण लगभग 13% कोलोरेक्टल कैंसर डीएनए बेमेल मरम्मत में कमी वाले होते हैं।[9] अधिकांश MLH1-कमी वाले विकीर्ण कोलोरेक्टल कैंसर के लिए, कमी MLH1 प्रमोटर मेथिलिकरण के कारण थी।[9] अन्य कैंसर प्रकारों में MLH1 हानि की उच्च आवृत्तियाँ होती हैं (नीचे दी गई तालिका देखें), जो फिर से बड़े पैमाने पर MLH1 जीन के प्रवर्तक के मिथाइलेशन का परिणाम हैं। एमएमआर कमियों में अंतर्निहित एक अलग एपिजेनेटिक तंत्र में एक माइक्रोआरएनए की अति-अभिव्यक्ति शामिल हो सकती है, उदाहरण के लिए miR-155 का स्तर कोलोरेक्टल कैंसर में एमएलएच1 या एमएसएच2 की अभिव्यक्ति के साथ विपरीत रूप से सहसंबंधित होते हैं।[10]
| Cancer type | Frequency of deficiency in cancer | Frequency of deficiency in adjacent field defect |
|---|---|---|
| Stomach | 32%[11][12] | 24%-28% |
| Stomach (foveolar type tumors) | 74%[13] | 71% |
| Stomach in high-incidence Kashmir Valley | 73%[14] | 20% |
| Esophageal | 73%[15] | 27% |
| Head and neck squamous cell carcinoma (HNSCC) | 31%-33%[16][17] | 20%-25% |
| Non-small cell lung cancer (NSCLC) | 69%[18] | 72% |
| Colorectal | 10%[9] |
क्षेत्र दोषों में एमएमआर विफलता
फील्ड डिफेक्ट (फील्ड कैंसराइजेशन) एपिथेलियम का एक क्षेत्र है जिसे एपिजेनेटिक या जेनेटिक परिवर्तनों द्वारा पूर्वनिर्मित किया गया है, जो इसे कैंसर के विकास की ओर अग्रसर करता है। जैसा कि रुबिन द्वारा बताया गया है ... इस बात के प्रमाण हैं कि उत्परिवर्ती फेनोटाइप मानव कोलोरेक्टल ट्यूमर में पाए जाने वाले 80% से अधिक दैहिक उत्परिवर्तन टर्मिनल क्लोनल विस्तार की शुरुआत से पहले होते हैं।[19][20] इसी तरह, वोगेलस्टीन एट अल।[21] बताते हैं कि ट्यूमर में पहचाने जाने वाले दैहिक उत्परिवर्तन के आधे से अधिक एक पूर्व-नियोप्लास्टिक चरण (एक क्षेत्र दोष में) में, स्पष्ट रूप से सामान्य कोशिकाओं के विकास के दौरान होते हैं।
MLH1 की कमी ट्यूमर के आसपास के क्षेत्र दोषों (हिस्टोलोगिक रूप से सामान्य ऊतकों) में आम थी; ऊपर तालिका देखें। एपिजेनेटिक रूप से खामोश या उत्परिवर्तित MLH1 संभवतः स्टेम सेल पर एक चयनात्मक लाभ प्रदान नहीं करेगा, हालांकि, यह उत्परिवर्तन दर में वृद्धि करेगा, और एक या अधिक उत्परिवर्तित जीन सेल को एक चयनात्मक लाभ प्रदान कर सकते हैं। जब उत्परिवर्तित स्टेम सेल एक विस्तारित क्लोन उत्पन्न करता है तो कमी एमएलएच 1 जीन को एक चुनिंदा निकट-तटस्थ यात्री (हिच-हाइकर) जीन के रूप में ले जाया जा सकता है। एपिजेनेटिक रूप से दमित MLH1 के साथ एक क्लोन की निरंतर उपस्थिति आगे उत्परिवर्तन उत्पन्न करना जारी रखेगी, जिनमें से कुछ ट्यूमर उत्पन्न कर सकते हैं।
मनुष्यों में एमएमआर घटक
मनुष्यों में, सात डीएनए मिसमैच रिपेयर (MMR) प्रोटीन (MLH1, MLH3, MSH2, MSH3, MSH6, PMS1 और PMS2) डीएनए बेमेल की मरम्मत शुरू करने के लिए अनुक्रमिक चरणों में समन्वित रूप से काम करते हैं।[22] इसके अलावा Exo1 -डिपेंडेंट और Exo1-इंडिपेंडेंट एमएमआर उपमार्ग हैं।[23]
मनुष्यों में बेमेल मरम्मत (MMR जीन द्वारा दीक्षा के बाद) में शामिल अन्य जीन उत्पादों में डीएनए पोलीमरेज़ डेल्टा, PCNA, RPA, HMGB1, RFC और DNA ligase I, प्लस हिस्टोन और क्रोमेटिन संशोधित कारक शामिल हैं।[24][25]
कुछ परिस्थितियों में, एमएमआर मार्ग एक त्रुटि-प्रवण डीएनए पोलीमरेज़ एटा (पीओएलएच) की भर्ती कर सकता है। यह बी-लिम्फोसाइट्स में दैहिक अतिपरिवर्तन के दौरान होता है, जहां पीओएलएच का उपयोग एंटीबॉडी जीन में आनुवंशिक भिन्नता लाने के लिए किया जाता है।[26] हालांकि, जीनोटॉक्सिन के संपर्क में आने पर यह त्रुटि-प्रवण एमएमआर मार्ग अन्य प्रकार की मानव कोशिकाओं में शुरू हो सकता है [27] और वास्तव में यह विभिन्न मानव कैंसर में व्यापक रूप से सक्रिय है, जिससे म्यूटेशन होता है जो पीओएलएच गतिविधि का संकेत देता है।[28]
एमएमआर और उत्परिवर्तन आवृत्ति
बेमेल और इंडल्स को पहचानना और मरम्मत करना कोशिकाओं के लिए महत्वपूर्ण है क्योंकि ऐसा करने में विफलता के परिणामस्वरूप माइक्रोसैटेलाइट अस्थिरता (MSI) और एक उन्नत सहज उत्परिवर्तन दर (म्यूटेटर फेनोटाइप) होता है। अन्य प्रकार के कैंसर की तुलना में, एमएमआर-कमी (एमएसआई) कैंसर में उत्परिवर्तन की बहुत अधिक आवृत्ति होती है, मेलेनोमा और फेफड़ों के कैंसर के करीब,[29] यूवी विकिरण और उत्परिवर्ती रसायनों के बहुत अधिक जोखिम के कारण होने वाले कैंसर के प्रकार।
एक बहुत ही उच्च उत्परिवर्तन बोझ के अलावा, एमएमआर की कमी के परिणामस्वरूप मानव जीनोम में दैहिक उत्परिवर्तन का असामान्य वितरण होता है: इससे पता चलता है कि एमएमआर अधिमानतः जीन-समृद्ध, प्रारंभिक-प्रतिकृति यूक्रोमैटिक क्षेत्रों की रक्षा करता है।[30] इसके विपरीत, जीन-समृद्ध, देर से प्रतिकृति करने वाले हेटरोक्रोमैटिक जीनोम क्षेत्र कई मानव ट्यूमर में उच्च उत्परिवर्तन दर प्रदर्शित करते हैं।[31]
हिस्टोन संशोधन H3K36me3, सक्रिय क्रोमैटिन का एक एपिजेनेटिक्स चिह्न, MSH2-MSH6 (hMutSα) परिसर को भर्ती करने की क्षमता रखता है।[32] लगातार, H3K36me3 के उच्च स्तर वाले मानव जीनोम के क्षेत्र MMR गतिविधि के कारण कम उत्परिवर्तन जमा करते हैं।[28]
ट्यूमर में कई डीएनए मरम्मत मार्गों का नुकसान
एमएमआर की कमी अधिकतर अन्य डीएनए मरम्मत जीनों के नुकसान के साथ समन्वय में होती है।[8]उदाहरण के लिए, MMR जीन MLH1 और MLH3 के साथ-साथ 11 अन्य डीएनए रिपेयर जीन (जैसे O-6-मिथाइलगुआनिन-डीएनए मिथाइलट्रांसफेरेज़ MGMT और कई न्यूक्लियोटाइड NER पाथवे जीन) सामान्य मस्तिष्क के ऊतकों के विपरीत निम्न ग्रेड के साथ-साथ उच्च ग्रेड एस्ट्रोसाइटोमास में काफी डाउन-रेगुलेटेड थे।[33] इसके अलावा MLH1 और MGMT अभिव्यक्ति को गैस्ट्रिक कैंसर के 135 नमूनों में बारीकी से सहसंबद्ध किया गया था और MLH1 और MGMT के नुकसान को ट्यूमर की प्रगति के दौरान समकालिक रूप से त्वरित किया गया था।[34]
कई डीएनए रिपेयर जीनों की त्रुटिपूर्ण अभिव्यक्ति अधिकतर कैंसर में पाई जाती है[8]और प्राय: कैंसर में पाए जाने वाले हजारों म्यूटेशन में योगदान कर सकते हैं (कैंसर में म्यूटेशन फ्रिक्वेंसी देखें)।
उम्र बढ़ने
एक लोकप्रिय विचार जो महत्वपूर्ण प्रयोगात्मक समर्थन प्राप्त करने में विफल रहा है, यह विचार है कि उत्परिवर्तन डीएनए क्षति से अलग उम्र बढ़ने का प्राथमिक कारण है। म्यूटल होमोलॉग Pms2 में दोषपूर्ण चूहे के सभी ऊतकों में लगभग 100 गुना उच्च उत्परिवर्तन आवृत्ति होती है, लेकिन यह अधिक तेजी से उम्र के लिए प्रकट नहीं होता है।[35] प्रारंभिक आरंभ में कार्सिनोजेनेसिस और पुरुष बांझपन को छोड़कर ये चूहे अधिकतर सामान्य विकास और जीवन प्रदर्शित करते हैं।
यह भी देखें
- आधार एक्सिशन मरम्मत
- न्यूक्लियोटाइड एक्सिशन मरम्मत
संदर्भ
- ↑ Iyer RR, Pluciennik A, Burdett V, Modrich PL (February 2006). "DNA mismatch repair: functions and mechanisms". Chemical Reviews. 106 (2): 302–23. doi:10.1021/cr0404794. PMID 16464007.
- ↑ Larrea AA, Lujan SA, Kunkel TA (May 2010). "SnapShot: DNA mismatch repair". Cell. 141 (4): 730–730.e1. doi:10.1016/j.cell.2010.05.002. PMID 20478261. S2CID 26969788.
- ↑ Heller RC, Marians KJ (December 2006). "रेप्लिसोम असेंबली और रुके हुए प्रतिकृति फोर्क्स का सीधा पुनरारंभ". Nature Reviews. Molecular Cell Biology. 7 (12): 932–43. doi:10.1038/nrm2058. PMID 17139333. S2CID 27666329.
- ↑ Pluciennik A, Dzantiev L, Iyer RR, Constantin N, Kadyrov FA, Modrich P (September 2010). "PCNA function in the activation and strand direction of MutLα endonuclease in mismatch repair". Proceedings of the National Academy of Sciences of the United States of America. 107 (37): 16066–71. doi:10.1073/pnas.1010662107. PMC 2941292. PMID 20713735.
- ↑ Flores-Rozas H, Clark D, Kolodner RD (November 2000). "Proliferating cell nuclear antigen and Msh2p-Msh6p interact to form an active mispair recognition complex". Nature Genetics. 26 (3): 375–8. doi:10.1038/81708. PMID 11062484. S2CID 20861705.
- ↑ Iyer RR, Pohlhaus TJ, Chen S, Hura GL, Dzantiev L, Beese LS, Modrich P (May 2008). "मानव डीएनए बेमेल मरम्मत में MutSalpha-proliferating सेल परमाणु प्रतिजन बातचीत". The Journal of Biological Chemistry. 283 (19): 13310–9. doi:10.1074/jbc.M800606200. PMC 2423938. PMID 18326858.
- ↑ Online Mendelian Inheritance in Man (OMIM): 276300
- ↑ 8.0 8.1 8.2 Bernstein C, Bernstein H (May 2015). "गैस्ट्रोइंटेस्टाइनल कैंसर के लिए प्रगति में डीएनए की मरम्मत की एपिजेनेटिक कमी". World Journal of Gastrointestinal Oncology. 7 (5): 30–46. doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ↑ 9.0 9.1 9.2 Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, et al. (May 2005). "Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer". Gastroenterology. 128 (5): 1160–71. doi:10.1053/j.gastro.2005.01.056. PMID 15887099.
- ↑ Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, et al. (April 2010). "Modulation of mismatch repair and genomic stability by miR-155". Proceedings of the National Academy of Sciences of the United States of America. 107 (15): 6982–7. Bibcode:2010PNAS..107.6982V. doi:10.1073/pnas.1002472107. PMC 2872463. PMID 20351277.
- ↑ Kupčinskaitė-Noreikienė R, Skiecevičienė J, Jonaitis L, Ugenskienė R, Kupčinskas J, Markelis R, et al. (2013). "CpG island methylation of the MLH1, MGMT, DAPK, and CASP8 genes in cancerous and adjacent noncancerous stomach tissues". Medicina. 49 (8): 361–6. doi:10.3390/medicina49080056. PMID 24509146.
- ↑ Waki T, Tamura G, Tsuchiya T, Sato K, Nishizuka S, Motoyama T (August 2002). "Promoter methylation status of E-cadherin, hMLH1, and p16 genes in nonneoplastic gastric epithelia". The American Journal of Pathology. 161 (2): 399–403. doi:10.1016/S0002-9440(10)64195-8. PMC 1850716. PMID 12163364.
- ↑ Endoh Y, Tamura G, Ajioka Y, Watanabe H, Motoyama T (September 2000). "Frequent hypermethylation of the hMLH1 gene promoter in differentiated-type tumors of the stomach with the gastric foveolar phenotype". The American Journal of Pathology. 157 (3): 717–22. doi:10.1016/S0002-9440(10)64584-1. PMC 1949419. PMID 10980110.
- ↑ Wani M, Afroze D, Makhdoomi M, Hamid I, Wani B, Bhat G, et al. (2012). "Promoter methylation status of DNA repair gene (hMLH1) in gastric carcinoma patients of the Kashmir valley" (PDF). Asian Pacific Journal of Cancer Prevention. 13 (8): 4177–81. doi:10.7314/apjcp.2012.13.8.4177. PMID 23098428.
- ↑ Chang Z, Zhang W, Chang Z, Song M, Qin Y, Chang F, et al. (January 2015). "Expression characteristics of FHIT, p53, BRCA2 and MLH1 in families with a history of oesophageal cancer in a region with a high incidence of oesophageal cancer". Oncology Letters. 9 (1): 430–436. doi:10.3892/ol.2014.2682. PMC 4246613. PMID 25436004.
- ↑ Tawfik HM, El-Maqsoud NM, Hak BH, El-Sherbiny YM (2011). "Head and neck squamous cell carcinoma: mismatch repair immunohistochemistry and promoter hypermethylation of hMLH1 gene". American Journal of Otolaryngology. 32 (6): 528–36. doi:10.1016/j.amjoto.2010.11.005. PMID 21353335.
- ↑ Zuo C, Zhang H, Spencer HJ, Vural E, Suen JY, Schichman SA, et al. (October 2009). "Increased microsatellite instability and epigenetic inactivation of the hMLH1 gene in head and neck squamous cell carcinoma". Otolaryngology–Head and Neck Surgery. 141 (4): 484–90. doi:10.1016/j.otohns.2009.07.007. PMID 19786217. S2CID 8357370.
- ↑ Safar AM, Spencer H, Su X, Coffey M, Cooney CA, Ratnasinghe LD, et al. (June 2005). "Methylation profiling of archived non-small cell lung cancer: a promising prognostic system". Clinical Cancer Research. 11 (12): 4400–5. doi:10.1158/1078-0432.CCR-04-2378. PMID 15958624.
- ↑ Rubin H (March 2011). "Fields and field cancerization: the preneoplastic origins of cancer: asymptomatic hyperplastic fields are precursors of neoplasia, and their progression to tumors can be tracked by saturation density in culture". BioEssays. 33 (3): 224–31. doi:10.1002/bies.201000067. PMID 21254148. S2CID 44981539.
- ↑ Tsao JL, Yatabe Y, Salovaara R, Järvinen HJ, Mecklin JP, Aaltonen LA, et al. (February 2000). "व्यक्तिगत कोलोरेक्टल ट्यूमर इतिहास का आनुवंशिक पुनर्निर्माण". Proceedings of the National Academy of Sciences of the United States of America. 97 (3): 1236–41. Bibcode:2000PNAS...97.1236T. doi:10.1073/pnas.97.3.1236. PMC 15581. PMID 10655514.
- ↑ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (March 2013). "कैंसर जीनोम परिदृश्य". Science. 339 (6127): 1546–58. Bibcode:2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ↑ Pal T, Permuth-Wey J, Sellers TA (August 2008). "डिम्बग्रंथि के कैंसर में बेमेल-मरम्मत की कमी की नैदानिक प्रासंगिकता की समीक्षा". Cancer. 113 (4): 733–42. doi:10.1002/cncr.23601. PMC 2644411. PMID 18543306.
{{cite journal}}: zero width space character in|title=at position 57 (help) - ↑ Goellner EM, Putnam CD, Kolodner RD (August 2015). "एक्सोन्यूक्लिज़ 1-आश्रित और स्वतंत्र बेमेल मरम्मत". DNA Repair. 32: 24–32. doi:10.1016/j.dnarep.2015.04.010. PMC 4522362. PMID 25956862.
- ↑ Li GM (January 2008). "डीएनए बेमेल मरम्मत के तंत्र और कार्य". Cell Research. 18 (1): 85–98. doi:10.1038/cr.2007.115. PMID 18157157.
- ↑ Li GM (July 2014). "New insights and challenges in mismatch repair: getting over the chromatin hurdle". DNA Repair. 19: 48–54. doi:10.1016/j.dnarep.2014.03.027. PMC 4127414. PMID 24767944.
- ↑ Chahwan R, Edelmann W, Scharff MD, Roa S (August 2012). "त्रुटि-प्रवण बेमेल मरम्मत द्वारा एंटीबॉडी विविधता की सहायता करना". Seminars in Immunology. 24 (4): 293–300. doi:10.1016/j.smim.2012.05.005. PMC 3422444. PMID 22703640.
- ↑ Hsieh P (September 2012). "DNA mismatch repair: Dr. Jekyll and Mr. Hyde?". Molecular Cell. 47 (5): 665–6. doi:10.1016/j.molcel.2012.08.020. PMC 3457060. PMID 22980456.
- ↑ 28.0 28.1 Supek F, Lehner B (July 2017). "क्लस्टर्ड म्यूटेशन सिग्नेचर से पता चलता है कि एरर-प्रोन डीएनए रिपेयर टार्गेट म्यूटेशन टू एक्टिव जीन". Cell. 170 (3): 534–547.e23. doi:10.1016/j.cell.2017.07.003. hdl:10230/35343. PMID 28753428.
- ↑ Tuna M, Amos CI (November 2013). "कैंसर में जीनोमिक अनुक्रमण". Cancer Letters. 340 (2): 161–70. doi:10.1016/j.canlet.2012.11.004. PMC 3622788. PMID 23178448.
- ↑ Supek F, Lehner B (May 2015). "डिफरेंशियल डीएनए मिसमैच रिपेयर मानव जीनोम में उत्परिवर्तन दर भिन्नता को रेखांकित करता है". Nature. 521 (7550): 81–4. Bibcode:2015Natur.521...81S. doi:10.1038/nature14173. PMC 4425546. PMID 25707793.
- ↑ Schuster-Böckler B, Lehner B (August 2012). "मानव कैंसर कोशिकाओं में क्षेत्रीय उत्परिवर्तन दर पर क्रोमैटिन संगठन एक बड़ा प्रभाव है". Nature. 488 (7412): 504–7. Bibcode:2012Natur.488..504S. doi:10.1038/nature11273. PMID 22820252. S2CID 205229634.
- ↑ Li F, Mao G, Tong D, Huang J, Gu L, Yang W, Li GM (April 2013). "The histone mark H3K36me3 regulates human DNA mismatch repair through its interaction with MutSα". Cell. 153 (3): 590–600. doi:10.1016/j.cell.2013.03.025. PMC 3641580. PMID 23622243.
- ↑ Jiang Z, Hu J, Li X, Jiang Y, Zhou W, Lu D (December 2006). "Expression analyses of 27 DNA repair genes in astrocytoma by TaqMan low-density array". Neuroscience Letters. 409 (2): 112–7. doi:10.1016/j.neulet.2006.09.038. PMID 17034947. S2CID 54278905.
- ↑ Kitajima Y, Miyazaki K, Matsukura S, Tanaka M, Sekiguchi M (2003). "Loss of expression of DNA repair enzymes MGMT, hMLH1, and hMSH2 during tumor progression in gastric cancer". Gastric Cancer. 6 (2): 86–95. doi:10.1007/s10120-003-0213-z. PMID 12861399.
- ↑ Narayanan L, Fritzell JA, Baker SM, Liskay RM, Glazer PM (April 1997). "Elevated levels of mutation in multiple tissues of mice deficient in the DNA mismatch repair gene Pms2". Proceedings of the National Academy of Sciences of the United States of America. 94 (7): 3122–7. Bibcode:1997PNAS...94.3122N. doi:10.1073/pnas.94.7.3122. PMC 20332. PMID 9096356.
अग्रिम पठन
- Hsieh P, Yamane K (2008). "DNA mismatch repair: molecular mechanism, cancer, and ageing". Mechanisms of Ageing and Development. 129 (7–8): 391–407. doi:10.1016/j.mad.2008.02.012. PMC 2574955. PMID 18406444.
- Iyer RR, Pluciennik A, Burdett V, Modrich PL (February 2006). "DNA mismatch repair: functions and mechanisms". Chemical Reviews. 106 (2): 302–23. doi:10.1021/cr0404794. PMID 16464007.
- Joseph N, Duppatla V, Rao DN (2006). Prokaryotic DNA mismatch repair. pp. 1–49. doi:10.1016/S0079-6603(06)81001-9. ISBN 9780125400817. PMID 16891168.
{{cite book}}:|journal=ignored (help) - Yang W (August 2000). "Structure and function of mismatch repair proteins". Mutation Research. 460 (3–4): 245–56. doi:10.1016/s0921-8777(00)00030-6. PMID 10946232.
- Griffiths JF, Gilbert WM, Lewontin RC, Wessler SR, Suzuki DT, Miller JH (2004). An introduction to genetic analysis (8th ed.). New York, NY: Freeman. ISBN 978-0-7167-4939-4.
- Kunkel TA, Erie DA (2005). "DNA mismatch repair". Annual Review of Biochemistry. 74: 681–710. doi:10.1146/annurev.biochem.74.082803.133243. PMID 15952900.
- Friedberg EC, Walker GC, Siede W, Wood RD, Schultz RA, Ellenberger (2005). DNA repair and mutagenesis (2nd ed.). Washington, D.C.: ASM Press. ISBN 978-1-55581-319-2.
बाहरी संबंध
- DNA Repair Archived 2018-02-12 at the Wayback Machine
- DNA+Mismatch+Repair at the US National Library of Medicine Medical Subject Headings (MeSH)