एक्स्ट्राक्रोमोसोमल डीएनए
एक्स्ट्राक्रोमोसोमल डीएनए (संक्षेप में ईसीडीएनए) कोई भी डीएनए है जो किसी कोशिका के केंद्रक के अंदर या बाहर गुणसूत्रों से पाया जाता है। एक व्यक्तिगत जीनोम में अधिकांश डीएनए नाभिक में मौजूद गुणसूत्रों में पाया जाता है। एक्स्ट्राक्रोमोसोमल डीएनए के कई रूप मौजूद हैं, और, इनमें से कुछ महत्वपूर्ण जैविक कार्य करते हैं,[1] वे कैंसर जैसी बीमारियों में भी भूमिका निभा सकते हैं।[2][3][4]
प्रोकैरियोट्स में, नॉनवायरल एक्स्ट्राक्रोमोसोमल डीएनए मुख्य रूप से प्लास्मिड में पाया जाता है, जबकि यूकेरियोट्स में एक्स्ट्राक्रोमोसोमल डीएनए मुख्य रूप से ऑर्गेनेल में पाया जाता है।[1] माइटोकॉन्ड्रियल डीएनए यूकेरियोट्स में इस एक्स्ट्राक्रोमोसोमल डीएनए का एक मुख्य स्रोत है।[5] तथ्य यह है कि इस अंगक में अपना स्वयं का डीएनए होता है जो इस परिकल्पना का समर्थन करता है कि माइटोकॉन्ड्रिया की उत्पत्ति पैतृक यूकेरियोटिक कोशिकाओं से घिरे जीवाणु कोशिकाओं के रूप में हुई थी।[6] एक्स्ट्राक्रोमोसोमल डीएनए का उपयोग अक्सर प्रतिकृति के अनुसंधान में किया जाता है क्योंकि इसे पहचानना और अलग करना आसान होता है।[1]
हालाँकि एक्स्ट्राक्रोमोसोमल सर्कुलर डीएनए (ईसीसीडीएनए) सामान्य यूकेरियोटिक कोशिकाओं में पाया जाता है, एक्स्ट्राक्रोमोसोमल डीएनए (ईसीडीएनए) एक विशिष्ट इकाई है जिसे कैंसर कोशिकाओं के नाभिक में पहचाना गया है और ड्राइवर ऑन्कोजीन की कई प्रतियां ले जाने के लिए दिखाया गया है।[7][8][3] ईसीडीएनए को जीन प्रवर्धन का एक प्राथमिक तंत्र माना जाता है, जिसके परिणामस्वरूप चालक ऑन्कोजीन की कई प्रतियां और बहुत आक्रामक कैंसर होते हैं।
साइटोप्लाज्म में एक्स्ट्राक्रोमोसोमल डीएनए संरचनात्मक रूप से परमाणु डीएनए से भिन्न पाया गया है। साइटोप्लाज्मिक डीएनए नाभिक के भीतर पाए जाने वाले डीएनए की तुलना में कम मिथाइलेटेड होता है। यह भी पुष्टि की गई कि साइटोप्लाज्मिक डीएनए के अनुक्रम एक ही जीव में परमाणु डीएनए से भिन्न थे, जिससे पता चलता है कि साइटोप्लाज्मिक डीएनए केवल परमाणु डीएनए के टुकड़े नहीं हैं।[9] कैंसर कोशिकाओं में, ईसीडीएनए को मुख्य रूप से नाभिक से पृथक दिखाया गया है ([2] में समीक्षा की गई है)।
कोशिकाओं में नाभिक के बाहर पाए जाने वाले डीएनए के अलावा, वायरल जीनोम द्वारा संक्रमण भी एक्स्ट्राक्रोमोसोमल डीएनए का एक उदाहरण प्रदान करता है।
प्रोकैरियोटिक
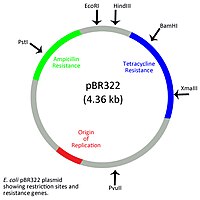
यद्यपि प्रोकैरियोटिक जीवों में यूकेरियोट्स की तरह एक झिल्ली-बद्ध नाभिक नहीं होता है, उनमें एक न्यूक्लियॉइड क्षेत्र होता है जिसमें मुख्य गुणसूत्र पाया जाता है। एक्स्ट्राक्रोमोसोमल डीएनए न्यूक्लियॉइड क्षेत्र के बाहर गोलाकार या रैखिक प्लास्मिड के रूप में प्रोकैरियोट्स में मौजूद होता है। बैक्टीरियल प्लास्मिड आम तौर पर छोटे अनुक्रम होते हैं, जिनमें 1 से कुछ सौ किलोबेस (केबी) खंड होते हैं, और इसमें प्रतिकृति की उत्पत्ति होती है जो प्लास्मिड को बैक्टीरिया गुणसूत्र से स्वतंत्र रूप से दोहराने की अनुमति देती है।[10] किसी कोशिका के भीतर किसी विशेष प्लास्मिड की कुल संख्या को प्रतिलिपि संख्या कहा जाता है और यह प्रति कोशिका दो प्रतियों से लेकर प्रति कोशिका कई सौ प्रतियों तक हो सकती है।[11] सर्कुलर बैक्टीरियल प्लास्मिड को प्लास्मिड पर एन्कोड किए गए जीन द्वारा प्रदान किए जाने वाले विशेष कार्यों के अनुसार वर्गीकृत किया जाता है। फर्टिलिटी प्लास्मिड, या एफ प्लास्मिड, संयुग्मन होने की अनुमति देते हैं जबकि प्रतिरोध प्लास्मिड, या आर प्लास्मिड, में ऐसे जीन होते हैं जो विभिन्न प्रकार के एंटीबायोटिक दवाओं जैसे एम्पीसिलीन और टेट्रासाइक्लिन के प्रति प्रतिरोध व्यक्त करते हैं। विषाणु प्लास्मिड में जीवाणुओं को रोगजनक बनने के लिए आवश्यक आनुवंशिक तत्व होते हैं। डिग्रेडेटिव प्लास्मिड में ऐसे जीन होते हैं जो बैक्टीरिया को विभिन्न प्रकार के पदार्थों जैसे कि सुगंधित यौगिकों और ज़ेनोबायोटिक्स को नष्ट करने की अनुमति देते हैं।[12] बैक्टीरियल प्लास्मिड रंगद्रव्य उत्पादन, नाइट्रोजन स्थिरीकरण और भारी धातुओं के प्रतिरोध में भी कार्य कर सकते हैं।[13]
स्वाभाविक रूप से उत्पन्न होने वाले वृत्ताकार प्लास्मिड को बहुप्रतिरोध जीनों और कई अद्वितीय प्रतिबंध साइटों को समाहित करने के लिए संशोधित किया जा सकता है, जिससे वे जैव प्रौद्योगिकी में क्लोनिंग वेक्टर के रूप में मूल्यवान उपकरण बन जाते हैं।[10] सर्कुलर बैक्टीरियल प्लास्मिड भी डीएनए टीकाकरण के उत्पादन का आधार हैं। प्लास्मिड डीएनए टीके जेनेटिक इंजीनियरिंग हैं जिसमें एक जीन होता है जो एक रोगजनक वायरस, जीवाणु या अन्य परजीवी द्वारा उत्पादित एंटीजन या प्रोटीन के लिए एन्कोड करता है।[14] एक बार मेजबान में वितरित होने के बाद, प्लाज्मिड जीन के उत्पाद मेजबान की सहज प्रतिरक्षा और अनुकूली प्रतिरक्षा दोनों को उत्तेजित करेंगे। मेजबान से प्रतिरक्षा प्रतिक्रिया को बढ़ाने के लिए प्लास्मिड अक्सर डिलीवरी से पहले किसी प्रकार के सहायक के साथ लेपित होते हैं।[15]
स्पिरोचैट की कई प्रजातियों में लीनियर बैक्टीरियल प्लास्मिड की पहचान की गई है, जिसमें जीनस बोरेलिया के सदस्य (जिससे लाइम रोग के लिए जिम्मेदार रोगज़नक़ शामिल है), ग्राम-ऋणात्मक जीवाणु की कई प्रजातियाँ जीनस स्ट्रेप्टोमाइसेस के मिट्टी के बैक्टीरिया और ग्राम में- ऋणात्मक जीवाणु प्रजाति थियोबैसिलस वर्सुटस, एक जीवाणु जो सल्फर को ऑक्सीकरण करता है। प्रोकैरियोट्स के रैखिक प्लास्मिड या तो एक हेयरपिन लूप या डीएनए अणु के टेलोमेर सिरों से जुड़े एक सहसंयोजक बंधन प्रोटीन से युक्त पाए जाते हैं। बोरेलिया बैक्टीरिया के एडेनिन-थाइमिन समृद्ध हेयरपिन लूप का आकार 5 किलोबेस जोड़े (केबी) से लेकर 200 केबी से अधिक तक होता है।[16] और बैक्टीरिया पर प्रमुख सतह प्रोटीन, या एंटीजन के एक समूह के उत्पादन के लिए जिम्मेदार जीन होते हैं जो इसे अपने संक्रमित मेजबान की प्रतिरक्षा प्रतिक्रिया से बचने की अनुमति देते हैं।[17] रैखिक प्लास्मिड जिसमें एक प्रोटीन होता है जो डीएनए स्ट्रैंड के 5' छोर से सहसंयोजक रूप से जुड़ा होता है, इनवर्ट्रोन के रूप में जाना जाता है और इसका आकार 9 केबी से लेकर 600 केबी से अधिक हो सकता है जिसमें उल्टे दोहराव शामिल होते हैं।[16] एक सहसंयोजक प्रोटीन के साथ रैखिक प्लास्मिड जीवाणु जीवाणु संयुग्मन और प्लास्मिड के जीनोम में एकीकरण के साथ सहायता कर सकते हैं। इस प्रकार के रैखिक प्लास्मिड एक्स्ट्राक्रोमोसोमल डीएनए के सबसे बड़े वर्ग का प्रतिनिधित्व करते हैं क्योंकि वे न केवल कुछ बैक्टीरिया कोशिकाओं में मौजूद होते हैं, बल्कि यूकेरियोटिक कोशिकाओं में पाए जाने वाले सभी रैखिक एक्स्ट्राक्रोमोसोमल डीएनए अणु भी इस इन्वर्ट्रोन संरचना को 5' छोर से जुड़े प्रोटीन के साथ लेते हैं।[16][17]
लंबे, रैखिक "बोर्ग" जो आर्कियोन की एक प्रजाति के साथ सह-घटित होते हैं - जो उन्हें होस्ट कर सकते हैं और उनके कई जीनों को साझा कर सकते हैं - एक्स्ट्राक्रोमोसोमल डीएनए संरचनाओं का एक अज्ञात रूप हो सकता है।[18][19]
यूकेरियोटिक
माइटोकॉन्ड्रियल
यूकेरियोटिक कोशिकाओं में मौजूद माइटोकॉन्ड्रिया में माइटोकॉन्ड्रियल मैट्रिक्स में माइटोकॉन्ड्रियल डीएनए (एमटीडीएनए) की कई प्रतियां होती हैं।[20] मनुष्यों सहित बहुकोशिकीय जानवरों में, गोलाकार एमटीडीएनए क्रोमोसोम में 13 जीन होते हैं जो प्रोटीन को एनकोड करते हैं जो इलेक्ट्रॉन परिवहन श्रृंखला का हिस्सा होते हैं और अन्य माइटोकॉन्ड्रियल प्रोटीन के लिए 24 जीन होते हैं; ये जीन 2 आरआरएनए जीन और 22 टीआरएनए जीन में टूट जाते हैं।[21] एक जानवर एमटीडीएनए प्लास्मिड का आकार लगभग 16.6 केबी है और, हालांकि इसमें टीआरएनए और एमआरएनए संश्लेषण के लिए जीन शामिल हैं, परमाणु जीन द्वारा कोडित प्रोटीन को अभी भी एमटीडीएनए को दोहराने या माइटोकॉन्ड्रियल प्रोटीन का अनुवाद करने के लिए आवश्यक है।[22] माइटोकॉन्ड्रियल गुणसूत्र का केवल एक क्षेत्र है जिसमें कोडिंग अनुक्रम नहीं होता है, 1 केबी क्षेत्र जिसे डी-लूप के रूप में जाना जाता है, जिससे परमाणु नियामक प्रोटीन बंधते हैं।[21] प्रति माइटोकॉन्ड्रिया में एमटीडीएनए अणुओं की संख्या प्रजातियों से प्रजातियों के साथ-साथ विभिन्न ऊर्जा मांगों वाली कोशिकाओं के बीच भिन्न होती है। उदाहरण के लिए, मांसपेशियों और यकृत कोशिकाओं में रक्त और त्वचा कोशिकाओं की तुलना में प्रति माइटोकॉन्ड्रिया में एमटीडीएनए की अधिक प्रतियां होती हैं।[22] माइटोकॉन्ड्रियल आंतरिक झिल्ली के भीतर इलेक्ट्रॉन परिवहन श्रृंखला की निकटता और प्रतिक्रियाशील ऑक्सीजन प्रजातियों (आरओएस) के उत्पादन के कारण, और इस तथ्य के कारण कि एमटीडीएनए अणु हिस्टोन द्वारा बाध्य या संरक्षित नहीं है, एमटीडीएनए परमाणु डीएनए की तुलना में डीएनए क्षति के प्रति अधिक संवेदनशील है।[23] ऐसे मामलों में जहां एमटीडीएनए क्षति होती है, डीएनए को या तो बेस एक्सिशन रिपेयर पाथवे के जरिए ठीक किया जा सकता है, या क्षतिग्रस्त एमटीडीएनए अणु को नष्ट कर दिया जाता है (माइटोकॉन्ड्रियन को नुकसान पहुंचाए बिना क्योंकि प्रति माइटोकॉन्ड्रियन में एमटीडीएनए की कई प्रतियां होती हैं)।[24]
मानक आनुवंशिक कोड जिसके द्वारा परमाणु जीन का अनुवाद किया जाता है, सार्वभौमिक है, जिसका अर्थ है कि डीएनए का प्रत्येक 3-आधार अनुक्रम एक ही अमीनो एसिड के लिए कोड करता है, चाहे डीएनए किसी भी प्रजाति से आता हो। हालाँकि, यह कोड काफी सार्वभौमिक है और कवक, जानवरों, प्रोटिस्ट और पौधों के माइटोकॉन्ड्रियल डीएनए में थोड़ा अलग है।[20] जबकि इन जीवों के एमटीडीएनए में अधिकांश 3-बेस अनुक्रम (कोडन) परमाणु आनुवंशिक कोड के समान अमीनो एसिड के लिए कोड करते हैं, कुछ भिन्न होते हैं।
| जेनेटिक कोड | अनुवाद सारणी | डीएनए कोडन शामिल | आरएनए कोडन शामिल | इस कोड के साथ अनुवाद | यूनिवर्सल कोड से तुलना |
|---|---|---|---|---|---|
| कशेरुकी माइटोकॉन्ड्रियल | 2 | AGA
|
AGA
|
Ter (*)
|
Arg (R)
|
AGG
|
AGG
|
Ter (*)
|
Arg (R)
| ||
ATA
|
AUA
|
Met (M)
|
Ile (I)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
| यीस्ट माइटोकॉन्ड्रियल | 3 | ATA
|
AUA
|
Met (M)
|
Ile (I)
|
CTT
|
CUU
|
Thr (T)
|
Leu (L)
| ||
CTC
|
CUC
|
Thr (T)
|
Leu (L)
| ||
CTA
|
CUA
|
Thr (T)
|
Leu (L)
| ||
CTG
|
CUG
|
Thr (T)
|
Leu (L)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
CGA
|
CGA
|
absent
|
Arg (R)
| ||
CGC
|
CGC
|
absent
|
Arg (R)
| ||
| मोल्ड, प्रोटोजोअन, और सीलेन्टरेट माइटोकॉन्ड्रियल | 4 and 7 | TGA
|
UGA
|
Trp (W)
|
Ter (*)
|
| अकशेरूकी माइटोकॉन्ड्रियल | 5 | AGA
|
AGA
|
Ser (S)
|
Arg (R)
|
AGG
|
AGG
|
Ser (S)
|
Arg (R)
| ||
ATA
|
AUA
|
Met (M)
|
Ile (I)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
| एकिनोडर्म और फ्लैटवर्म माइटोकॉन्ड्रियल | 9 | AAA
|
AAA
|
Asn (N)
|
Lys (K)
|
AGA
|
AGA
|
Ser (S)
|
Arg (R)
| ||
AGG
|
AGG
|
Ser (S)
|
Arg (R)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
| एस्किडियन माइटोकॉन्ड्रियल | 13 | AGA
|
AGA
|
Gly (G)
|
Arg (R)
|
AGG
|
AGG
|
Gly (G)
|
Arg (R)
| ||
ATA
|
AUA
|
Met (M)
|
Ile (I)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
| वैकल्पिक फ़्लैटवॉर्म माइटोकॉन्ड्रियल | 14 | AAA
|
AAA
|
Asn (N)
|
Lys (K)
|
AGA
|
AGA
|
Ser (S)
|
Arg (R)
| ||
AGG
|
AGG
|
Ser (S)
|
Arg (R)
| ||
TAA
|
UAA
|
Tyr (Y)
|
Ter (*)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
| ||
| क्लोरोफाइशियन माइटोकॉन्ड्रियल | 16 | TAG
|
UAG
|
Leu (L)
|
Ter (*)
|
| ट्रेमेटोड माइटोकॉन्ड्रियल | 21 | TGA
|
UGA
|
Trp (W)
|
Ter (*)
|
ATA
|
AUA
|
Met (M)
|
Ile (I)
| ||
AGA
|
AGA
|
Ser (S)
|
Arg (R)
| ||
AGG
|
AGG
|
Ser (S)
|
Arg (R)
| ||
AAA
|
AAA
|
Asn (N)
|
Lys (K)
| ||
| सीनेडेसमस ओब्लिकस माइटोकॉन्ड्रियल | 22 | TCA
|
UCA
|
Ter (*)
|
Ser (S)
|
TAG
|
UAG
|
Leu (L)
|
Ter (*)
| ||
| थ्रोस्टोचिट्रियम माइटोकॉन्ड्रियल | 23 | TTA
|
UUA
|
Ter (*)
|
Leu (L)
|
| टेरोब्रैन्चिया माइटोकॉन्ड्रियल | 24 | AGA
|
AGA
|
Ser (S)
|
Arg (R)
|
AGG
|
AGG
|
Lys (K)
|
Arg (R)
| ||
TGA
|
UGA
|
Trp (W)
|
Ter (*)
|
| अमीनो एसिड के जैव रासायनिक गुण | अध्रुवीय | ध्रुवीय | मूलभूत | एसिडिक | समापन: कोडन बंद करो |
ऐसा माना जाता है कि कोडिंग अंतर ट्रांसफर आरएनए में रासायनिक संशोधनों का परिणाम है जो एमटीडीएनए अनुक्रमों को लिखने के परिणामस्वरूप उत्पन्न मैसेंजर आरएनए के साथ बातचीत करते हैं।[25]
क्लोरोप्लास्ट
यूकेरियोटिक क्लोरोप्लास्ट, साथ ही अन्य पौधों के प्लास्टिड्स में एक्स्ट्राक्रोमोसोमल डीएनए अणु भी होते हैं। अधिकांश क्लोरोप्लास्ट अपनी सभी आनुवंशिक सामग्री को एक ही चक्राकार गुणसूत्र में रखते हैं, हालाँकि कुछ प्रजातियों में कई छोटे चक्राकार प्लास्मिड के प्रमाण मिलते हैं।[26][27][28] एक हालिया सिद्धांत जो अंगूठी के आकार के क्लोरोप्लास्ट डीएनए (सीपीडीएनए) के वर्तमान मानक मॉडल पर सवाल उठाता है, सुझाव देता है कि सीपीडीएनए आमतौर पर एक रैखिक आकार ले सकता है।[29] सीपीडीएनए के एक अणु में 100-200 जीन हो सकते हैं[30] और विभिन्न प्रजातियों के आकार में भिन्नता होती है। उच्च पौधों में सीपीडीएनए का आकार लगभग 120-160 केबी होता है।[20] एमआरएनए के लिए सीपीडीएनए कोड पर पाए जाने वाले जीन प्रकाश संश्लेषक मार्ग के आवश्यक घटकों के उत्पादन के साथ-साथ टीआरएनए, आरआरएनए, आरएनए पोलीमरेज़ सबयूनिट और राइबोसोमल प्रोटीन सबयूनिट के लिए कोडिंग के लिए जिम्मेदार हैं।[31] एमटीडीएनए की तरह, सीपीडीएनए पूरी तरह से स्वायत्त नहीं है और क्लोरोप्लास्ट प्रोटीन की प्रतिकृति और उत्पादन के लिए परमाणु जीन उत्पादों पर निर्भर करता है। क्लोरोप्लास्ट में सीपीडीएनए की कई प्रतियां होती हैं और संख्या न केवल प्रजातियों से प्रजातियों या कोशिका प्रकार से कोशिका प्रकार तक भिन्न हो सकती है, बल्कि कोशिका की आयु और विकास के चरण के आधार पर एक कोशिका के भीतर भी भिन्न हो सकती है। उदाहरण के लिए, युवा कोशिकाओं के क्लोरोप्लास्ट में सीपीडीएनए सामग्री, विकास के शुरुआती चरणों के दौरान, जहां क्लोरोप्लास्ट अस्पष्ट प्रोप्लास्टिड के रूप में होते हैं, उस समय मौजूद कोशिकाओं की तुलना में बहुत अधिक होती है जब वह कोशिका परिपक्व होती है और विस्तारित होती है, जिसमें पूरी तरह से परिपक्व प्लास्टिड होते हैं।[32]
सर्कुलर
एक्स्ट्राक्रोमोसोमल सर्कुलर डीएनए (ईसीसीडीएनए) सभी यूकेरियोटिक कोशिकाओं में मौजूद होते हैं, आमतौर पर जीनोमिक डीएनए से प्राप्त होते हैं, और क्रोमोसोम के कोडिंग और गैर-कोडिंग दोनों क्षेत्रों में पाए जाने वाले डीएनए के दोहराव वाले अनुक्रमों से बने होते हैं। ईसीसीडीएनए का आकार 2000 से कम बेस जोड़े से लेकर 20,000 से अधिक बेस जोड़े तक हो सकता है।[33] पौधों में, ईसीसीडीएनए में उन अनुक्रमों के समान बार-बार अनुक्रम होते हैं जो गुणसूत्रों के सेंट्रोमेरिक क्षेत्रों और दोहराए जाने वाले उपग्रह डीएनए में पाए जाते हैं।[34] जानवरों में, ईसीसीडीएनए अणुओं में दोहराव वाले अनुक्रम पाए जाते हैं जो उपग्रह डीएनए, 5S राइबोसोमल डीएनए और टेलोमेयर डीएनए में देखे जाते हैं।[33] कुछ जीव, जैसे कि यीस्ट, ईसीसीडीएनए का उत्पादन करने के लिए क्रोमोसोमल डीएनए प्रतिकृति पर निर्भर होते हैं[34] जबकि ईसीसीडीएनए का गठन अन्य जीवों, जैसे स्तनधारियों, में प्रतिकृति प्रक्रिया से स्वतंत्र रूप से हो सकता है।[35] ईसीसीडीएनए के कार्य का व्यापक रूप से अध्ययन नहीं किया गया है, लेकिन यह प्रस्तावित किया गया है कि जीनोमिक डीएनए अनुक्रमों से ईसीसीडीएनए तत्वों का उत्पादन यूकेरियोटिक जीनोम की प्लास्टिसिटी को बढ़ाता है और जीनोम स्थिरता, कोशिका उम्र बढ़ने और गुणसूत्रों के विकास को प्रभावित कर सकता है।[36]
एक विशिष्ट प्रकार का एक्स्ट्राक्रोमोसोमल डीएनए, जिसे ईसीडीएनए के रूप में जाना जाता है, आमतौर पर मानव कैंसर कोशिकाओं में देखा जाता है।[2][3][4] कैंसर कोशिकाओं में पाए जाने वाले ईसीडीएनए में एक या एक से अधिक जीन होते हैं जो चयनात्मक लाभ प्रदान करते हैं। ईसीडीएनए, ईसीसीडीएनए से बहुत बड़े होते हैं और प्रकाश माइक्रोस्कोपी द्वारा दृश्यमान होते हैं। कैंसर में ईसीडीएनए आम तौर पर 1-3 एमबी और उससे अधिक आकार का होता है।[2] मानव कैंसर कोशिकाओं के नाभिक में बड़े ईसीडीएनए अणु पाए गए हैं और यह दिखाया गया है कि वे चालक ऑन्कोजीन की कई प्रतियां ले जाते हैं, जो ट्यूमर कोशिकाओं में स्थानांतरित हो जाते हैं। इस साक्ष्य के आधार पर यह माना जाता है कि ईसीडीएनए कैंसर के विकास में योगदान देता है।
विशिष्ट उपकरण मौजूद हैं जो ईसीडीएनए की पहचान करने की अनुमति देते हैं, जैसे
- पॉल मिशेल और विनीत बाफना द्वारा विकसित सॉफ्टवेयर जो सूक्ष्म छवियों में ईसीडीएनए को पहचानने की अनुमति देता है।
- "सर्कल-सेक, कोशिकाओं से ईसीडीएनए को भौतिक रूप से अलग करने, एंजाइमों के साथ किसी भी शेष रैखिक डीएनए को हटाने और जो गोलाकार डीएनए रहता है उसे अनुक्रमित करने की एक विधि", कोपेनहेगन विश्वविद्यालय में बिरगिट रेगेनबर्ग और उनकी टीम द्वारा विकसित की गई।[37]
वायरल
वायरल डीएनए एक्स्ट्राक्रोमोसोमल डीएनए का उदाहरण है। वायरस के विकास और उत्परिवर्तन को समझने के लिए वायरल जीनोम को समझना बहुत महत्वपूर्ण है।[38] कुछ वायरस, जैसे कि एचआईवी और ऑन्कोजेनिक वायरस, अपने स्वयं के डीएनए को मेजबान कोशिका के जीनोम में शामिल करते हैं।[39] वायरल जीनोम सिंगल स्ट्रैंडेड डीएनए (एसएसडीएनए), डबल स्ट्रैंडेड डीएनए (डीएसडीएनए) से बना हो सकता है और रैखिक और गोलाकार दोनों रूपों में पाया जा सकता है।[40]
एक्स्ट्राक्रोमोसोमल डीएनए के रूप में गठित वायरस के संक्रमण का एक उदाहरण ह्यूमन पेपिलोमावायरस (एचपीवी) है। एचपीवी डीएनए जीनोम प्रतिकृति के तीन अलग-अलग चरणों से होकर गुजरता है: स्थापना, रखरखाव और प्रवर्धन। एचपीवी एनोजिनिटल ट्रैक्ट और मौखिक गुहा में उपकला कोशिकाओं को संक्रमित करता है। आम तौर पर, एचपीवी का पता लगाया जाता है और प्रतिरक्षा प्रणाली द्वारा उसे ख़त्म कर दिया जाता है। वायरल डीएनए की पहचान प्रतिरक्षा प्रतिक्रियाओं का एक महत्वपूर्ण भाग है। इस वायरस के बने रहने के लिए, कोशिका विभाजन के दौरान वृत्ताकार जीनोम को दोहराया जाना चाहिए और विरासत में मिला होना चाहिए।[41]
मेजबान कोशिका द्वारा अनुभूति
कोशिकाएँ विदेशी साइटोप्लास्मिक डीएनए को पहचान सकती हैं। पहचान के मार्गों को समझने से रोगों की रोकथाम और उपचार पर प्रभाव पड़ता है।[42] कोशिकाओं में सेंसर होते हैं जो विशेष रूप से वायरल डीएनए जैसे टोल-लाइक रिसेप्टर (टीएलआर) मार्ग को पहचान सकते हैं।[43]
टोल पाथवे को, सबसे पहले कीड़ों में, एक ऐसे मार्ग के रूप में मान्यता दी गई थी जो कुछ प्रकार की कोशिकाओं को विभिन्न प्रकार के बैक्टीरिया या वायरल जीनोम और पीएमपीएस (रोगज़नक़ से जुड़े आणविक पैटर्न) का पता लगाने में सक्षम सेंसर के रूप में कार्य करने की अनुमति देता है। पीएएमपी को जन्मजात प्रतिरक्षा सिग्नलिंग के शक्तिशाली सक्रियकर्ता के रूप में जाना जाता है। लगभग 10 मानव टोल-लाइक रिसेप्टर्स (टीएलआर) हैं। मनुष्यों में अलग-अलग टीएलआर अलग-अलग पीएएमपीएस का पता लगाते हैं: टीएलआर4 द्वारा लिपोपॉलीसेकेराइड, टीएलआर3 द्वारा वायरल डीएसआरएनए, टीएलआर7/टीएलआर8 द्वारा वायरल एसएसआरएनए, टीएलआर9 द्वारा वायरल या बैक्टीरियल अनमिथाइलेटेड डीएनए। टीएलआर9 आमतौर पर बैक्टीरिया और वायरस में पाए जाने वाले सीपीजी डीएनए का पता लगाने और आईएफएन (प्रकार I इंटरफेरॉन) और अन्य साइटोकिन्स का उत्पादन शुरू करने के लिए विकसित हुआ है।[43]
वंशानुक्रम
एक्स्ट्राक्रोमोसोमल डीएनए का वंशानुक्रम गुणसूत्रों में पाए जाने वाले परमाणु डीएनए के वंशानुक्रम से भिन्न होता है। क्रोमोसोम के विपरीत, ईसीडीएनए में सेंट्रोमियर नहीं होते हैं और इसलिए यह एक गैर-मेंडेलियन वंशानुक्रम पैटर्न प्रदर्शित करता है जो विषम कोशिका आबादी को जन्म देता है। मनुष्यों में, वस्तुतः संपूर्ण कोशिकाद्रव्य माँ के अंडे से विरासत में मिलता है।[44] इस कारण से, एमटीडीएनए समेत ऑर्गेनेल डीएनए मां से विरासत में मिला है। एमटीडीएनए या अन्य साइटोप्लाज्मिक डीएनए में उत्परिवर्तन भी मां से विरासत में मिलेगा। यह एकतरफा विरासत गैर-मेंडेलियन विरासत का एक उदाहरण है। पौधे एकपक्षीय एमटीडीएनए वंशानुक्रम भी दर्शाते हैं। अधिकांश पौधों को एमटीडीएनए मातृ रूप से विरासत में मिलता है, जिसमें एक उल्लेखनीय अपवाद रेडवुड सिकोइया सेपरविरेन्स है जो एमटीडीएनए पैतृक रूप से विरासत में मिलता है।[45]
ऐसे दो सिद्धांत हैं कि क्यों पैतृक एमटीडीएनए शायद ही कभी संतानों में प्रसारित होता है। एक तो बस यह तथ्य है कि पैतृक एमटीडीएनए मातृ एमटीडीएनए की तुलना में इतनी कम सांद्रता पर है और इस प्रकार संतानों में इसका पता नहीं लगाया जा सकता है। दूसरा, अधिक जटिल सिद्धांत, वंशानुक्रम को रोकने के लिए पैतृक एमटीडीएनए का पाचन शामिल है। यह सिद्धांत दिया गया है कि एमटीडीएनए की एकतरफा विरासत, जिसमें उच्च उत्परिवर्तन दर है, साइटोप्लाज्मिक डीएनए की होमोप्लाज्मी को बनाए रखने के लिए एक तंत्र हो सकती है।[45]
नैदानिक महत्व
कभी-कभी ईई कहा जाता है, एक्स्ट्राक्रोमोसोमल तत्व, यूकेरियोट्स में जीनोमिक अस्थिरता से जुड़े होते हैं। छोटे पॉलीडिस्पर्स्ड डीएनए (एसपीसीडीएनए), एक प्रकार का ईसीसीडीएनए, आमतौर पर जीनोम अस्थिरता के संयोजन में पाए जाते हैं। SpcDNAs उपग्रह डीएनए, रेट्रोवायरस-जैसे डीएनए तत्वों और जीनोम में ट्रांसपोजेबल तत्वों जैसे दोहराव वाले अनुक्रमों से प्राप्त होते हैं। उन्हें जीन पुनर्व्यवस्था के उत्पाद माना जाता है।
कैंसर में पाए जाने वाले एक्स्ट्राक्रोमोसोमल डीएनए (ईसीडीएनए) को ऐतिहासिक रूप से डबल मिनट क्रोमोसोम (डीएम) कहा जाता है, जो हल्की माइक्रोस्कोपी के तहत युग्मित क्रोमेटिन निकायों के रूप में मौजूद होते हैं। डबल मिनट क्रोमोसोम एकल निकायों सहित ईसीडीएनए के कैंसर युक्त स्पेक्ट्रम के ~ 30% का प्रतिनिधित्व करते हैं और एकल निकायों के रूप में समान जीन सामग्री शामिल पाए गए हैं।[3]ईसीडीएनए संकेतन कैंसर कोशिकाओं में पाए जाने वाले बड़े, ओंकोजीन युक्त, एक्स्ट्राक्रोमोसोमल डीएनए के सभी रूपों को शामिल करता है। इस प्रकार का ईसीडीएनए आमतौर पर विभिन्न हिस्टोलोजी के कैंसर कोशिकाओं में देखा जाता है, लेकिन वास्तव में कभी भी सामान्य कोशिकाओं में नहीं होता है।[3]माना जाता है कि ईसीडीएनए गुणसूत्रों में डबल स्ट्रैंड टूटना के माध्यम से या जीव में डीएनए की अति-प्रतिकृति के माध्यम से उत्पन्न होता है। अध्ययनों से पता चलता है कि कैंसर और अन्य जीनोमिक अस्थिरता के मामलों में, ईई के उच्च स्तर देखे जा सकते हैं।[5]
माइटोकॉन्ड्रियल डीएनए विभिन्न तरीकों से रोग की शुरुआत में भूमिका निभा सकता है। एमटीडीएनए के बिंदु उत्परिवर्तन या वैकल्पिक जीन व्यवस्था को कई बीमारियों से जोड़ा गया है जो हृदय, केंद्रीय तंत्रिका तंत्र, अंतःस्रावी तंत्र, जठरांत्र संबंधी मार्ग, आंख और गुर्दे को प्रभावित करते हैं।[21] माइटोकॉन्ड्रिया में मौजूद एमटीडीएनए की मात्रा के नुकसान से माइटोकॉन्ड्रियल कमी सिंड्रोम (एमडीडी) के रूप में जाने जाने वाले रोगों का एक पूरा उपसमूह हो सकता है जो यकृत, केंद्रीय और परिधीय तंत्रिका तंत्र प्रणाली, चिकनी मांसपेशियों और मनुष्यों में सुनवाई को प्रभावित करता है।[22] मिश्रित, और कभी-कभी परस्पर विरोधी, अध्ययन के परिणाम हैं जो mtDNA प्रतिलिपि संख्या को कुछ कैंसर के विकास के जोखिम से जोड़ने का प्रयास करते हैं। अध्ययन आयोजित किए गए हैं जो एमटीडीएनए स्तरों में वृद्धि और कमी दोनों के बीच संबंध दिखाते हैं और स्तन कैंसर के विकास के जोखिम में वृद्धि करते हैं। बढ़े हुए एमटीडीएनए स्तरों और गुर्दे का ट्यूमर के विकास के बढ़ते जोखिम के बीच एक सकारात्मक संबंध देखा गया है लेकिन एमटीडीएनए स्तरों और पेट के कैंसर के विकास के बीच कोई संबंध प्रतीत नहीं होता है।[46] एक्स्ट्राक्रोमोसोमल डीएनए एपिकॉमप्लेक्सा में पाया जाता है, जो प्रोटोजोआ का एक समूह है। मलेरिया परजीवी (जीनस प्लाज्मोडियम), एड्स से संबंधित रोगज़नक़ (टैक्सोप्लाज्मा और Cryptosporidium ) दोनों एपिकॉम्प्लेक्स समूह के सदस्य हैं। माइटोकॉन्ड्रियल डीएनए (mtDNA) मलेरिया परजीवी में पाया गया।[47] मलेरिया परजीवियों में एक्स्ट्राक्रोमोसोमल डीएनए के दो रूप पाए जाते हैं। इनमें से एक 6-kb लीनियर DNA है और दूसरा 35-kb सर्कुलर DNA है। इन डीएनए अणुओं पर एंटीबायोटिक दवाओं के लिए संभावित न्यूक्लियोटाइड लक्ष्य स्थलों के रूप में शोध किया गया है।[48]
कैंसर में ईसीडीएनए की भूमिका
जीन प्रवर्धन ऑन्कोजीन सक्रियण के सबसे सामान्य तंत्रों में से एक है। कैंसर में जीन प्रवर्धन अक्सर एक्स्ट्राक्रोमोसोमल, सर्कुलर तत्वों पर होता है।[49][4]कैंसर में ईसीडीएनए के प्राथमिक कार्यों में से एक ट्यूमर को तेजी से उच्च प्रतिलिपि-संख्या भिन्नता तक पहुंचने में सक्षम बनाना है, जबकि तेजी से, बड़े पैमाने पर सेल-टू-सेल आनुवंशिक विषमता को बढ़ावा देना भी है।[3][8] कैंसर में सबसे अधिक प्रवर्धित ओंकोजीन ईसीडीएनए पर पाए जाते हैं और अत्यधिक गतिशील, गैर-देशी गुणसूत्रों में सजातीय धुंधला क्षेत्रों (एचएसआर) के रूप में पुन: एकीकृत होते हुए दिखाए गए हैं।[50][3]और विभिन्न दवा उपचारों के जवाब में प्रतिलिपि संख्या और संरचना को बदलना।[51][7][52] ईसीडीएनए बड़ी संख्या में अधिक उन्नत और सबसे गंभीर कैंसर के साथ-साथ कैंसर विरोधी दवाओं के प्रतिरोध के लिए जिम्मेदार है।[53] ईसीडीएनए का गोलाकार आकार क्रोमोसोमल डीएनए की रैखिक संरचना से सार्थक तरीके से भिन्न होता है जो कैंसर रोगजनन को प्रभावित करता है।[54] ईसीडीएनए पर एन्कोड किए गए ओंकोजीन में बड़े पैमाने पर ट्रांसक्रिप्शनल आउटपुट होता है, जो पूरे transcriptome में शीर्ष 1% जीनों में रैंकिंग करता है। बैक्टीरियल प्लास्मिड या माइटोकॉन्ड्रियल डीएनए के विपरीत, ईसीडीएनए को क्रोमैटिनाइज़ किया जाता है, जिसमें उच्च स्तर के सक्रिय हिस्टोन के निशान होते हैं, लेकिन दमनकारी हिस्टोन के निशान की कमी होती है। ईसीडीएनए क्रोमैटिन आर्किटेक्चर में क्रोमोसोमल डीएनए पर मौजूद उच्च-क्रम संघनन का अभाव है और पूरे कैंसर जीनोम में सबसे सुलभ डीएनए में से एक है।
EcDNAs को नाभिक के भीतर एक साथ जोड़ा जा सकता है, जिसे ईसीडीएनए हब कहा जा सकता है।[55] विशेष रूप से, ईसीडीएनए हब ऑन्कोजीन ओवरएक्प्रेशन को बढ़ावा देने के लिए इंटरमॉलिक्युलर एन्हांसर-जीन इंटरैक्शन का कारण बन सकता है।
संदर्भ
- ↑ 1.0 1.1 1.2 Rush MG, Misra R (November 1985). "यूकेरियोट्स में एक्स्ट्राक्रोमोसोमल डीएनए". Plasmid. 14 (3): 177–91. doi:10.1016/0147-619X(85)90001-0. PMID 3912782.
- ↑ 2.0 2.1 2.2 2.3 Verhaak RG, Bafna V, Mischel PS (May 2019). "ट्यूमर रोगजनन और विकास में एक्स्ट्राक्रोमोसोमल ऑन्कोजीन प्रवर्धन". Nature Reviews. Cancer. 19 (5): 283–288. doi:10.1038/s41568-019-0128-6. PMC 7168519. PMID 30872802.
- ↑ 3.0 3.1 3.2 3.3 3.4 3.5 3.6 Turner KM, Deshpande V, Beyter D, Koga T, Rusert J, Lee C, et al. (March 2017). "एक्स्ट्राक्रोमोसोमल ऑन्कोजीन प्रवर्धन ट्यूमर के विकास और आनुवंशिक विषमता को संचालित करता है". Nature. 543 (7643): 122–125. Bibcode:2017Natur.543..122T. doi:10.1038/nature21356. PMC 5334176. PMID 28178237.
- ↑ 4.0 4.1 4.2 "क्रोमोसोम के बाहर डीएनए द्वारा कैंसर को प्रेरित किया जा सकता है". The Scientist Magazine® (in English). Retrieved 2021-10-05.
- ↑ 5.0 5.1 Kuttler F, Mai S (February 2007). "विकास, विभेदीकरण और ऑन्कोजेनेसिस के दौरान गैर-यादृच्छिक एक्स्ट्राक्रोमोसोमल तत्वों का निर्माण". Seminars in Cancer Biology. 17 (1): 56–64. doi:10.1016/j.semcancer.2006.10.007. PMID 17116402.
- ↑ Alberts B, Bray D, Hopkin K, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2014). आवश्यक कोशिका जीव विज्ञान (Fourth ed.). New York, New York, USA: Garland Science. p. 449. ISBN 978-0-8153-4454-4.
- ↑ 7.0 7.1 Nathanson DA, Gini B, Mottahedeh J, Visnyei K, Koga T, Gomez G, et al. (January 2014). "एक्स्ट्राक्रोमोसोमल म्यूटेंट ईजीएफआर डीएनए के गतिशील विनियमन द्वारा मध्यस्थता से लक्षित चिकित्सा प्रतिरोध". Science. 343 (6166): 72–6. Bibcode:2014Sci...343...72N. doi:10.1126/science.1241328. PMC 4049335. PMID 24310612.
- ↑ 8.0 8.1 deCarvalho AC, Kim H, Poisson LM, Winn ME, Mueller C, Cherba D, et al. (May 2018). "क्रोमोसोमल और एक्स्ट्राक्रोमोसोमल डीएनए तत्वों की असंगत विरासत ग्लियोब्लास्टोमा में गतिशील रोग विकास में योगदान करती है". Nature Genetics. 50 (5): 708–717. doi:10.1038/s41588-018-0105-0. PMC 5934307. PMID 29686388.
- ↑ Koch J, Vogt G, Kissel W (May 1983). "साइटोप्लाज्मिक डीएनए परमाणु डीएनए से संरचनात्मक रूप से अलग है". Die Naturwissenschaften. 70 (5): 252–4. Bibcode:1983NW.....70..252K. doi:10.1007/BF00405447. PMID 6877387. S2CID 9721603.
- ↑ 10.0 10.1 Nelson D (2008). जैव रसायन के लेहिंगर सिद्धांत. New York: W. H. Freeman and Company. pp. 307–308. ISBN 978-0-7167-7108-1.
- ↑ Watson J (2007). Recombinant RNA: Genes and Genomes- A Short Course. New York: W. H. Freeman and Company. p. 81. ISBN 978-0-7167-2866-5.
- ↑ Dib JR, Liebl W, Wagenknecht M, Farías ME, Meinhardt F (January 2013). "माइक्रोकोकस में एक्स्ट्राक्रोमोसोमल जेनेटिक तत्व". Applied Microbiology and Biotechnology. 97 (1): 63–75. doi:10.1007/s00253-012-4539-5. PMID 23138713. S2CID 5623260.
- ↑ Barnum S (2005). जैव प्रौद्योगिकी- एक परिचय. California: Brooks / Cole. pp. 62–63. ISBN 978-0-495-11205-1.
- ↑ Laddy DJ, Weiner DB (2006). "From plasmids to protection: a review of DNA vaccines against infectious diseases". International Reviews of Immunology. 25 (3–4): 99–123. doi:10.1080/08830180600785827. PMID 16818367. S2CID 38432882.
- ↑ Ongkudon CM, Ho J, Danquah MK (March 2011). "Mitigating the looming vaccine crisis: production and delivery of plasmid-based vaccines" (PDF). Critical Reviews in Biotechnology. 31 (1): 32–52. doi:10.3109/07388551.2010.483460. PMID 20879832. S2CID 207467603.
- ↑ 16.0 16.1 16.2 Hinnebusch J, Tilly K (December 1993). "बैक्टीरिया में रैखिक प्लास्मिड और गुणसूत्र". Molecular Microbiology. 10 (5): 917–22. doi:10.1111/j.1365-2958.1993.tb00963.x. PMID 7934868. S2CID 23852021.
- ↑ 17.0 17.1 Meinhardt F, Schaffrath R, Larsen M (April 1997). "माइक्रोबियल रैखिक प्लास्मिड". Applied Microbiology and Biotechnology. 47 (4): 329–36. doi:10.1007/s002530050936. PMID 9163946. S2CID 8092364.
- ↑ Dance, Amber (16 July 2021). "बड़े पैमाने पर डीएनए 'बोर्ग' वैज्ञानिकों को हैरान कर देता है". Nature (in English). 595 (7869): 636. Bibcode:2021Natur.595..636D. doi:10.1038/d41586-021-01947-3. S2CID 236001172.
- ↑ Andrew, Shakespeare, William Gurr (30 July 2021). "पहले अनदेखा डीएनए 'बोर्ग' कैलिफोर्निया आर्द्रभूमि पर पाया गया". The Independent (in English). Retrieved 13 August 2021.
{{cite news}}: CS1 maint: multiple names: authors list (link) - ↑ 20.0 20.1 20.2 Lodish H (2013). Molecular Cell Biology, 7th edition. New York: W.H. Freeman and Company. pp. 245–251. ISBN 978-1-4641-2398-6.
- ↑ 21.0 21.1 21.2 Chinnery PF, Turnbull DM (July 1999). "माइटोकॉन्ड्रियल डीएनए और रोग". Lancet. 354 Suppl 1 (9176): SI17-21. doi:10.1016/S0140-6736(99)90244-1. PMID 10437851. S2CID 38869885.
- ↑ 22.0 22.1 22.2 Dimmock D, Tang LY, Schmitt ES, Wong LJ (July 2010). "माइटोकॉन्ड्रियल डीएनए रिक्तीकरण सिंड्रोम का मात्रात्मक मूल्यांकन". Clinical Chemistry. 56 (7): 1119–27. doi:10.1373/clinchem.2009.141549. PMID 20448188.
- ↑ Bohr VA, Anson RM (August 1999). "माइटोकॉन्ड्रियल डीएनए की मरम्मत के रास्ते". Journal of Bioenergetics and Biomembranes. 31 (4): 391–8. doi:10.1023/A:1005484004167. PMID 10665528. S2CID 43100873.
- ↑ Bendich AJ (June 2010). "माइटोकॉन्ड्रियल डीएनए, क्लोरोप्लास्ट डीएनए और यूकेरियोटिक जीवों में विकास की उत्पत्ति". Biology Direct. 5 (42): 42. doi:10.1186/1745-6150-5-42. PMC 2907347. PMID 20587059.
- ↑ Bernt M, Braband A, Schierwater B, Stadler PF (November 2013). "माइटोकॉन्ड्रियल जीनोम विकास के आनुवंशिक पहलू". Molecular Phylogenetics and Evolution. 69 (2): 328–38. doi:10.1016/j.ympev.2012.10.020. PMID 23142697.
- ↑ Clegg MT, Gaut BS, Learn GH, Morton BR (July 1994). "क्लोरोप्लास्ट डीएनए विकास की दरें और पैटर्न". Proceedings of the National Academy of Sciences of the United States of America. 91 (15): 6795–801. Bibcode:1994PNAS...91.6795C. doi:10.1073/pnas.91.15.6795. PMC 44285. PMID 8041699.
- ↑ Lilly JW, Havey MJ, Jackson SA, Jiang J (February 2001). "साइटोजेनोमिक विश्लेषण से उच्च पौधों में क्लोरोप्लास्ट जीनोम की संरचनात्मक प्लास्टिसिटी का पता चलता है". The Plant Cell. 13 (2): 245–54. doi:10.1105/tpc.13.2.245. PMC 102240. PMID 11226183.
- ↑ Aronsson H, Sandelius AS (2009). क्लोरोप्लास्ट पर्यावरण के साथ परस्पर क्रिया करता है ([Online-Ausg.]. ed.). Berlin: Springer. p. 18. ISBN 978-3540686965.
- ↑ Bendich AJ (July 2004). "Circular chloroplast chromosomes: the grand illusion". The Plant Cell. 16 (7): 1661–6. doi:10.1105/tpc.160771. PMC 514151. PMID 15235123.
- ↑ Wang Y, Ding J, Daniell H, Hu H, Li X (September 2012). "मोटिफ विश्लेषण क्लोरोप्लास्ट जीन के संभावित सह-नियमन और क्लोरोप्लास्ट प्रोटीन को कूटने वाले परमाणु जीन का खुलासा करता है". Plant Molecular Biology. 80 (2): 177–87. doi:10.1007/s11103-012-9938-6. PMID 22733202. S2CID 18567678.
- ↑ Pfalz J, Pfannschmidt T (April 2013). "प्रारंभिक क्लोरोप्लास्ट विकास में आवश्यक न्यूक्लियॉइड प्रोटीन". Trends in Plant Science. 18 (4): 186–94. doi:10.1016/j.tplants.2012.11.003. PMID 23246438.
- ↑ Rowan BA, Bendich AJ (2009). "The loss of DNA from chloroplasts as leaves mature: fact or artefact?". Journal of Experimental Botany. 60 (11): 3005–10. doi:10.1093/jxb/erp158. PMID 19454766.
- ↑ 33.0 33.1 Cohen S, Houben A, Segal D (March 2008). "एक्स्ट्राक्रोमोसोमल सर्कुलर डीएनए पौधों में अग्रानुक्रमिक रूप से दोहराए जाने वाले जीनोमिक अनुक्रमों से प्राप्त होता है". The Plant Journal. 53 (6): 1027–34. doi:10.1111/j.1365-313X.2007.03394.x. PMID 18088310.
- ↑ Cohen Z, Lavi S (July 2009). Sullivan BA (ed.). "स्तनधारी कोशिका-मुक्त प्रणाली में एक्स्ट्राक्रोमोसोमल सर्कुलर डीएनए का प्रतिकृति स्वतंत्र गठन". PLOS ONE. 4 (7): e6126. Bibcode:2009PLoSO...4.6126C. doi:10.1371/journal.pone.0006126. PMC 2699479. PMID 19568438.
- ↑ Cohen S, Agmon N, Sobol O, Segal D (March 2010). "Extrachromosomal circles of satellite repeats and 5S ribosomal DNA in human cells". Mobile DNA. 1 (1): 11. doi:10.1186/1759-8753-1-11. PMC 3225859. PMID 20226008.
- ↑ cen.acs.org https://cen.acs.org/pharmaceuticals/oncology/curious-DNA-circles-make-treating/98/i40. Retrieved 2021-10-02.
{{cite web}}: Missing or empty|title=(help) - ↑ Sanjuán R, Nebot MR, Chirico N, Mansky LM, Belshaw R (October 2010). "वायरल उत्परिवर्तन दर". Journal of Virology. 84 (19): 9733–48. doi:10.1128/JVI.00694-10. PMC 2937809. PMID 20660197.
- ↑ Silverthorn DU (2007). मानव मनोविज्ञान. Peason/Benjamin Cummings.
- ↑ "वायरल जीनोम".
- ↑ Lorenz LD, Rivera Cardona J, Lambert PF (October 2013). Roman A (ed.). "Inactivation of p53 rescues the maintenance of high risk HPV DNA genomes deficient in expression of E6". PLOS Pathogens. 9 (10): e1003717. doi:10.1371/journal.ppat.1003717. PMC 3812038. PMID 24204267.
- ↑ {{cite journal | vauthors = Barber GN | title = साइटोप्लाज्मिक डीएनए जन्मजात प्रतिरक्षा मार्ग| journal = Immunological Reviews | volume = 243 | issue = 1 | pages = 99–108 | date = September 2011 | pmid = 21884170 | doi = 10.1111/j.1600-065X.2011.01051.x | s2cid = 205826027 }
- ↑ 43.0 43.1 Barber GN (February 2011). "सहज प्रतिरक्षा डीएनए संवेदन मार्ग: STING, AIMII और इंटरफेरॉन उत्पादन और भड़काऊ प्रतिक्रियाओं का विनियमन". Current Opinion in Immunology. 23 (1): 10–20. doi:10.1016/j.coi.2010.12.015. PMC 3881186. PMID 21239155.
- ↑ Griffiths AJ (2000). आनुवंशिक विश्लेषण का एक परिचय. New York: W.H.Freeman.
- ↑ 45.0 45.1 Sato M, Sato K (August 2013). "पैतृक माइटोकॉन्ड्रियल डीएनए को खत्म करने के लिए विभिन्न तंत्रों द्वारा माइटोकॉन्ड्रियल डीएनए की मातृ विरासत". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1833 (8): 1979–84. doi:10.1016/j.bbamcr.2013.03.010. PMID 23524114.
- ↑ Thyagarajan B, Wang R, Nelson H, Barcelo H, Koh WP, Yuan JM (June 2013). Bai Y (ed.). "माइटोकॉन्ड्रियल डीएनए कॉपी नंबर स्तन कैंसर के खतरे से जुड़ा है". PLOS ONE. 8 (6): e65968. Bibcode:2013PLoSO...865968T. doi:10.1371/journal.pone.0065968. PMC 3680391. PMID 23776581.
- ↑ Wilson RJ, Williamson DH (March 1997). "Apicomplexa में एक्स्ट्राक्रोमोसोमल डीएनए". Microbiology and Molecular Biology Reviews. 61 (1): 1–16. doi:10.1128/mmbr.61.1.1-16.1997. PMC 232597. PMID 9106361.
- ↑ Creasey A, Mendis K, Carlton J, Williamson D, Wilson I, Carter R (May 1994). "मलेरिया परजीवियों में एक्स्ट्राक्रोमोसोमल डीएनए की मातृ विरासत". Molecular and Biochemical Parasitology. 65 (1): 95–8. doi:10.1016/0166-6851(94)90118-X. PMID 7935632.
- ↑ Kim H, Nguyen NP, Turner K, Wu S, Gujar AD, Luebeck J, et al. (August 2020). "एक्स्ट्राक्रोमोसोमल डीएनए ऑन्कोजीन प्रवर्धन और कई कैंसर में खराब परिणाम से जुड़ा है". Nature Genetics. 52 (8): 891–897. doi:10.1038/s41588-020-0678-2. PMC 7484012. PMID 32807987.
- ↑ Storlazzi CT, Lonoce A, Guastadisegni MC, Trombetta D, D'Addabbo P, Daniele G, et al. (September 2010). "Gene amplification as double minutes or homogeneously staining regions in solid tumors: origin and structure". Genome Research. 20 (9): 1198–206. doi:10.1101/gr.106252.110. PMC 2928498. PMID 20631050.
- ↑ Von Hoff DD, McGill JR, Forseth BJ, Davidson KK, Bradley TP, Van Devanter DR, Wahl GM (September 1992). "मानव ट्यूमर कोशिकाओं से एक्स्ट्राक्रोमोसोमली प्रवर्धित MYC जीन का उन्मूलन उनकी ट्यूमरजन्यता को कम करता है". Proceedings of the National Academy of Sciences of the United States of America. 89 (17): 8165–9. Bibcode:1992PNAS...89.8165V. doi:10.1073/pnas.89.17.8165. PMC 49877. PMID 1518843.
- ↑ Xu K, Ding L, Chang TC, Shao Y, Chiang J, Mulder H, et al. (January 2019). "ब्रेन ट्यूमर के निदान और पुनरावर्तन में दोहरे मिनट की संरचना और विकास". Acta Neuropathologica. 137 (1): 123–137. doi:10.1007/s00401-018-1912-1. PMC 6338707. PMID 30267146.
- ↑ "'Bond villain' DNA could transform cancer treatment, scientists say". February 18, 2023. doi:10.1038/s41588-022-01177-x.
- ↑ Wu S, Turner KM, Nguyen N, Raviram R, Erb M, Santini J, et al. (November 2019). "परिपत्र ईसीडीएनए सुलभ क्रोमैटिन और उच्च ऑन्कोजीन अभिव्यक्ति को बढ़ावा देता है". Nature. 575 (7784): 699–703. Bibcode:2019Natur.575..699W. doi:10.1038/s41586-019-1763-5. PMC 7094777. PMID 31748743.
- ↑ Hung, King L.; Yost, Kathryn E.; Xie, Liangqi; Shi, Quanming; Helmsauer, Konstantin; Luebeck, Jens; Schöpflin, Robert; Lange, Joshua T.; Chamorro González, Rocío; Weiser, Natasha E.; Chen, Celine (December 2021). "ecDNA हब सहकारी इंटरमॉलिक्युलर ऑन्कोजीन अभिव्यक्ति को संचालित करता है". Nature (in English). 600 (7890): 731–736. Bibcode:2021Natur.600..731H. doi:10.1038/s41586-021-04116-8. ISSN 1476-4687. PMC 9126690. PMID 34819668.
अग्रिम पठन
- Bogenhagen DF (2012). "Mitochondrial DNA nucleoid structure". Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1819 (9–10): 914–20. doi:10.1016/j.bbagrm.2011.11.005. PMID 22142616.
- Cara A, Reitz MS (September 1997). "New insight on the role of extrachromosomal retroviral DNA". Leukemia. 11 (9): 1395–9. doi:10.1038/sj.leu.2400776. PMID 9305590.
- Cohen S, Yacobi K, Segal D (June 2003). "Extrachromosomal circular DNA of tandemly repeated genomic sequences in Drosophila". Genome Research. 13 (6A): 1133–45. doi:10.1101/gr.907603. PMC 403641. PMID 12799349.
- Cohen S, Méchali M (December 2002). "Formation of extrachromosomal circles from telomeric DNA in Xenopus laevis". EMBO Reports. 3 (12): 1168–74. doi:10.1093/embo-reports/kvf240. PMC 1308322. PMID 12446568.
- Colosimo A, Guida V, Palka G, Dallapiccola B (June 2002). "Extrachromosomal genes: a powerful tool in gene targeting approaches". Gene Therapy. 9 (11): 679–82. doi:10.1038/sj.gt.3301749. PMID 12032686.
- Cummings D (1979). Extrachromosomal DNA. New York: Academic Press Inc.
- Goebel W (August 1970). "Studies on extrachromosomal DNA elements. Replication of the colicinogenic factor Col E1 in two temperature sensitive mutants of Escherichia coli defective in DNA replication". European Journal of Biochemistry. 15 (2): 311–20. doi:10.1111/j.1432-1033.1970.tb01009.x. PMID 4926129.
- Jabaji-Hare SH, Burger G, Forget L, Lang BF (May 1994). "Extrachromosomal plasmids in the plant pathogenic fungus Rhizoctonia solani". Current Genetics. 25 (5): 423–31. doi:10.1007/BF00351781. PMID 8082188. S2CID 20902405.
- Preer JR (1971). "Extrachromosomal inheritance: hereditary symbionts, mitochondria, chiloroplasts". Annual Review of Genetics. 5: 361–406. doi:10.1146/annurev.ge.05.120171.002045. PMID 16097660.
- Shibata Y, Kumar P, Layer R, Willcox S, Gagan JR, Griffith JD, Dutta A (April 2012). "Extrachromosomal microDNAs and chromosomal microdeletions in normal tissues". Science. 336 (6077): 82–6. Bibcode:2012Sci...336...82S. doi:10.1126/science.1213307. PMC 3703515. PMID 22403181.
- Sloan DB (December 2013). "One ring to rule them all? Genome sequencing provides new insights into the 'master circle' model of plant mitochondrial DNA structure". The New Phytologist. 200 (4): 978–85. doi:10.1111/nph.12395. PMID 24712049. S2CID 13445964.
- Watve MM, Dahanukar N, Watve MG (February 2010). Getz WM (ed.). "Sociobiological control of plasmid copy number in bacteria". PLOS ONE. 5 (2): e9328. Bibcode:2010PLoSO...5.9328W. doi:10.1371/journal.pone.0009328. PMC 2827543. PMID 20195362.