अभिकलनात्मक जीवविज्ञान (कम्प्यूटेशनल बायोलॉजी)
कम्प्यूटेशनल जीव विज्ञान जैविक प्रणालियों और संबंधों को समझने के लिए डेटा विश्लेषण, गणितीय मॉडलिंग और कंप्यूटर सिमुलेशन के उपयोग को संदर्भित करता है।[1]कंप्यूटर विज्ञान, जीव विज्ञान, और बड़े डेटा का एक चौराहा, इस क्षेत्र में अनुप्रयुक्त गणित, रसायन विज्ञान और आनुवंशिकी में भी नींव है।[2]यह जैविक कंप्यूटिंग से अलग है, कंप्यूटर इंजीनियरिंग का एक उपक्षेत्र जो कंप्यूटर बनाने के लिए जैव अभियांत्रिकी का उपयोग करता है।
इतिहास
जैव सूचना विज्ञान, जैविक प्रणालियों में सूचना विज्ञान प्रक्रियाओं का विश्लेषण, 1970 के दशक की शुरुआत में शुरू हुआ। इस समय, कृत्रिम होशियारी में अनुसंधान नए एल्गोरिदम उत्पन्न करने के लिए मानव मस्तिष्क के नेटवर्क मॉडल का उपयोग कर रहा था। जैविक डेटा के इस उपयोग ने जैविक शोधकर्ताओं को अपने क्षेत्र में बड़े डेटा सेटों का मूल्यांकन और तुलना करने के लिए कंप्यूटर का उपयोग करने के लिए प्रेरित किया।[3] 1982 तक, शोधकर्ताओं ने छिद्रित कार्ड के माध्यम से जानकारी साझा की। 1980 के दशक के अंत तक डेटा की मात्रा में तेजी से वृद्धि हुई, प्रासंगिक जानकारी की त्वरित व्याख्या के लिए नए कम्प्यूटेशनल तरीकों की आवश्यकता थी।[3]
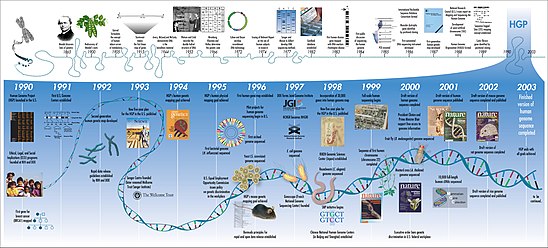
कम्प्यूटेशनल जीव विज्ञान का शायद सबसे प्रसिद्ध उदाहरण, ह्यूमन जीनोम प्रोजेक्ट, आधिकारिक तौर पर 1990 में शुरू हुआ।[4] 2003 तक, परियोजना ने अपने शुरुआती लक्ष्यों को पूरा करते हुए मानव जीनोम का लगभग 85% मानचित्रण किया था।[5] हालाँकि, काम जारी रहा, और 2021 के स्तर तक पूर्ण जीनोम तक पहुँच गया, जिसमें केवल 0.3% शेष आधार संभावित मुद्दों से आच्छादित थे।[6][7] लापता Y गुणसूत्र जनवरी 2022 में जोड़ा गया था।
1990 के दशक के उत्तरार्ध से, कम्प्यूटेशनल जीव विज्ञान, जीव विज्ञान का एक महत्वपूर्ण हिस्सा बन गया है, जिससे कई उपक्षेत्र बन गए हैं।[8] आज, कम्प्यूटेशनल जीवविज्ञान के लिए अंतर्राष्ट्रीय सोसायटी 21 अलग-अलग 'विशेष रुचि के समुदायों' को मान्यता देती है, प्रत्येक बड़े क्षेत्र के एक टुकड़े का प्रतिनिधित्व करता है।[9] मानव जीनोम के अनुक्रम में मदद करने के अलावा, कम्प्यूटेशनल जीव विज्ञान ने मानव मस्तिष्क, जीनोम आर्किटेक्चर मैपिंग और नमूना जैविक प्रणालियों के सटीक मॉडल बनाने में मदद की है।[3]
अनुप्रयोग
एनाटॉमी
कम्प्यूटेशनल एनाटॉमी दृश्य या सकल शरीर रचना पर शारीरिक आकार और रूप का अध्ययन है आकृति विज्ञान (जीव विज्ञान) का पैमाना। इसमें जैविक संरचनाओं के मॉडलिंग और अनुकरण के लिए कम्प्यूटेशनल गणितीय और डेटा-विश्लेषणात्मक तरीकों का विकास शामिल है। यह मेडिकल इमेजिंग उपकरणों के बजाय इमेज की जा रही संरचनात्मक संरचनाओं पर केंद्रित है। चुंबकीय अनुनाद इमेजिंग जैसी प्रौद्योगिकियों के माध्यम से घने 3डी माप की उपलब्धता के कारण, कम्प्यूटेशनल एनाटॉमी 3डी में मोर्फेम स्केल पर एनाटोमिकल कोऑर्डिनेट सिस्टम निकालने के लिए मेडिकल इमेजिंग और बायोइंजीनियरिंग के एक उपक्षेत्र के रूप में उभरा है।
कम्प्यूटेशनल एनाटॉमी का मूल सूत्रीकरण रूपांतरणों के माध्यम से कार्य करने वाले उदाहरणों से आकार और रूप के एक जनरेटिव मॉडल के रूप में है।[10] अलग-अलग समन्वय प्रणालियों का अध्ययन करने के लिए अलग-अलग समन्वय प्रणालियों का उपयोग किया जाता है, जैसा कि एक संरचनात्मक विन्यास से प्रवाह क्षेत्र के लैग्रेंजियन और यूलेरियन विनिर्देश के माध्यम से उत्पन्न होता है। दूसरे करने के लिए। यह आकृति के आँकड़ों और morphometrics से संबंधित है, इस अंतर के साथ कि अलग-अलग समन्वय प्रणालियों को मानचित्रित करने के लिए उपयोग किया जाता है, जिनके अध्ययन को डिफियोमोर्फोमेट्री के रूप में जाना जाता है।
डेटा और मॉडलिंग
गणितीय जीव विज्ञान जैविक प्रणालियों में संरचना, विकास और व्यवहार को नियंत्रित करने वाली प्रणालियों की जांच करने के लिए जीवित जीवों के गणितीय मॉडल का उपयोग है। यह प्रायोगिक जीव विज्ञान के अपने अधिक अनुभवजन्य-दिमाग वाले समकक्ष के बजाय समस्याओं के लिए अधिक सैद्धांतिक दृष्टिकोण पर जोर देता है।[11] गणितीय जीव विज्ञान असतत गणित, टोपोलॉजी (कम्प्यूटेशनल मॉडलिंग के लिए भी उपयोगी), बायेसियन सांख्यिकी, रैखिक बीजगणित और बूलियन बीजगणित पर आधारित है।[12]
इन गणितीय दृष्टिकोणों ने जैविक डेटा के भंडारण, पुनर्प्राप्ति और विश्लेषण के लिए डेटाबेस और अन्य विधियों के निर्माण को सक्षम किया है, जिसे जैव सूचना विज्ञान के रूप में जाना जाता है। आमतौर पर, इस प्रक्रिया में आनुवंशिकी और जीन का विश्लेषण शामिल होता है।
बड़े डेटासेट को इकट्ठा करने और उसका विश्लेषण करने से डेटा खनन जैसे बढ़ते शोध क्षेत्रों के लिए जगह बन गई है।[12] और कम्प्यूटेशनल बायोमॉडलिंग, जो कंप्यूटर मॉडल के निर्माण और जैविक प्रणालियों की संवर्धित वास्तविकता को संदर्भित करता है। यह शोधकर्ताओं को यह अनुमान लगाने की अनुमति देता है कि ऐसी प्रणालियाँ विभिन्न वातावरणों पर कैसे प्रतिक्रिया देंगी, जो यह निर्धारित करने के लिए उपयोगी है कि क्या कोई प्रणाली बाहरी और आंतरिक गड़बड़ी के विरुद्ध अपनी स्थिति और कार्यों को बनाए रख सकती है।Cite error: Invalid <ref> tag; invalid names, e.g. too many जनसंख्या वृद्धि या प्राकृतिक चयन इतिहास के बारे में निष्कर्ष निकालने के लिए डीएनए डेटा के लिए या कोलेसेंट सिद्धांत)।
- पहले सिद्धांतों से विकासवादी प्रणालियों के जनसंख्या आनुवंशिकी मॉडल का निर्माण करना ताकि भविष्यवाणी की जा सके कि क्या विकसित होने की संभावना है
जीनोमिक्स
कम्प्यूटेशनल जीनोमिक्स सेल (जीव विज्ञान) और जीवों के जीनोम का अध्ययन है। मानव जीनोम परियोजना कम्प्यूटेशनल जीनोमिक्स का एक उदाहरण है। यह प्रोजेक्ट पूरे मानव जीनोम को डेटा के एक सेट में अनुक्रमित करने के लिए दिखता है। एक बार पूरी तरह लागू होने के बाद, यह डॉक्टरों को एक व्यक्तिगत रोगी के जीनोम का विश्लेषण करने की अनुमति दे सकता है।[13] यह व्यक्तिगत चिकित्सा की संभावना को खोलता है, किसी व्यक्ति के पहले से मौजूद आनुवंशिक पैटर्न के आधार पर उपचार निर्धारित करता है। शोधकर्ता जानवरों, पौधों, जीवाणुओं और अन्य सभी प्रकार के जीवन के जीनोम को अनुक्रमित करने की कोशिश कर रहे हैं। <रेफरी नाम = कूनिन 2001 155-158>Koonin, Eugene (6 March 2001). "कम्प्यूटेशनल जीनोमिक्स". Curr. Biol. 11 (5): 155–158. doi:10.1016/S0960-9822(01)00081-1. PMID 11267880. S2CID 17202180.</रेफरी>
जिन मुख्य तरीकों में जीनोम की तुलना की जाती है उनमें से एक अनुक्रम समरूपता है। होमोलॉजी विभिन्न जीवों में जैविक संरचनाओं और न्यूक्लियोटाइड अनुक्रमों का अध्ययन है जो एक सामान्य पूर्वज से आते हैं। शोध से पता चलता है कि नव अनुक्रमित प्रोकैरियोट जीनोम में 80 से 90% जीनों की पहचान इस तरह से की जा सकती है।Cite error: Invalid <ref> tag; invalid names, e.g. too many
अनुक्रम संरेखण जैविक अनुक्रमों या जीनों के बीच समानता की तुलना और पता लगाने के लिए एक और प्रक्रिया है। अनुक्रम संरेखण कई जैव सूचना विज्ञान अनुप्रयोगों में उपयोगी है, जैसे कि दो जीनों की सबसे लंबी सामान्य अनुगामी समस्या की गणना करना या कुछ बीमारियों के वेरिएंट की तुलना करना। रेफरी>"अनुक्रम संरेखण - एक सिंहावलोकन | ScienceDirect विषय". www.sciencedirect.com. Retrieved 2022-04-18.</रेफरी>
कम्प्यूटेशनल जीनोमिक्स में एक अनछुआ प्रोजेक्ट इंटरजेनिक क्षेत्रों का विश्लेषण है, जिसमें मानव जीनोम का लगभग 97% हिस्सा शामिल है। <रेफरी नाम = कूनिन 2001 155-158 /> शोधकर्ता मानव जीनोम के गैर-कोडिंग क्षेत्रों के कार्यों को समझने के लिए काम कर रहे हैं। कम्प्यूटेशनल और सांख्यिकीय विधियों के विकास के माध्यम से और ENCODE और एपिजेनोम#रोडमैप एपिजेनोमिक्स प्रोजेक्ट जैसे बड़े संघ परियोजनाओं के माध्यम से।
अणु, कोशिका (जीव विज्ञान), और जीव स्तर पर किसी जीव के जीव विज्ञान में व्यक्तिगत जीन कैसे योगदान करते हैं, यह समझना जीन ओन्टोलॉजी के रूप में जाना जाता है। जीन ओन्टोलॉजी कंसोर्टियम का मिशन आणविक स्तर से लेकर बड़े रास्ते, सेलुलर और जीव-स्तरीय प्रणालियों तक जैविक प्रणालियों का एक अप-टू-डेट, व्यापक, कम्प्यूटेशनल मॉडल विकसित कशाही सेना है। जीन ओन्टोलॉजी संसाधन मनुष्यों से लेकर जीवाणुओं तक, कई अलग-अलग जीवों से जीन के कार्यों (या, अधिक ठीक से, प्रोटीन और गैर-कोडिंग आरएनए अणु जीन द्वारा उत्पादित) के बारे में वर्तमान वैज्ञानिक ज्ञान का एक कम्प्यूटेशनल प्रतिनिधित्व प्रदान करता है। रेफरी>"जीन सत्तामीमांसा संसाधन". Gene Ontology Resource. Retrieved 2022-04-18.</रेफरी>
3डी जीनोमिक्स कम्प्यूटेशनल जीव विज्ञान में एक उपखंड है जो एक यूकेरियोटिक सेल के भीतर संगठन और जीन की बातचीत पर केंद्रित है। 3डी जीनोमिक डेटा एकत्र करने के लिए इस्तेमाल की जाने वाली एक विधि जीनोम आर्किटेक्चर मैपिंग (जीएएम) के माध्यम से होती है। जीएएम क्रायोसेक्शनिंग, डीएनए की जांच करने के लिए नाभिक से एक पट्टी काटने की प्रक्रिया, लेजर माइक्रोडिसेक्शन के साथ संयोजन करके जीनोम में क्रोमेटिन और डीएनए की 3डी दूरी को मापता है। एक परमाणु प्रोफ़ाइल केवल यह पट्टी या टुकड़ा है जो नाभिक से ली गई है। प्रत्येक परमाणु प्रोफ़ाइल में जीनोमिक खिड़कियां होती हैं, जो न्यूक्लियोटाइड्स के कुछ अनुक्रम हैं - डीएनए की आधार इकाई। जीएएम पूरे सेल में जटिल, बहु बढ़ाने वाले क्रोमैटिन संपर्कों के जीनोम नेटवर्क को कैप्चर करता है। रेफरी>Beagrie, Robert A.; Scialdone, Antonio; Schueler, Markus; Kraemer, Dorothee C. A.; Chotalia, Mita; Xie, Sheila Q.; Barbieri, Mariano; de Santiago, Inês; Lavitas, Liron-Mark; Branco, Miguel R.; Fraser, James (March 2017). "जीनोम आर्किटेक्चर मैपिंग द्वारा कैप्चर किए गए जटिल मल्टी-एन्हांसर संपर्क". Nature (in English). 543 (7646): 519–524. Bibcode:2017Natur.543..519B. doi:10.1038/nature21411. ISSN 1476-4687. PMC 5366070. PMID 28273065.</रेफरी>
तंत्रिका विज्ञान
कम्प्यूटेशनल तंत्रिका विज्ञान तंत्रिका तंत्र के सूचना प्रसंस्करण गुणों के संदर्भ में मस्तिष्क के कार्य का अध्ययन है। न्यूरोसाइंस का एक सबसेट, यह न्यूरोलॉजिकल सिस्टम के विशिष्ट पहलुओं की जांच करने के लिए मस्तिष्क को मॉडल करता है।[14] मस्तिष्क के मॉडल में शामिल हैं:
- यथार्थवादी मस्तिष्क मॉडल: ये मॉडल मस्तिष्क के हर पहलू का प्रतिनिधित्व करते हैं, जिसमें सेलुलर स्तर पर जितना संभव हो उतना विवरण शामिल है। यथार्थवादी मॉडल मस्तिष्क के बारे में सबसे अधिक जानकारी प्रदान करते हैं, लेकिन त्रुटि के लिए सबसे बड़ा मार्जिन भी होता है। एक मस्तिष्क मॉडल में अधिक चर अधिक त्रुटि होने की संभावना पैदा करते हैं। ये मॉडल सेलुलर संरचना के उन हिस्सों के बारे में नहीं बताते हैं जिनके बारे में वैज्ञानिक नहीं जानते हैं। यथार्थवादी मस्तिष्क मॉडल सबसे कम्प्यूटेशनल रूप से भारी और लागू करने के लिए सबसे महंगे हैं।[15] * सरलीकृत मस्तिष्क मॉडल: ये मॉडल न्यूरोलॉजिकल सिस्टम की एक विशिष्ट भौतिक संपत्ति का आकलन करने के लिए एक मॉडल के दायरे को सीमित करने के लिए देखते हैं। यह गहन कम्प्यूटेशनल समस्याओं को हल करने की अनुमति देता है, और यथार्थवादी मस्तिष्क मॉडल से संभावित त्रुटि की मात्रा को कम करता है।[15]वर्तमान में ऐसी गणनाओं की गति बढ़ाने के लिए उपयोग किए जाने वाले एल्गोरिदम और डेटा संरचनाओं में सुधार करना कम्प्यूटेशनल न्यूरोसाइंटिस्ट का काम है।
कम्प्यूटेशनल neuropsychiatry एक उभरता हुआ क्षेत्र है जो मानसिक विकारों में शामिल मस्तिष्क तंत्र के गणितीय और कंप्यूटर-समर्थित मॉडलिंग का उपयोग करता है। कई पहलों ने प्रदर्शित किया है कि कम्प्यूटेशनल मॉडलिंग न्यूरोनल सर्किट को समझने में एक महत्वपूर्ण योगदान है जो मानसिक कार्यों और शिथिलता उत्पन्न कर सकता है।[16][17][18]
फार्माकोलॉजी
कम्प्यूटेशनल फार्माकोलॉजी विशिष्ट जीनोटाइप और बीमारियों और फिर दवा की खोज के बीच संबंध खोजने के लिए जीनोमिक डेटा के प्रभावों का अध्ययन है।[19] दवा उद्योग को दवा डेटा का विश्लेषण करने के तरीकों में बदलाव की आवश्यकता है। फार्माकोलॉजिस्ट दवाओं की प्रभावशीलता से संबंधित रासायनिक और जीनोमिक डेटा की तुलना करने के लिए Microsoft Excel का उपयोग करने में सक्षम थे। हालाँकि, उद्योग एक्सेल बैरिकेड्स के रूप में जाना जाता है। यह स्प्रेडशीट पर पहुंच योग्य सीमित संख्या में सेल से उत्पन्न होता है। इस विकास ने कम्प्यूटेशनल फार्माकोलॉजी की आवश्यकता को जन्म दिया। वैज्ञानिक और शोधकर्ता इन विशाल डेटा सेटों का विश्लेषण करने के लिए कम्प्यूटेशनल तरीके विकसित करते हैं। यह उल्लेखनीय डेटा बिंदुओं के बीच एक कुशल तुलना की अनुमति देता है और अधिक सटीक दवाओं को विकसित करने की अनुमति देता है।[20] विश्लेषकों का अनुमान है कि यदि पेटेंट के कारण प्रमुख दवाएं विफल हो जाती हैं, तो बाजार पर मौजूदा दवाओं को बदलने के लिए कम्प्यूटेशनल जीव विज्ञान आवश्यक होगा। कम्प्यूटेशनल जीव विज्ञान में डॉक्टरेट छात्रों को डॉक्टरेट के बाद के पदों को लेने के बजाय उद्योग में करियर बनाने के लिए प्रोत्साहित किया जा रहा है। यह नई दवाओं के उत्पादन के लिए आवश्यक बड़े डेटा सेट के अधिक योग्य विश्लेषकों की आवश्यकता वाली प्रमुख दवा कंपनियों का प्रत्यक्ष परिणाम है।[20]
इसी तरह, कम्प्यूटेशनल ऑन्कोलॉजी का उद्देश्य एल्गोरिथम दृष्टिकोण के माध्यम से कैंसर में भविष्य के उत्परिवर्तन को निर्धारित करना है। इस क्षेत्र में अनुसंधान ने उच्च-थ्रूपुट मापन का उपयोग करने के लिए प्रेरित किया है जो रोबोटिक्स और अन्य संवेदन उपकरणों का उपयोग करते हुए लाखों डेटा बिंदु हैं। यह डेटा डीएनए, आरएनए और अन्य जैविक संरचनाओं से एकत्र किया जाता है। फोकस के क्षेत्रों में सूजन की विशेषताओं का निर्धारण करना, अणुओं का विश्लेषण करना जो कैंसर पैदा करने में नियतात्मक हैं, और यह समझना कि मानव जीनोम ट्यूमर और कैंसर के कारण से कैसे संबंधित है।[21][22]
तकनीक
कम्प्यूटेशनल जीवविज्ञानी अपने शोध करने के लिए सॉफ्टवेयर और एल्गोरिदम की एक विस्तृत श्रृंखला का उपयोग करते हैं।
अनपर्यवेक्षित लर्निंग
अनियंत्रित शिक्षा एक प्रकार का एल्गोरिदम है जो बिना लेबल वाले डेटा में पैटर्न ढूंढता है। एक उदाहरण k- साधन क्लस्टरिंग है, जिसका उद्देश्य n डेटा बिंदुओं को k समूहों में विभाजित करना है, जिसमें प्रत्येक डेटा बिंदु निकटतम माध्य वाले क्लस्टर से संबंधित है। एक अन्य संस्करण k-मेडोइड्स एल्गोरिथम है, जो क्लस्टर केंद्र या क्लस्टर सेंट्रोइड का चयन करते समय, सेट में इसके डेटा बिंदुओं में से एक को चुनेगा, न कि केवल क्लस्टर का औसत।
एल्गोरिथ्म इन चरणों का पालन करता है:
- यादृच्छिक रूप से k विशिष्ट डेटा बिंदुओं का चयन करें। ये शुरुआती क्लस्टर हैं।
- प्रत्येक बिंदु और प्रत्येक 'के' क्लस्टर के बीच की दूरी को मापें। (यह प्रत्येक बिंदु k से बिंदुओं की दूरी है)।
- प्रत्येक बिंदु को निकटतम क्लस्टर में असाइन करें।
- प्रत्येक क्लस्टर (मेडॉइड) का केंद्र खोजें।
- तब तक दोहराएं जब तक कि क्लस्टर अब नहीं बदलते।
- प्रत्येक क्लस्टर के भीतर भिन्नता जोड़कर क्लस्टरिंग की गुणवत्ता का आकलन करें।
- k के विभिन्न मानों के साथ प्रक्रियाओं को दोहराएं।
- 'k' के लिए सबसे अच्छा मान चुनें, उस प्लॉट में कोहनी ढूंढकर जिसमें k मान सबसे कम विचरण करता है।
जीव विज्ञान में इसका एक उदाहरण जीनोम के 3डी मानचित्रण में प्रयोग किया जाता है। क्रोमोसोम 13 के माउस के HIST1 क्षेत्र की जानकारी सभी के लिए जीन अभिव्यक्ति से एकत्रित की जाती है।[23] इस जानकारी में डेटा शामिल है जिस पर परमाणु प्रोफाइल कुछ जीनोमिक क्षेत्रों में दिखाई देते हैं। इस जानकारी के साथ, सभी लोकी के बीच सामान्यीकृत दूरी खोजने के लिए जैकार्ड दूरी का उपयोग किया जा सकता है।
ग्राफ एनालिटिक्स
ग्राफ़ एनालिटिक्स, या नेटवर्क सिद्धांत, ग्राफ़ का अध्ययन है जो विभिन्न वस्तुओं के बीच कनेक्शन का प्रतिनिधित्व करता है। जीव विज्ञान में ग्राफ़ सभी प्रकार के नेटवर्क का प्रतिनिधित्व कर सकते हैं जैसे प्रोटीन-प्रोटीन इंटरैक्शन | प्रोटीन-प्रोटीन इंटरैक्शन नेटवर्क, नियामक नेटवर्क, मेटाबोलिक और जैव रासायनिक नेटवर्क और बहुत कुछ। इन नेटवर्कों का विश्लेषण करने के कई तरीके हैं। जिनमें से एक रेखांकन में केंद्रीयता को देख रहा है। ग्राफ़ में केंद्रीयता ढूँढना नोड रैंकिंग को उनकी लोकप्रियता या ग्राफ़ में केंद्रीयता प्रदान करता है। यह पता लगाने में उपयोगी हो सकता है कि कौन से नोड सबसे महत्वपूर्ण हैं। जीव विज्ञान में यह कई तरह से बहुत उपयोगी हो सकता है। उदाहरण के लिए, यदि हमारे पास एक निश्चित समय अवधि में जीन की गतिविधि पर डेटा होना था, तो हम यह देखने के लिए डिग्री केंद्रीयता का उपयोग कर सकते हैं कि कौन से जीन पूरे नेटवर्क में सबसे अधिक सक्रिय हैं, या कौन से जीन पूरे नेटवर्क में दूसरों के साथ सबसे अधिक बातचीत करते हैं। इससे हमें यह समझने में मदद मिल सकती है कि कुछ जीन नेटवर्क में क्या भूमिका निभाते हैं।
ग्राफ़ में केंद्रीयता की गणना करने के कई तरीके हैं, जिनमें से सभी केंद्रीयता पर विभिन्न प्रकार की जानकारी दे सकते हैं। जीव विज्ञान में केंद्रीयताओं को खोजना कई अलग-अलग परिस्थितियों में लागू किया जा सकता है, जिनमें से कुछ जीन विनियामक, प्रोटीन इंटरैक्शन और मेटाबोलिक नेटवर्क हैं।[24]
पर्यवेक्षित शिक्षण
पर्यवेक्षित अध्ययन is a type of algorithm that learns from labeled data and learns how to assign labels to future data that is unlabeled. In biology supervised learning can be helpful when we have data that we know how to categorize and we would like to categorize more data into those categories.
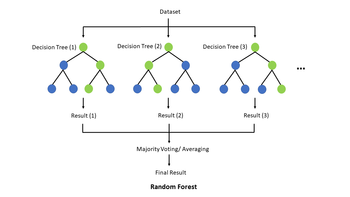
एक सामान्य पर्यवेक्षित शिक्षण एल्गोरिथ्म यादृच्छिक वन है, जो एक डेटासेट को वर्गीकृत करने के लिए एक मॉडल को प्रशिक्षित करने के लिए कई निर्णय ट्री सीखने का उपयोग करता है। यादृच्छिक जंगल के आधार पर, एक निर्णय पेड़ एक संरचना है जिसका उद्देश्य उस डेटा की कुछ ज्ञात विशेषताओं का उपयोग करके डेटा के कुछ सेट को वर्गीकृत करना या लेबल करना है। इसका एक व्यावहारिक जैविक उदाहरण एक व्यक्ति के आनुवंशिक डेटा को लेना और यह भविष्यवाणी करना होगा कि क्या वह व्यक्ति एक निश्चित बीमारी या कैंसर विकसित करने के लिए पूर्वनिर्धारित है या नहीं। प्रत्येक आंतरिक नोड पर एल्गोरिद्म ठीक एक विशेषता के लिए डेटासेट की जाँच करता है, पिछले उदाहरण में एक विशिष्ट जीन, और फिर परिणाम के आधार पर बाएँ या दाएँ शाखाएँ। फिर प्रत्येक लीफ नोड पर, डिसीजन ट्री डेटासेट को एक क्लास लेबल प्रदान करता है। इसलिए व्यवहार में, एल्गोरिथ्म निर्णय ट्री के माध्यम से इनपुट डेटासेट के आधार पर एक विशिष्ट रूट-टू-लीफ पथ पर चलता है, जिसके परिणामस्वरूप उस डेटासेट का वर्गीकरण होता है। आमतौर पर, डिसीजन ट्री में टार्गेट वेरिएबल्स होते हैं जो असतत मान लेते हैं, जैसे हां/नहीं, जिस स्थिति में इसे वर्गीकरण चार्ट के रूप में संदर्भित किया जाता है, लेकिन यदि टारगेट वेरिएबल निरंतर है तो इसे प्रतिगमन वृक्ष कहा जाता है। एक निर्णय वृक्ष का निर्माण करने के लिए, इसे पहले एक प्रशिक्षण सेट का उपयोग करके प्रशिक्षित किया जाना चाहिए ताकि यह पता चल सके कि लक्ष्य चर के सबसे अच्छे भविष्यवक्ता कौन से हैं।
खुला स्रोत सॉफ्टवेयर
ओपन सोर्स सॉफ्टवेयर कम्प्यूटेशनल बायोलॉजी के लिए एक मंच प्रदान करता है जहां हर कोई अनुसंधान में विकसित सॉफ्टवेयर तक पहुंच और लाभ उठा सकता है। पीएलओएस उद्धृत करता है[citation needed] ओपन सोर्स सॉफ़्टवेयर के उपयोग के चार मुख्य कारण:
- पुनरुत्पादन: यह शोधकर्ताओं को जैविक डेटा के बीच संबंधों की गणना करने के लिए उपयोग की जाने वाली सटीक विधियों का उपयोग करने की अनुमति देता है।
- तेजी से विकास: डेवलपर्स और शोधकर्ताओं को मामूली कार्यों के लिए मौजूदा कोड को फिर से बनाने की जरूरत नहीं है। इसके बजाय वे बड़ी परियोजनाओं के विकास और कार्यान्वयन पर समय बचाने के लिए पहले से मौजूद कार्यक्रमों का उपयोग कर सकते हैं।
- बढ़ी हुई गुणवत्ता: एक ही विषय का अध्ययन करने वाले कई शोधकर्ताओं के इनपुट होने से आश्वासन की एक परत मिलती है कि त्रुटियाँ कोड में नहीं होंगी।
- दीर्घकालिक उपलब्धता: ओपन सोर्स प्रोग्राम किसी व्यवसाय या पेटेंट से बंधे नहीं हैं। यह उन्हें कई वेब पृष्ठों पर पोस्ट करने की अनुमति देता है और यह सुनिश्चित करता है कि वे भविष्य में उपलब्ध हों।[25]
अनुसंधान
कम्प्यूटेशनल बायोलॉजी से संबंधित कई बड़े सम्मेलन हैं। कुछ उल्लेखनीय उदाहरण आण्विक जीवविज्ञान के लिए इंटेलिजेंट सिस्टम, कम्प्यूटेशनल जीवविज्ञान पर यूरोपीय सम्मेलन और कम्प्यूटेशनल आण्विक जीवविज्ञान में अनुसंधान हैं।
कम्प्यूटेशनल जीव विज्ञान के लिए समर्पित कई पत्रिकाएँ भी हैं। कुछ उल्लेखनीय उदाहरणों में कम्प्यूटेशनल जीवविज्ञान के जर्नल और पीएलओएस कम्प्यूटेशनल बायोलॉजी शामिल हैं, एक सहकर्मी-समीक्षित ओपन एक्सेस जर्नल जिसमें कम्प्यूटेशनल जीव विज्ञान के क्षेत्र में कई उल्लेखनीय शोध परियोजनाएं हैं। वे सॉफ़्टवेयर पर समीक्षा प्रदान करते हैं, ओपन सोर्स सॉफ्टवेयर के लिए ट्यूटोरियल और आगामी कम्प्यूटेशनल जीव विज्ञान सम्मेलनों पर जानकारी प्रदर्शित करते हैं।[citation needed]
संबंधित क्षेत्र
कम्प्यूटेशनल जीव विज्ञान, जैव सूचना विज्ञान और गणितीय और सैद्धांतिक जीव विज्ञान जीवन विज्ञान की सूची के लिए सभी अंतःविषय दृष्टिकोण हैं जो गणित और सूचना विज्ञान जैसे मात्रात्मक विषयों से आते हैं। नेशनल इंस्टीट्यूट ऑफ हेल्थ कम्प्यूटेशनल/गणितीय जीव विज्ञान को जीव विज्ञान में सैद्धांतिक और प्रयोगात्मक प्रश्नों को संबोधित करने के लिए कम्प्यूटेशनल/गणितीय दृष्टिकोण के उपयोग के रूप में वर्णित करता है, और इसके विपरीत, जैव सूचना विज्ञान जटिल जीवन-विज्ञान डेटा को समझने के लिए सूचना विज्ञान के अनुप्रयोग के रूप में है।[1]
विशेष रूप से, एनआईएच परिभाषित करता है
Computational biology: The development and application of data-analytical and theoretical methods, mathematical modeling and computational simulation techniques to the study of biological, behavioral, and social systems.[1]
Bioinformatics: Research, development, or application of computational tools and approaches for expanding the use of biological, medical, behavioral or health data, including those to acquire, store, organize, archive, analyze, or visualize such data.[1]
जबकि प्रत्येक क्षेत्र अलग है, उनके इंटरफ़ेस पर महत्वपूर्ण ओवरलैप हो सकता है,[1]इतना अधिक कि कई लोगों के लिए, जैव सूचना विज्ञान और कम्प्यूटेशनल जीव विज्ञान ऐसे शब्द हैं जिनका परस्पर विनिमय किया जाता है।
कम्प्यूटेशनल जीव विज्ञान और विकासवादी संगणना शब्द का एक समान नाम है, लेकिन उन्हें भ्रमित नहीं होना चाहिए। कम्प्यूटेशनल जीव विज्ञान के विपरीत, विकासवादी संगणना जैविक डेटा के मॉडलिंग और विश्लेषण से संबंधित नहीं है। इसके बजाय यह विभिन्न प्रजातियों में विकास के विचारों के आधार पर एल्गोरिदम बनाता है। कभी-कभी जेनेटिक एल्गोरिद्म के रूप में संदर्भित, इस क्षेत्र का शोध कम्प्यूटेशनल जीवविज्ञान पर लागू किया जा सकता है। जबकि विकासवादी गणना स्वाभाविक रूप से कम्प्यूटेशनल जीव विज्ञान का हिस्सा नहीं है, कम्प्यूटेशनल विकासवादी जीव विज्ञान इसका एक उपक्षेत्र है।[26]
यह भी देखें
- Artificial life
- Bioinformatics
- Biological computing
- Biological simulation
- Biosimulation
- Biostatistics
- Computational audiology
- Computational chemistry
- Computational science
- Computational history
- DNA sequencing
- Functional genomics
- International Society for Computational Biology
- List of bioinformatics institutions
- List of biological databases
- Mathematical biology
- Monte Carlo method
- Molecular modeling
- Network biology
- Phylogenetics
- Proteomics
- Structural genomics
- Synthetic biology
- Systems biology
संदर्भ
- ↑ 1.0 1.1 1.2 1.3 1.4 "NIH working definition of bioinformatics and computational biology" (PDF). Biomedical Information Science and Technology Initiative. 17 July 2000. Archived from the original (PDF) on 5 September 2012. Retrieved 18 August 2012.
- ↑ "About the CCMB". Center for Computational Molecular Biology. Retrieved 18 August 2012.
- ↑ 3.0 3.1 3.2 Hogeweg, Paulien (7 March 2011). "The Roots of Bioinformatics in Theoretical Biology". PLOS Computational Biology. 3. 7 (3): e1002021. Bibcode:2011PLSCB...7E2021H. doi:10.1371/journal.pcbi.1002021. PMC 3068925. PMID 21483479.
- ↑ "The Human Genome Project". The Human Genome Project. 22 December 2020. Retrieved 13 April 2022.
- ↑ "Human Genome Project FAQ". Genome.gov (in English). Retrieved 2022-04-20.
- ↑ "T2T-CHM13v1.1 - Genome - Assembly - NCBI". www.ncbi.nlm.nih.gov. Retrieved 2022-04-20.
- ↑ "Genome List - Genome - NCBI". www.ncbi.nlm.nih.gov. Retrieved 2022-04-20.
- ↑ Bourne, Philip (2012). "Rise and Demise of Bioinformatics? Promise and Progress". PLOS Computational Biology. 8 (4): e1002487. Bibcode:2012PLSCB...8E2487O. doi:10.1371/journal.pcbi.1002487. PMC 3343106. PMID 22570600.
- ↑ "COSI Information". www.iscb.org. Retrieved 2022-04-21.
- ↑ Grenander, Ulf; Miller, Michael I. (1998-12-01). "Computational Anatomy: An Emerging Discipline". Q. Appl. Math. 56 (4): 617–694. doi:10.1090/qam/1668732.
- ↑ "Mathematical Biology | Faculty of Science". www.ualberta.ca. Retrieved 2022-04-18.
- ↑ 12.0 12.1 "The Sub-fields of Computational Biology". Ninh Laboratory of Computational Biology (in English). 2013-02-18. Retrieved 2022-04-18.
- ↑ "Genome Sequencing to the Rest of Us". Scientific American.
- ↑ "Computational Neuroscience | Neuroscience". www.bu.edu.
- ↑ 15.0 15.1 Sejnowski, Terrence; Christof Koch; Patricia S. Churchland (9 September 1988). "Computational Neuroscience". Science. 4871. 241 (4871): 1299–306. Bibcode:1988Sci...241.1299S. doi:10.1126/science.3045969. PMID 3045969.
- ↑ Dauvermann, Maria R.; Whalley, Heather C.; Schmidt, Andrã©; Lee, Graham L.; Romaniuk, Liana; Roberts, Neil; Johnstone, Eve C.; Lawrie, Stephen M.; Moorhead, Thomas W. J. (2014). "Computational Neuropsychiatry – Schizophrenia as a Cognitive Brain Network Disorder". Frontiers in Psychiatry. 5: 30. doi:10.3389/fpsyt.2014.00030. PMC 3971172. PMID 24723894.
- ↑ Tretter, F.; Albus, M. (December 2007). "'Computational Neuropsychiatry' of Working Memory Disorders in Schizophrenia: The Network Connectivity in Prefrontal Cortex - Data and Models". Pharmacopsychiatry. 40 (S 1): S2–S16. doi:10.1055/S-2007-993139. S2CID 18574327.
- ↑ Marin-Sanguino, A.; Mendoza, E. (2008). "Hybrid Modeling in Computational Neuropsychiatry". Pharmacopsychiatry. 41: S85–S88. doi:10.1055/s-2008-1081464. PMID 18756425.
- ↑ Price, Michael (2012-04-13). "Computational Biologists: The Next Pharma Scientists?".
- ↑ 20.0 20.1 Jessen, Walter (2012-04-15). "Pharma's shifting strategy means more jobs for computational biologists".
- ↑ Barbolosi, Dominique; Ciccolini, Joseph; Lacarelle, Bruno; Barlesi, Fabrice; Andre, Nicolas (2016). "Computational oncology--mathematical modelling of drug regimens for precision medicine". Nature Reviews Clinical Oncology. 13 (4): 242–254. doi:10.1038/nrclinonc.2015.204. PMID 26598946. S2CID 22492353.
- ↑ Yakhini, Zohar (2011). "Cancer Computational Biology". BMC Bioinformatics. 12: 120. doi:10.1186/1471-2105-12-120. PMC 3111371. PMID 21521513.
- ↑ [1]
- ↑ Koschützki, Dirk; Schreiber, Falk (2008-05-15). "Centrality Analysis Methods for Biological Networks and Their Application to Gene Regulatory Networks". Gene Regulation and Systems Biology. 2: 193–201. doi:10.4137/grsb.s702. ISSN 1177-6250. PMC 2733090. PMID 19787083.
- ↑ Prlić, Andreas; Lapp, Hilmar (2012). "The PLOS Computational Biology Software Section". PLOS Computational Biology. 8 (11): e1002799. Bibcode:2012PLSCB...8E2799P. doi:10.1371/journal.pcbi.1002799. PMC 3510099.
- ↑ Foster, James (June 2001). "Evolutionary Computation". Nature Reviews Genetics. 2 (6): 428–436. doi:10.1038/35076523. PMID 11389459. S2CID 205017006.