क्रायोजेनिक इलेक्ट्रॉन माइक्रोस्कोपी
क्रायोजेनिक इलेक्ट्रॉन माइक्रोस्कोपी (क्रायो-ईएम) एक क्रायोमाइक्रोस्कोपी प्रणाली है जिसे क्रायोजेनिक तापमान पर ठंडा किए गए नमूनों पर प्रयुक्त किया जाता है। जैविक नमूनों के लिए, संरचना को अनाकार बर्फ के वातावरण में एम्बेड करके संरक्षित किया जाता है। जलीय नमूना समाधान एक ग्रिड-जाल और तरल ईथेन या तरल ईथेन और प्रोपेन के मिश्रण में जमे हुए जमे हुए पर प्रयुक्त होता है।[1] जबकि प्रणाली का विकास 1970 के दशक में प्रारंभ हुआ था, अनुवेदक प्रौद्योगिकी और सॉफ्टवेयर एल्गोरिदम में वर्तमान प्रगति ने निकट-परमाणु विश्लेषण पर जैव-आणविक संरचनाओं के निर्धारण की अनुमति दी है।[2] इसने क्रिस्टलीकरण की आवश्यकता के बिना मैक्रोमोलेक्युलर संरचना निर्धारण के लिए एक्स - रे क्रिस्टलोग्राफी या एनएमआर स्पेक्ट्रोस्कोपी के विकल्प के रूप में दृष्टिकोण पर व्यापक ध्यान आकर्षित किया है।[3]
2017 में, रसायन विज्ञान में नोबेल पुरस्कार जैक्स डबोचेट, जोआचिम फ्रैंक और रिचर्ड हेंडरसन (जीवविज्ञानी) को समाधान में जैव-अणुओं के उच्च- विश्लेषण संरचना निर्धारण के लिए क्रायो-इलेक्ट्रॉन माइक्रोस्कोपी विकसित करने के लिए दिया गया था।[4]प्रकृति के तरीके ने 2015 में क्रायो-ईएम को वर्ष की विधि के रूप में नामित किया।[5]
फेडरल इंस्टीट्यूट ऑफ टेक्नोलॉजी, लॉज़ेन विश्वविद्यालय और जिनेवा विश्वविद्यालय ने नवंबर 2021 के अंत में डबोचेट सेंटर फ़ॉर इमेजिंग (डीसीआई) खोला, जिसमें क्रायोजेनिक इलेक्ट्रॉन माइक्रोस्कोपी की प्रणाली को सर्वोत्तम संभव तरीके से प्रयुक्त किया जाना है। और दूसरी ओर और विकसित [6] सार्स-कोव-2 ओमिक्रॉन वैरिएंट की पहली पहचान के एक महीने से भी कम समय में, डीसीआई के शोधकर्ता इसकी संरचना को परिभाषित करने, व्यक्तिगत टीकों को दरकिनार करने के लिए महत्वपूर्ण म्यूटेशन की पहचान करने और नए चिकित्सीय दृष्टिकोणों के लिए अंतर्दृष्टि प्रदान करने में सक्षम थे। <रेफरी नाम = स्विसइंफो>"वैज्ञानिक सूक्ष्मदर्शी का उपयोग करके ओमिक्रॉन वैरिएंट रहस्यों को उजागर करते हैं". swissinfo.ch. 2021-12-30. Retrieved 2022-04-30.</रेफरी>
ट्रांसमिशन इलेक्ट्रॉन क्रायोमाइक्रोस्कोपी
ट्रांसमिशन इलेक्ट्रॉन क्रायोमाइक्रोस्कोपी (क्रायो-टीईएम) ट्रांसमिशन इलेक्ट्रॉन माइक्रोस्कोपी प्रणाली है जिसका उपयोग संरचनात्मक जीव विज्ञान और सामग्री विज्ञान में किया जाता है।
- इलेक्ट्रॉन क्रायोटोमोग्राफी (क्रायो-ईटी), विशेष अनुप्रयोग जहां नमूनों को झुकाए जाने पर उनकी छवि बनाई जाती है
- इलेक्ट्रॉन क्रिस्टलोग्राफी, टीईएम का उपयोग करके ठोस पदार्थों में परमाणुओं की व्यवस्था निर्धारित करने की विधि
- माइक्रोक्रिस्टल इलेक्ट्रॉन विवर्तन,[7] 3डी क्रिस्टल से इलेक्ट्रॉन विवर्तन का उपयोग करके प्रोटीन, पेप्टाइड, कार्बनिक अणुओं और अकार्बनिक यौगिकों की संरचना निर्धारित करने की विधि[8][9][10]
- एकल कण विश्लेषण क्रायो-ईएम, मोनोडिस्पर्स नमूनों से प्रोटीन संरचना निर्धारित करने के लिए औसत विधि[11]
Cryo-TEM image of GroEL suspended in amorphous ice at 50000× magnification
Structure of alcohol oxidase from Pichia pastoris by Cryo-TEM
इतिहास
प्रारंभिक विकास
1960 के दशक में, उच्च ऊर्जा इलेक्ट्रॉन बीम के कारण विकिरण क्षति के कारण संरचना निर्धारण विधियों के लिए संचरण इलेक्ट्रॉन माइक्रोस्कोपी का उपयोग सीमित था। वैज्ञानिकों ने परिकल्पना की कि कम तापमान पर नमूनों की जांच करने से बीम-प्रेरित विकिरण क्षति कम हो जाएगी।[13] तरल हीलियम (−269 सेल्सियस|°C या 4 केल्विन या -452.2 फ़ारेनहाइट|°F) और तरल नाइट्रोजन (-195.79 °C या 77 K या -320 °F) दोनों को क्रायोजेन माना जाता था। 1980 में, इरविन नापेक और जैक्स डबोचेट ने क्रायोजेनिक तापमान पर बीम क्षति पर टिप्पणियों को साझा करते हुए टिप्पणियां प्रकाशित की:
<ब्लॉककोट>कार्बन फिल्म पर लगे पतले क्रिस्टल कमरे के तापमान की तुलना में 4 K पर 30 से 300 गुना अधिक बीम-प्रतिरोधी पाए गए... हमारे अधिकांश परिणामों को यह मानकर समझाया जा सकता है कि 4 K के क्षेत्र में क्रायोप्रोटेक्शन है दृढ़ता से तापमान पर निर्भर।[14]</ब्लॉककोट>
चूंकि, ये परिणाम प्रतिलिपि प्रस्तुत करने योग्य नहीं थे और केवल दो साल बाद प्रकृति (पत्रिका) में संशोधन प्रकाशित किए गए थे, जिसमें सूचित किया गया था कि बीम प्रतिरोध प्रारंभिक अनुमान से कम महत्वपूर्ण था। एल-वेलिन के मानक नमूनों के लिए 4K पर प्राप्त सुरक्षा दस गुना के करीब थी,[15] पहले जो कहा गया था, उससे कहीं अधिक थी।
1981 में, यूरोपीय आणविक जीवविज्ञान प्रयोगशाला के वैज्ञानिकों अलास्डेयर मैकडॉवल और जैक्स डबोचेट ने क्रायो-ईएम के पहले सफल कार्यान्वयन की सूचना दी।[16] मैकडॉवल और डबोचेट विट्रिफिकेशन शुद्ध पानी को पतली फिल्म में हाइड्रोफिलिक कार्बन फिल्म पर छिड़क कर शुद्ध करते हैं जो तेजी से क्रायोजेनिक्स (तरल प्रोपेन या तरल एटैन को 77 K तक ठंडा किया जाता है) में डुबोया गया था। अनाकार बर्फ की पतली परत 1 माइक्रोमीटर से कम मोटी थी और इलेक्ट्रॉन विवर्तन पैटर्न ने अनाकार/कांचयुक्त बर्फ की उपस्थिति की पुष्टि की। 1984 में, डबोचेट के समूह ने संरचनात्मक जीव विज्ञान में क्रायो-ईएम की शक्ति का प्रदर्शन कांच में रूपांतर एडेनोवाइरिडे टाइप 2, टी 4 बैक्टीरियोफेज, सेमलिकी वन विषाणु, बैक्टीरियोफेज सीबीके और वेसिकुलर-स्टोमाटाइटिस-वायरस के विश्लेषण के साथ किया।[17]
2017 रसायन विज्ञान में नोबेल पुरस्कार
"व्याख्या में जैव-अणुओं के उच्च-विश्लेषण संरचना निर्धारण के लिए क्रायो-इलेक्ट्रॉन माइक्रोस्कोपी विकसित करने के लिए" क्रायो-ईएम के जैव रसायन पर पड़ने वाले प्रभाव की मान्यता में, तीन वैज्ञानिकों, जैक्स डबोचेट, जोआचिम फ्रैंक और रिचर्ड हेंडरसन को रसायन विज्ञान में नोबेल पुरस्कार से सम्मानित किया गया था।[4]
एक्स-रे क्रिस्टलोग्राफी के लिए संभावित प्रतिद्वंद्वी
परंपरागत रूप से, जैविक अणुओं की 3डी संरचनाओं को निर्धारित करने के लिए एक्स-रे क्रिस्टलोग्राफी सबसे लोकप्रिय प्रणाली रही है।[18] चूंकि, क्रायो-ईएम में उपरोक्त सुधारों ने जैविक अणुओं के विवरण की जांच के लिए उपकरण के रूप में इसकी लोकप्रियता में वृद्धि की है। तुलना के रूप में, एक्स-रे क्रिस्टलोग्राफी का उपयोग 169,077 जैविक अणुओं (30 सितंबर, 2022 तक) की 3डी संरचनाओं को निर्धारित करने के लिए किया गया है, जबकि क्रायो-ईएम का उपयोग 12,647 पर कम जैविक अणुओं को निर्धारित करने के लिए किया गया है।[19]
चूंकि, नेचर (जर्नल) के अनुसार, कैम्ब्रिज विश्वविद्यालय ट्रांसमिशन इलेक्ट्रॉन माइक्रोस्कोपी के लिए अनुवेदक अधिकांशतः डायरेक्ट डिटेक्शन डिवाइस या डीडीडी के रूप में संदर्भित) के लिए अनुवेदकों में प्रगति[20] और एसपीटी लैबटेक द्वारा नमूना उत्पादन का स्वचालन[21] जैविक क्षेत्रों में उपयोग में वृद्धि हुई है,[22] क्रायो-ईएम को संभावित प्रतिद्वंद्वी बनाना।
एक्स-रे क्रिस्टलोग्राफी का विश्लेषण क्रिस्टल शुद्धता द्वारा सीमित है,[23] और जैविक अणुओं को क्रिस्टलीय अवस्था में ले जाने में बहुत समय लग सकता है, जिसमें महीनों या वर्षों तक का समय लग सकता है।[22]चूंकि क्रायो-ईएम के लिए नमूना तैयार करना अभी भी श्रमसाध्य है, <रेफरी नाम = भेला 515-519>Nakane T, Kotecha A, Sente A, McMullan G, Masiulis S, Brown PM, et al. (November 2020). "परमाणु विभेदन पर एकल-कण क्रायो-ईएम". Nature. 587 (7832): 152–156. Bibcode:2020Natur.587..152N. doi:10.1038/s41586-020-2829-0. PMC 7611073. PMID 33087931.</रेफ> इसमें ये मुद्दे नहीं हैं क्योंकि इसमें क्रिस्टल बनाने के लिए नमूने की आवश्यकता नहीं होती है, बल्कि क्रायो-ईएम के नमूने फ्लैश-फ्रोजन होते हैं और उनके निकट-देशी राज्यों में जांच की जाती है। रेफरी नाम = :2 >Wang HW, Wang JW (2016-08-20). "कैसे क्रायो-इलेक्ट्रॉन माइक्रोस्कोपी और एक्स-रे क्रिस्टलोग्राफी एक दूसरे के पूरक हैं". Protein Science. 26 (1): 32–39. doi:10.1002/pro.3022. PMC 5192981. PMID 27543495.</रेफरी>
प्रोटिओपेडिया के अनुसार, प्रोटीन डाटा बैंक पर एक्स-रे क्रिस्टलोग्राफी (19 मई, 2019 तक) द्वारा प्राप्त औसत विश्लेषण 2.05 Å है,[23]और रिकॉर्ड पर प्राप्त उच्चतम विश्लेषण (30 सितंबर, 2022 तक) 0.48 ए है।[24] 2020 तक, क्रायो-ईएम द्वारा निर्धारित अधिकांश प्रोटीन संरचनाएं 3–4 Å के कम विश्लेषण पर हैं।[25] चूंकि, 2020 तक, सबसे अच्छा क्रायो-ईएम विश्लेषण 1.22 Å पर अंकित किया गया है,[26] कुछ स्थितियोंं में यह विश्लेषण में एक प्रतियोगी बन गया है।
सहसंबंधी प्रकाश क्रायो-टीईएम और क्रायो-ईटी
2019 में, सहसंबंधी प्रकाश-इलेक्ट्रॉन माइक्रोस्कोपी क्रायो-टीईएम और क्रायो-ईटी का उपयोग न्यूरोनल कोशिकाओं में टनलिंग नैनोट्यूब (टीएनटी) का निरीक्षण करने के लिए किया गया था।[27]
स्कैनिंग इलेक्ट्रॉन क्रायोमाइक्रोस्कोपी
स्कैनिंग इलेक्ट्रॉन क्रायोमाइक्रोस्कोपी (क्रायो ईएम) स्कैनिंग इलेक्ट्रॉन माइक्रोस्कोपी प्रणाली है जिसमें क्रायोजेनिक कक्ष में स्कैनिंग इलेक्ट्रॉन माइक्रोस्कोप कोल्ड स्टेज होता है।
यह भी देखें
- क्रायोफिक्सेशन
- इलेक्ट्रॉन टोमोग्राफी (ईटी)
संदर्भ
- ↑ Tivol WF, Briegel A, Jensen GJ (October 2008). "An improved cryogen for plunge freezing". Microscopy and Microanalysis. 14 (5): 375–379. Bibcode:2008MiMic..14..375T. doi:10.1017/S1431927608080781. PMC 3058946. PMID 18793481.
- ↑ Cheng Y, Grigorieff N, Penczek PA, Walz T (April 2015). "A primer to single-particle cryo-electron microscopy". Cell. 161 (3): 438–449. doi:10.1016/j.cell.2015.03.050. PMC 4409659. PMID 25910204.
- ↑ Stoddart C (1 March 2022). "Structural biology: How proteins got their close-up". Knowable Magazine. doi:10.1146/knowable-022822-1. Retrieved 25 March 2022.
- ↑ 4.0 4.1 "The Nobel Prize in Chemistry 2017". NobelPrize.org (in English). Retrieved 2022-09-30.
- ↑ Doerr A (January 2017). "Cryo-electron tomography". Nature Methods. 14 (1): 34. doi:10.1038/nmeth.4115. ISSN 1548-7091. S2CID 27162203.
- ↑ "यूएनआईजीई, यूएनआईएल और ईपीएफएल के परिसरों में डबोचेट सेंटर फॉर इमेजिंग (डीसीआई) का उद्घाटन". unige.ch. 2021-11-30. Retrieved 2022-04-30.
- ↑ Nannenga BL, Shi D, Leslie AG, Gonen T (September 2014). "High-resolution structure determination by continuous-rotation data collection in MicroED". Nature Methods. 11 (9): 927–930. doi:10.1038/nmeth.3043. PMC 4149488. PMID 25086503.
- ↑ Jones CG, Martynowycz MW, Hattne J, Fulton TJ, Stoltz BM, Rodriguez JA, et al. (November 2018). "The CryoEM Method MicroED as a Powerful Tool for Small Molecule Structure Determination". ACS Central Science. 4 (11): 1587–1592. doi:10.1021/acscentsci.8b00760. PMC 6276044. PMID 30555912.
- ↑ de la Cruz MJ, Hattne J, Shi D, Seidler P, Rodriguez J, Reyes FE, et al. (February 2017). "Atomic-resolution structures from fragmented protein crystals with the cryoEM method MicroED". Nature Methods. 14 (4): 399–402. doi:10.1038/nmeth.4178. PMC 5376236. PMID 28192420.
- ↑ Gruene T, Wennmacher JT, Zaubitzer C, Holstein JJ, Heidler J, Fecteau-Lefebvre A, et al. (December 2018). "Rapid Structure Determination of Microcrystalline Molecular Compounds Using Electron Diffraction". Angewandte Chemie. 57 (50): 16313–16317. doi:10.1002/anie.201811318. PMC 6468266. PMID 30325568.
- ↑ Cheng Y (August 2018). "Single-particle cryo-EM-How did it get here and where will it go". Science. 361 (6405): 876–880. Bibcode:2018Sci...361..876C. doi:10.1126/science.aat4346. PMC 6460916. PMID 30166484.
- ↑ Xiao, C., Fischer, M.G., Bolotaulo, D.M., Ulloa-Rondeau, N., Avila, G.A., and Suttle, C.A. (2017) "Cryo-EM reconstruction of the Cafeteria roenbergensis virus capsid suggests novel assembly pathway for giant viruses". Scientific Reports, 7: 5484. doi:10.1038/s41598-017-05824-w.
- ↑ Dubochet J, Knapek E (April 2018). "Ups and downs in early electron cryo-microscopy". PLOS Biology. 16 (4): e2005550. doi:10.1371/journal.pbio.2005550. PMC 5929567. PMID 29672565.
- ↑ Knapek E, Dubochet J (August 1980). "Beam damage to organic material is considerably reduced in cryo-electron microscopy". Journal of Molecular Biology. 141 (2): 147–161. doi:10.1016/0022-2836(80)90382-4. PMID 7441748.
- ↑ Newmark P (30 September 1982). "Cryo-transmission microscopy Fading hopes". Nature. 299 (5882): 386–387. Bibcode:1982Natur.299..386N. doi:10.1038/299386c0.
- ↑ Dubochet J, McDowall AW (December 1981). "Vitrification of Pure Water for Electron Microscopy". Journal of Microscopy (in English). 124 (3): 3–4. doi:10.1111/j.1365-2818.1981.tb02483.x.
- ↑ Adrian M, Dubochet J, Lepault J, McDowall AW (March 1984). "Cryo-electron microscopy of viruses" (PDF). Nature. 308 (5954): 32–36. Bibcode:1984Natur.308...32A. doi:10.1038/308032a0. PMID 6322001. S2CID 4319199.
- ↑ Smyth MS, Martin JH (February 2000). "x ray crystallography". Molecular Pathology. 53 (1): 8–14. doi:10.1136/mp.53.1.8. PMC 1186895. PMID 10884915.
- ↑ "RCSB PDB Statistics". www.rcsb.org. Retrieved 2020-09-30.
- ↑ Callaway E (September 2015). "The revolution will not be crystallized: a new method sweeps through structural biology". Nature. 525 (7568): 172–174. Bibcode:2015Natur.525..172C. doi:10.1038/525172a. PMID 26354465.
- ↑ Baker M (September 2018). "Cryo-electron microscopy shapes up". Nature. 561 (7724): 565–567. Bibcode:2018Natur.561..565B. doi:10.1038/d41586-018-06791-6. PMID 30254359.
- ↑ 22.0 22.1 Callaway E (February 2020). "Revolutionary cryo-EM is taking over structural biology". Nature. 578 (7794): 201. Bibcode:2020Natur.578..201C. doi:10.1038/d41586-020-00341-9. PMID 32047310.
- ↑ 23.0 23.1 "Resolution - Proteopedia, life in 3D". proteopedia.org. Retrieved 2020-10-27.
- ↑ Schmidt A, Teeter M, Weckert E, Lamzin VS (April 2011). "Crystal structure of small protein crambin at 0.48 Å resolution". Acta Crystallographica. Section F, Structural Biology and Crystallization Communications. 67 (Pt 4): 424–428. doi:10.1107/S1744309110052607. PMC 3080141. PMID 21505232.
- ↑ Yip KM, Fischer N, Paknia E, Chari A, Stark H (November 2020). "Atomic-resolution protein structure determination by cryo-EM". Nature. 587 (7832): 157–161. Bibcode:2020Natur.587..157Y. doi:10.1038/s41586-020-2833-4. PMID 33087927. S2CID 224823207.
- ↑ Cite error: Invalid
<ref>tag; no text was provided for refs namedभेला - ↑ Sartori-Rupp A, Cordero Cervantes D, Pepe A, Gousset K, Delage E, Corroyer-Dulmont S, et al. (January 2019). "Correlative cryo-electron microscopy reveals the structure of TNTs in neuronal cells". Nature Communications. 10 (1): 342. Bibcode:2019NatCo..10..342S. doi:10.1038/s41467-018-08178-7. PMC 6341166. PMID 30664666.


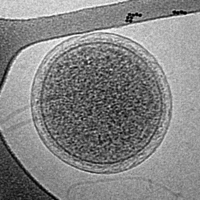
![Cryo-electron micrograph of the CroV giant marine virus (scale bar represents 200 nm)[12]](https://upload.wikimedia.org/wikipedia/commons/thumb/6/6e/CroV_TEM_%28cropped%29.jpg/200px-CroV_TEM_%28cropped%29.jpg)