सिस्टम जीवविज्ञान: Difference between revisions
No edit summary |
No edit summary |
||
| Line 3: | Line 3: | ||
[[File:Genomics GTL Pictorial Program.jpg|thumb|जीव विज्ञान के लिए प्रणाली दृष्टिकोण का उदाहरण]]'''प्रणाली [[ जीवविज्ञान |जीवविज्ञान]]''' समष्टि जैविक प्रणालियों का [[कम्प्यूटेशनल मॉडलिंग]] और [[गणितीय]] विश्लेषण और मॉडलिंग है। यह अध्ययन का जीव विज्ञान-आधारित अंतःविषय क्षेत्र है जो जैविक अनुसंधान के लिए समग्र दृष्टिकोण (अधिक पारंपरिक न्यूनीकरणवादी के अतिरिक्त समग्रता) का उपयोग करते हुए, जैविक प्रणालियों के अंदर समष्टि अंतःक्रियाओं पर ध्यान केंद्रित करता है।<ref><रेफ नाम = तवासोली 487-500 >{{Cite journal|last1=Tavassoly|first1=Iman|last2=Goldfarb|first2=Joseph|last3=Iyengar|first3=Ravi|date=2018-10-04|title=सिस्टम बायोलॉजी प्राइमर: बुनियादी तरीके और दृष्टिकोण|journal=Essays in Biochemistry|volume=62|issue=4|pages=487–500|doi=10.1042/EBC20180003|issn=0071-1365|pmid=30287586|s2cid=52922135}}<nowiki></ref> | [[File:Genomics GTL Pictorial Program.jpg|thumb|जीव विज्ञान के लिए प्रणाली दृष्टिकोण का उदाहरण]]'''प्रणाली [[ जीवविज्ञान |जीवविज्ञान]]''' समष्टि जैविक प्रणालियों का [[कम्प्यूटेशनल मॉडलिंग]] और [[गणितीय]] विश्लेषण और मॉडलिंग है। यह अध्ययन का जीव विज्ञान-आधारित अंतःविषय क्षेत्र है जो जैविक अनुसंधान के लिए समग्र दृष्टिकोण (अधिक पारंपरिक न्यूनीकरणवादी के अतिरिक्त समग्रता) का उपयोग करते हुए, जैविक प्रणालियों के अंदर समष्टि अंतःक्रियाओं पर ध्यान केंद्रित करता है।<ref><रेफ नाम = तवासोली 487-500 >{{Cite journal|last1=Tavassoly|first1=Iman|last2=Goldfarb|first2=Joseph|last3=Iyengar|first3=Ravi|date=2018-10-04|title=सिस्टम बायोलॉजी प्राइमर: बुनियादी तरीके और दृष्टिकोण|journal=Essays in Biochemistry|volume=62|issue=4|pages=487–500|doi=10.1042/EBC20180003|issn=0071-1365|pmid=30287586|s2cid=52922135}}<nowiki></ref> | ||
विशेष रूप से वर्ष 2000 के बाद से, इस अवधारणा का जीव विज्ञान में विभिन्न संदर्भों में व्यापक रूप से उपयोग किया गया है। [[मानव जीनोम परियोजना]] जीव विज्ञान में व्यावहारिक प्रणालियों की सोच का उदाहरण है जिसने आनुवंशिकी के जैविक क्षेत्र में समस्याओं पर काम करने के नए, सहयोगात्मक विधियो को उत्पन्न करता है।<ref>रेफरी>{{cite book|last1=Zewail|first1=Ahmed|title=भौतिक जीवविज्ञान: परमाणुओं से औषधि तक|date=2008|publisher=Imperial College Press|page=339}}<nowiki></ref> | विशेष रूप से वर्ष 2000 के बाद से, इस अवधारणा का जीव विज्ञान में विभिन्न संदर्भों में व्यापक रूप से उपयोग किया गया है। [[मानव जीनोम परियोजना]] जीव विज्ञान में व्यावहारिक प्रणालियों की सोच का उदाहरण है जिसने आनुवंशिकी के जैविक क्षेत्र में समस्याओं पर काम करने के नए, सहयोगात्मक विधियो को उत्पन्न करता है।<ref>रेफरी>{{cite book|last1=Zewail|first1=Ahmed|title=भौतिक जीवविज्ञान: परमाणुओं से औषधि तक|date=2008|publisher=Imperial College Press|page=339}}<nowiki></ref> प्रणाली जीवविज्ञान का उद्देश्य अनपेक्षित विशेषताएं, कोशिका ([[जीव]] विज्ञान) के गुणों, ऊतक (जीव विज्ञान) और [[प्रणाली]] के रूप में कार्य करने वाले जीवों का मॉडल और खोज करना है, जिसका सैद्धांतिक विवरण केवल प्रणाली जीवविज्ञान की विधियो का उपयोग करके संभव है। .<ref><संदर्भ नाम = तवासोली 487-500 /> रेफरी>{{Cite book|title=जीवों पर परिप्रेक्ष्य - स्प्रिंगर|last1=Longo|first1=Giuseppe|last2=Montévil|first2=Maël|doi=10.1007/978-3-642-35938-5|series=Lecture Notes in Morphogenesis|year=2014|isbn=978-3-642-35937-8|s2cid=27653540}}<nowiki> | ||
प्रणाली जीवविज्ञान का उद्देश्य | |||
</ref> | </ref> | ||
Revision as of 21:31, 22 July 2023
| Complex systems |
|---|
| Topics |
प्रणाली जीवविज्ञान समष्टि जैविक प्रणालियों का कम्प्यूटेशनल मॉडलिंग और गणितीय विश्लेषण और मॉडलिंग है। यह अध्ययन का जीव विज्ञान-आधारित अंतःविषय क्षेत्र है जो जैविक अनुसंधान के लिए समग्र दृष्टिकोण (अधिक पारंपरिक न्यूनीकरणवादी के अतिरिक्त समग्रता) का उपयोग करते हुए, जैविक प्रणालियों के अंदर समष्टि अंतःक्रियाओं पर ध्यान केंद्रित करता है।[1]
विशेष रूप से वर्ष 2000 के बाद से, इस अवधारणा का जीव विज्ञान में विभिन्न संदर्भों में व्यापक रूप से उपयोग किया गया है। मानव जीनोम परियोजना जीव विज्ञान में व्यावहारिक प्रणालियों की सोच का उदाहरण है जिसने आनुवंशिकी के जैविक क्षेत्र में समस्याओं पर काम करने के नए, सहयोगात्मक विधियो को उत्पन्न करता है।[2] प्रणाली जीवविज्ञान का उद्देश्य अनपेक्षित विशेषताएं, कोशिका (जीव विज्ञान) के गुणों, ऊतक (जीव विज्ञान) और प्रणाली के रूप में कार्य करने वाले जीवों का मॉडल और खोज करना है, जिसका सैद्धांतिक विवरण केवल प्रणाली जीवविज्ञान की विधियो का उपयोग करके संभव है। .[3]
इनमें समान्यत: उपापचय नेटवर्क या सेल सिग्नलिंग नेटवर्क सम्मिलित होते हैं।[4] [5]
अवलोकन
प्रणाली जीवविज्ञान पर कई अलग-अलग पहलुओं से विचार किया जा सकता है।
अध्ययन के क्षेत्र के रूप में विशेष रूप से, जैविक प्रणालियों के घटकों के बीच इंटरैक्शन का अध्ययन, और ये इंटरैक्शन उस प्रणाली के कार्य और व्यवहार को कैसे उत्पन्न कर देती है (उदाहरण के लिए, उपापचय पथ या दिल की धड़कन में एंजाइमों और मेटाबोलाइट्स).[6][7][8]
एक प्रतिमान के रूप में, प्रणाली जीवविज्ञान को समान्यत: तथाकथित न्यूनीकरणवादी प्रतिमान (जैविक संगठन) के विपरीत परिभाषित किया जाता है चूंकि यह वैज्ञानिक पद्धति के अनुरूप है। दो प्रतिमानों के बीच अंतर को इन उद्धरणों में संदर्भित किया गया है: न्यूनीकरणवाद दृष्टिकोण ने अधिकांश घटकों और कई इंटरैक्शन की सफलतापूर्वक पहचान की है, किन्तु दुर्भाग्य से, यह समझने के लिए कोई ठोस अवधारणा या विधियां प्रदान नहीं करता है कि प्रणाली गुण कैसे उभरते हैं ... का बहुलवाद जैविक नेटवर्क में कारणों और प्रभावों को मात्रात्मक उपायों के माध्यम से, साथ कई घटकों का अवलोकन करके और गणितीय मॉडल के साथ रिगोरयस डेटा एकीकरण द्वारा उत्तम विधि से संबोधित किया जाता है। (सॉयर एट अल.)[9] प्रणाली बायोलॉजी... अलग करने के अतिरिक्त साथ रखने घटाने के अतिरिक्त एकीकरण के बारे में है। इसके लिए आवश्यक है कि हम एकीकरण के बारे में सोचने के ऐसे विधि विकसित करें जो हमारे न्यूनतावादी कार्यक्रमों के समान कठोर हों, किन्तु भिन्न हों। ...इसका अर्थ है, शब्द के पूर्ण अर्थ में, हमारे दर्शन को बदलना है। (डेनिस नोबल)[8]
अनुसंधान करने के लिए उपयोग किए जाने वाले परिचालन प्रोटोकॉल (प्राकृतिक विज्ञान) की श्रृंखला के रूप में, अर्थात् जैविक प्रणाली के बारे में विशिष्ट परीक्षण योग्य परिकल्पनाओं का प्रस्ताव करने के लिए सिद्धांत, गणितीय मॉडल या कम्प्यूटेशनल मॉडलिंग से बना चक्र, प्रयोगात्मक सत्यापन, और फिर नए अधिग्रहीत मात्रात्मक विवरण का उपयोग करना कम्प्यूटेशनल मॉडल या सिद्धांत को परिष्कृत करने के लिए कोशिकाएँ या कोशिका प्रक्रियाएँ है [10] चूँकि उद्देश्य एक प्रणाली में अंतःक्रियाओं का मॉडल है प्रायोगिक तकनीकें जो प्रणाली जीव विज्ञान के लिए सबसे उपयुक्त हैं वे वह हैं जो प्रणाली-व्यापी हैं और यथासंभव पूर्ण होने का प्रयास करती हैं। इसलिए, मॉडल के निर्माण और सत्यापन के लिए मात्रात्मक डेटा एकत्र करने के लिए ट्रांसक्रिप्टोमिक्स, उपापचय , प्रोटिओमिक्स और उच्च परिणाम स्क्रीनिंग या हाई-थ्रूपुट विधियो का उपयोग किया जाता है।[11]
आणविक जीव विज्ञान में गतिशील प्रणाली सिद्धांत के अनुप्रयोग के रूप से वास्तव में अध्ययन की गई प्रणालियों की गतिशीलता पर ध्यान केंद्रित करना प्रणाली जीव विज्ञान और जैव सूचना विज्ञान के बीच मुख्य वैचारिक अंतर है।[12]
अंतःविषय उपकरणों और कर्मियों का उपयोग करके विविध प्रयोगात्मक स्रोतों से जैविक प्रणालियों में इंटरैक्शन के बारे में समष्टि डेटा के एकीकरण को आगे बढ़ाने की रणनीति द्वारा परिभाषित सामाजिक-वैज्ञानिक विवादों की घटना के रूप में उपयोग किया जाता है।[13]
इतिहास
प्रणाली जीवविज्ञान की प्रारंभिक 2000 के आसपास विज्ञान के नए क्षेत्र के रूप में हुई थी जब कम्प्यूटेशनल प्रकार के लोगों को लुभाने के प्रयास में सिएटल में प्रणाली जीवविज्ञान संस्थान की स्थापना की गई थी, जिनके बारे में यह अनुभव किया गया था कि वे विश्वविद्यालय की शैक्षणिक सेटिंग्स के प्रति आकर्षित नहीं थे। संस्थान के पास इस बात की स्पष्ट परिभाषा नहीं थी कि क्षेत्र वास्तव में क्या था: सामान्यतः नए विधियो से जीव विज्ञान का समग्र अध्ययन करने के लिए कंप्यूटर का उपयोग करने के लिए विभिन्न क्षेत्रों के लोगों को साथ लाना है।[14] हार्वर्ड मेडिकल स्कूल में प्रणाली जीवविज्ञान विभाग 2003 में प्रारंभ किया गया था।[15] 2006 में यह अनुमान लगाया गया था कि बहुत फैशनेबल नई अवधारणा से उत्पन्न चर्चा के कारण सभी प्रमुख विश्वविद्यालयों को प्रणाली जीवविज्ञान विभाग की आवश्यकता होगी जिससे कंप्यूटर प्रोग्रामिंग और जीव विज्ञान में थोड़ी सी क्षमता वाले स्नातकों के लिए करियर उपलब्ध हो सकेगा।[14] 2006 में राष्ट्रीय विज्ञान संस्था ने संपूर्ण कोशिका का गणितीय मॉडल बनाने की चुनौती सामने रखी गई। 2012 में माइकोप्लाज्मा जेनिटलियम का पहला पूर्ण-कोशिका मॉडल न्यूयॉर्क में माउंट सिनाई स्कूल ऑफ मेडिसिन में कर्र प्रयोगशाला द्वारा प्राप्त किया गया था। संपूर्ण-कोशिका मॉडल आनुवंशिक उत्परिवर्तन के उत्तर में एम. जेनिटेलियम कोशिकाओं की व्यवहार्यता की पूर्वानुमान करने में सक्षम है।[16]
एक विशिष्ट अनुशासन के रूप में प्रणाली जीवविज्ञान का पूर्ववर्ती अग्रदूत प्रणाली सिद्धांतकार मिहाज्लो मेसारोविक द्वारा 1966 में क्लीवलैंड, ओहियो में केस वेस्टर्न रिजर्व यूनिवर्सिटी में प्रणाली सिद्धांत और जीवविज्ञान शीर्षक से अंतरराष्ट्रीय संगोष्ठी के साथ हो सकता है। मेसारोविक ने पूर्वानुमान की कि संभवतः भविष्य में प्रणाली जीवविज्ञान जैसी कोई चीज़ होगी।[17][18] अन्य प्रारंभिक अग्रदूत जिन्होंने इस दृष्टिकोण पर ध्यान केंद्रित किया कि जीव विज्ञान का विश्लेषण भागों के सरल संग्रह के अतिरिक्त प्रणाली के रूप में किया जाना चाहिए, वे थे मेटाबोलिक नियंत्रण विश्लेषण, जिसे हेनरिक कैसर और जिम बर्न्स द्वारा विकसित किया गया था।[19] बाद में पूरी तरह से संशोधित किया गया था,[20] और रेनहार्ट हेनरिक और टॉम रैपोपोर्ट,[21] और जैव रासायनिक प्रणाली सिद्धांत माइकल सावेग्यू द्वारा विकसित की गई थी[22][23][24]
1960 के दशक में रॉबर्ट रोसेन (सैद्धांतिक जीवविज्ञानी) के अनुसार, 20वीं सदी की प्रारंभिक तक समग्र जीव विज्ञान अप्रचलित हो गया था, क्योंकि आणविक रसायन विज्ञान पर प्रभावित अधिक अनुभवजन्य विज्ञान लोकप्रिय हो गया था।[18] चालीस साल बाद 2006 में उनकी बात दोहराते हुए क्लिंग लिखते हैं कि 20वीं सदी में आणविक जीव विज्ञान की सफलता ने समग्र कम्प्यूटेशनल विधियो को दबा दिया था।[14] 2011 तक राष्ट्रीय स्वास्थ्य संस्थान ने संयुक्त राज्य अमेरिका में दस से अधिक प्रणाली जीवविज्ञान केंद्रों को समर्थन देने के लिए अनुदान राशि उपलब्ध कराई थी,[25] किन्तु 2012 तक हंटर लिखते हैं कि प्रणाली जीवविज्ञान को अपनी पूरी क्षमता प्राप्त करने के लिए अभी भी कुछ न कुछ करना शेष है। तथापि, समर्थकों को उम्मीद थी कि यह भविष्य में बार और अधिक उपयोगी सिद्ध हो सकता है।[26]
प्रणाली जीवविज्ञान के विकास में एक महत्वपूर्ण मील का पत्थर अंतर्राष्ट्रीय प्रोजेक्ट फिजियोम बन गया है।
संबद्ध अनुशासन

प्रणाली जीवविज्ञान की व्याख्या के अनुसार अंतःविषय उपकरणों का उपयोग करके बड़े डेटा समूह का उपयोग करना विशिष्ट अनुप्रयोग मेटाबोलॉमिक्स है, जो जीव, कोशिका या ऊतक स्तर पर प्रणाली में सभी उपापचय उत्पादों, मेटाबोलाइट्स का पूरा समूह है।[27]
आइटम जो कंप्यूटर डेटाबेस हो सकते हैं उनमें सम्मिलित हैं: एपिजेनोमिक्स, फेनोटाइप में जीव संबंधी भिन्नता क्योंकि यह अपने जीवन काल के समय बदलता है; जीनोमिक्स, ऑर्गेनिज्मल डिऑक्सीराइबोन्यूक्लिक अम्ल (डीएनए) अनुक्रम, जिसमें इंट्रा-ऑर्गेनिज्मल सेल विशिष्ट भिन्नता सम्मिलित है। (अथार्त , टेलोमेयर लंबाई भिन्नता); एपिजीनोमिक्स/एपिजेनेटिक्स, ऑर्गेनिज्मल और संबंधित कोशिका विशिष्ट ट्रांसक्रिप्टोमिक विनियमन कारक जो जीनोमिक अनुक्रम में अनुभवजन्य रूप से कोडित नहीं हैं। (अथार्त , डीएनए मिथाइलेशन, हिस्टोन एसिटिलेशन और डीएसिटिलेशन, आदि); डीएनए माइक्रोएरे द्वारा ट्रांसक्रिपटॉमिक्स, जीव, ऊतक या संपूर्ण कोशिका जीन अभिव्यक्ति माप या जीन अभिव्यक्ति का क्रमिक विश्लेषण; इंटरफेरोमिक्स, ऑर्गैज़्मल, टिशू, या सेल-लेवल ट्रांस्क्रिप्ट सुधार कारक (अथार्त , आरएनए हस्तक्षेप), प्रोटिओमिक्स, ऑर्गैज़्मल, टिशू, या सेल स्तर पर प्रोटीन और पेप्टाइड्स का माप दो-आयामी जेल वैद्युतकणसंचलन, मास स्पेक्ट्रोमेट्री या बहु-आयामी प्रोटीन पहचान विधियो के माध्यम से (मास स्पेक्ट्रोमेट्री के साथ युग्मित उन्नत उच्च-प्रदर्शन तरल क्रोमैटोग्राफी प्रणाली ) उप-विषयों में फॉस्फोप्रोटिओमिक्स, ग्लाइकोप्रोटिओमिक्स और रासायनिक रूप से संशोधित प्रोटीन का पता लगाने के अन्य विधि सम्मिलित हैं; जो कार्बोहाइड्रेट का ग्लाइकोमिक्स, जीव, ऊतक, या कोशिका-स्तरीय माप; लिपिडोमिक्स, जीव, ऊतक, या कोशिका स्तर पर लिपिड का माप है।
कोशिका के अंदर आणविक अंतःक्रियाओं का भी अध्ययन किया जाता है, इसे इंटरेक्टॉमिक्स कहा जाता है।[28] अध्ययन के इस क्षेत्र में अनुशासन प्रोटीन-प्रोटीन अंतःक्रिया है, चूंकि अंतःक्रिया विज्ञान में अन्य अणुओं की अंतःक्रिया सम्मिलित है। न्यूरोइलेक्ट्रोडायनामिक्स, जहां गतिशील प्रणाली के रूप में कंप्यूटर या मस्तिष्क के कंप्यूटिंग कार्य का उसके (जैव)भौतिक तंत्र के साथ अध्ययन किया जाता है;[29] और फ्लक्सोमिक्स, जैविक प्रणाली (कोशिका, ऊतक या जीव) में उपापचय प्रतिक्रियाओं की दर का माप है।[27]
प्रणाली जीवविज्ञान समस्या से सामना करने के लिए दो मुख्य दृष्टिकोण हैं। ये ऊपर से नीचे और नीचे से ऊपर का दृष्टिकोण हैं। ऊपर से नीचे का दृष्टिकोण यथासंभव प्रणाली को ध्यान में रखता है और अधिकत्तर सीमा तक प्रयोगात्मक परिणामों पर निर्भर करता है। आरएनए-Seq तकनीक प्रायोगिक टॉप डाउन दृष्टिकोण का उदाहरण है। इसके विपरीत, प्रयोगात्मक डेटा को सम्मिलित करते हुए विस्तृत मॉडल बनाने के लिए नीचे से ऊपर दृष्टिकोण का उपयोग किया जाता है। बॉटम अप दृष्टिकोण का उदाहरण सरल जीन नेटवर्क का वर्णन करने के लिए परिपथ मॉडल का उपयोग है।[30]
एमआरएनए, प्रोटीन और पोस्ट-ट्रांसलेशनल संशोधनों में गतिशील परिवर्तनों को पकड़ने के लिए विभिन्न विधियो का उपयोग किया जाता है। यांत्रिक जीव विज्ञान, सभी स्तरों पर बल और भौतिक गुण, अन्य नियामक तंत्रों के साथ उनकी परस्पर क्रिया[31] बायोसेमियोटिक्स , किसी जीव या अन्य बायोसिस्टम के संकेत संबंधों की प्रणाली का विश्लेषण; फिजियोमिक्स, जीव विज्ञान में फिजियोम का व्यवस्थित अध्ययन किया जाता है।
कैंसर प्रणाली जीवविज्ञान प्रणाली जीवविज्ञान दृष्टिकोण का उदाहरण है, जिसे अध्ययन की विशिष्ट वस्तु (ट्यूमरोजेनेसिस और कैंसर उपचार) द्वारा अलग किया जा सकता है। यह विशिष्ट डेटा (रोगी के नमूने, रोगी के ट्यूमर के नमूनों में कैंसर जीनोम अनुक्रमण को चिह्नित करने पर विशेष ध्यान देने के साथ उच्च-थ्रूपुट डेटा) और उपकरणों (अमर कैंसर कोशिका रेखाएं, ट्यूमरजेनिसिस के कृंतकों पर पशु परीक्षण, ज़ेनोग्राफ़्ट मॉडल, उच्च-थ्रूपुट अनुक्रमण विधियों) के साथ काम करता है। , siRNA-आधारित जीन उच्च-थ्रूपुट स्क्रीनिंग को विफल कर रहा है, दैहिक उत्परिवर्तन और जीनोम अस्थिरता के परिणामों की कम्प्यूटेशनल मॉडलिंग)।[32] कैंसर के प्रणाली जीवविज्ञान का दीर्घकालिक उद्देश्य कैंसर का उत्तम निदान करने, इसे वर्गीकृत करने और सुझाए गए उपचार के परिणाम की उत्तम पूर्वानुमान करने की क्षमता है, जो व्यक्तिगत चिकित्सा या कैंसर प्रबंधन और अधिक दूर की संभावनाओं में वर्चुअल फिजियोलॉजिकल ह्यूमन का आधार है। कैंसर के कम्प्यूटेशनल प्रणाली जीव विज्ञान में विभिन्न ट्यूमर के यथार्थवादी बहु-स्तरीय सिलिको मॉडल बनाने में महत्वपूर्ण प्रयास किए गए हैं।[33]
प्रणाली जीव विज्ञान दृष्टिकोण में अधिकांशतः यांत्रिक(जीव विज्ञान) मॉडल का विकास सम्मिलित होता है, जैसे कि उनके प्राथमिक भवन ब्लॉकों के मात्रात्मक गुणों से गतिशील प्रणालियों का पुनर्निर्माण[34][35][36][37] उदाहरण के लिए, सेलुलर नेटवर्क को रासायनिक गतिकी से आने वाली विधियों का उपयोग करके गणितीय रूप से मॉडल किया जा सकता है[38] और नियंत्रण सिद्धांत. सेलुलर नेटवर्क में बड़ी संख्या में मापदंडों, चर और बाधाओं के कारण, संख्यात्मक और कम्प्यूटेशनल विधियो का अधिकांशतः उपयोग किया जाता है (उदाहरण के लिए, फ्लक्स संतुलन विश्लेषण)।[36][38]
जैव सूचना विज्ञान और डेटा विश्लेषण
प्रणाली जीवविज्ञान में कंप्यूटर विज्ञान, सूचना विज्ञान और सांख्यिकी के अन्य पहलुओं का भी उपयोग किया जाता है। इनमें कम्प्यूटेशनल मॉडल के नए रूप सम्मिलित हैं, जैसे कि जैविक प्रक्रियाओं को मॉडल करने के लिए प्रक्रिया कैलकुली का उपयोग (उल्लेखनीय दृष्टिकोण में स्टोकेस्टिक π-कैलकुलस, बायोएम्बिएंट्स, बीटा बाइंडर्स, बायोपीईपीए और ब्रैन कैलकुलस सम्मिलित हैं) और बाधा प्रोग्रामिंग-आधारित मॉडलिंग; सूचना निष्कर्षण और टेक्स्ट मिनींग की विधियो का उपयोग करके साहित्य से जानकारी का एकीकरण;[39] डेटा और मॉडल साझा करने के लिए ऑनलाइन डेटाबेस और रिपॉजिटरी का विकास, सॉफ्टवेयर, वेबसाइटों और डेटाबेस, या वाणिज्यिक सूट के लूज़ युग्मन के माध्यम से डेटाबेस एकीकरण और सॉफ्टवेयर इंटरऑपरेबिलिटी के दृष्टिकोण; उच्च आयामी जीनोमिक डेटा समूह का विश्लेषण करने के लिए नेटवर्क-आधारित दृष्टिकोण उदाहरण के लिए, भारित सहसंबंध नेटवर्क विश्लेषण का उपयोग अधिकांशतः समूहों (मॉड्यूल के रूप में संदर्भित) की पहचान करने, समूहों के बीच संबंधों को मॉडलिंग करने, क्लस्टर (मॉड्यूल) सदस्यता के अस्पष्ट उपायों की गणना करने, इंट्रामॉड्यूलर हब की पहचान करने और अन्य डेटा सेटों में क्लस्टर संरक्षण का अध्ययन करने के लिए किया जाता है; ओमिक्स डेटा विश्लेषण के लिए मार्ग-आधारित विधियाँ, उदा. उनके जीन, प्रोटीन, या मेटाबोलाइट सदस्यों की विभेदक गतिविधि के साथ मार्गों की पहचान करने और स्कोर करने के लिए दृष्टिकोण[40] जीनोमिक डेटा समूह के अधिकांश विश्लेषण में सहसंबंधों की पहचान करना भी सम्मिलित है। इसके अतिरिक्त चूंकि अधिकांश जानकारी विभिन्न क्षेत्रों से आती है, इसलिए जैविक मॉडल का प्रतिनिधित्व करने के वाक्यात्मक और शब्दार्थिक रूप से ध्वनि विधियो के विकास की आवश्यकता है।[41]
जैविक मॉडल बनाना
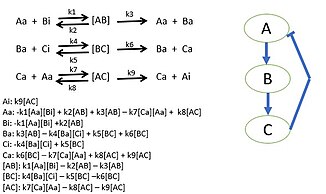
शोधकर्ता जैविक मार्ग चुनकर और सभी प्रोटीन अंतःक्रियाओं का आरेख बनाकर प्रारंभिक करते हैं। प्रोटीन की सभी अंतःक्रियाओं को निर्धारित करने के बाद प्रणाली में प्रतिक्रियाओं की गति का वर्णन करने के लिए मास एक्शन कैनेटीक्स का उपयोग किया जाता है। मास एक्शन कैनेटीक्स जैविक प्रणाली को गणितीय मॉडल के रूप में मॉडल करने के लिए डिफरेंशियल समीकरण प्रदान करेगा जिसमें प्रयोग डिफरेंशियल समीकरणों में उपयोग करने के लिए पैरामीटर मान निर्धारित कर सकते हैं।[43] ये पैरामीटर मान प्रणाली में प्रत्येक प्रोटीन इंटरैक्शन की प्रतिक्रिया दर होंगे। यह मॉडल जैविक प्रणालियों में कुछ प्रोटीनों के व्यवहार को निर्धारित करता है और व्यक्तिगत प्रोटीनों की विशिष्ट गतिविधियों के बारे में नई जानकारी लाता है। कभी-कभी किसी प्रणाली की सभी प्रतिक्रिया दरें एकत्र करना संभव नहीं होता है। अज्ञात प्रतिक्रिया दरें ज्ञात मापदंडों और लक्ष्य व्यवहार के मॉडल का अनुकरण करके निर्धारित की जाती हैं जो संभावित पैरामीटर मान प्रदान करता है।[44][42]
जीनोम-स्केल मॉडल का उपयोग करके उपापचय फेनोटाइप का अनुकरण और पूर्वानुमान करने के लिए प्रणाली जीवविज्ञानियों के बीच बाधा-आधारित पुनर्निर्माण और विश्लेषण (सीओबीआरए) विधियों का उपयोग लोकप्रिय हो गया है। विधियों में से फ्लक्स बैलेंस विश्लेषण (एफबीए) दृष्टिकोण है, जिसके द्वारा कोई जैव रासायनिक नेटवर्क का अध्ययन कर सकता है और रुचि की वस्तु को अधिकतम करके विशेष उपापचय नेटवर्क के माध्यम से मेटाबोलाइट्स के प्रवाह का विश्लेषण कर सकता है।[45]
धियों का उपयोग लोकप्रिय हो गया है। विधियों में से फ्लक्स बैलेंस विश्लेषण (एफबीए) दृष्टिकोण है, जिसके द्वारा कोई जैव रासायनिक नेटवर्क का अध्ययन कर सकता है और रुचि की व
यह भी देखें
- जैविक गणना
- बायोसिस्टम्स|बायोसिस्टम्स (पत्रिका)
- कम्प्यूटेशनल बायोलॉजी
- एक्सपोज़ोम
- इंटरैक्टोम
- जीव विज्ञान में ओमिक्स विषयों की सूची
- जीवित प्रणालियाँ
- मेटाबोलिक नेटवर्क मॉडलिंग
- जैविक प्रणालियों की मॉडलिंग
- आणविक रोगविज्ञान महामारी विज्ञान
- नेटवर्क जीव विज्ञान
- नेटवर्क चिकित्सा
- नोजेनेसिस
- संश्लेषित जीव विज्ञान
- सिस्टम बायोमेडिसिन
- सिस्टम इम्यूनोलॉजी
- सिस्टम मेडिसिन
- टिअरा (डेटाबेस)
संदर्भ
- ↑ <रेफ नाम = तवासोली 487-500 >Tavassoly, Iman; Goldfarb, Joseph; Iyengar, Ravi (2018-10-04). "सिस्टम बायोलॉजी प्राइमर: बुनियादी तरीके और दृष्टिकोण". Essays in Biochemistry. 62 (4): 487–500. doi:10.1042/EBC20180003. ISSN 0071-1365. PMID 30287586. S2CID 52922135.<nowiki>
- ↑ रेफरी>Zewail, Ahmed (2008). भौतिक जीवविज्ञान: परमाणुओं से औषधि तक. Imperial College Press. p. 339.<nowiki>
- ↑ <संदर्भ नाम = तवासोली 487-500 /> रेफरी>Longo, Giuseppe; Montévil, Maël (2014). जीवों पर परिप्रेक्ष्य - स्प्रिंगर. Lecture Notes in Morphogenesis. doi:10.1007/978-3-642-35938-5. ISBN 978-3-642-35937-8. S2CID 27653540.<nowiki>
- ↑ Cite error: Invalid
<ref>tag; no text was provided for refs namedTavassoli - ↑ रेफरी नाम = pmid21570668 >Bu Z, Callaway DJ (2011). "Proteins MOVE! Protein dynamics and long-range allostery in cell signaling". प्रोटीन संरचना और रोग. Advances in Protein Chemistry and Structural Biology. Vol. 83. pp. 163–221. doi:10.1016/B978-0-12-381262-9.00005-7. ISBN 978-0-123-81262-9. PMID 21570668.<nowiki>
- ↑ Snoep, Jacky L; Westerhoff, Hans V (2005). "From isolation to integration, a systems biology approach for building the Silicon Cell". In Alberghina, Lilia; Westerhoff, Hans V (eds.). Systems Biology: Definitions and Perspectives. Topics in Current Genetics. Vol. 13. Berlin: Springer-Verlag. pp. 13–30. doi:10.1007/b106456. ISBN 978-3-540-22968-1.
- ↑ "Systems Biology: the 21st Century Science". Institute for Systems Biology. Retrieved 15 June 2011.
- ↑ 8.0 8.1 Noble, Denis (2006). The music of life: Biology beyond the genome. Oxford: Oxford University Press. p. 176. ISBN 978-0-19-929573-9.
- ↑ Sauer, Uwe; Heinemann, Matthias; Zamboni, Nicola (27 April 2007). "Genetics: Getting Closer to the Whole Picture". Science. 316 (5824): 550–551. doi:10.1126/science.1142502. PMID 17463274. S2CID 42448991.
- ↑ Kholodenko, Boris N; Sauro, Herbert M (2005). "Mechanistic and modular approaches to modeling and inference of cellular regulatory networks". In Alberghina, Lilia; Westerhoff, Hans V (eds.). Systems Biology: Definitions and Perspectives. Topics in Current Genetics. Vol. 13. Berlin: Springer-Verlag. pp. 357–451. doi:10.1007/b136809. ISBN 978-3-540-22968-1.
- ↑ Chiara Romualdi; Gerolamo Lanfranchi (2009). "Statistical Tools for Gene Expression Analysis and Systems Biology and Related Web Resources". In Stephen Krawetz (ed.). Bioinformatics for Systems Biology (2nd ed.). Humana Press. pp. 181–205. doi:10.1007/978-1-59745-440-7_11. ISBN 978-1-59745-440-7.
- ↑ Voit, Eberhard (2012). सिस्टम बायोलॉजी में पहला कोर्स. Garland Science. ISBN 9780815344674.
- ↑ Baitaluk, M. (2009). "System Biology of Gene Regulation". बायोमेडिकल सूचना विज्ञान. Methods in Molecular Biology. Vol. 569. pp. 55–87. doi:10.1007/978-1-59745-524-4_4. ISBN 978-1-934115-63-3. PMID 19623486.
- ↑ 14.0 14.1 14.2 Kling, Jim (3 March 2006). "Working the Systems". Science. Retrieved 15 June 2011.
- ↑ "एचएमएस ने सिस्टम बायोलॉजी का अध्ययन करने के लिए नया विभाग लॉन्च किया". Harvard Gazette. September 23, 2003.
- ↑ Karr, Jonathan R.; Sanghvi, Jayodita C.; Macklin, Derek N.; Gutschow, Miriam V.; Jacobs, Jared M.; Bolival, Benjamin; Assad-Garcia, Nacyra; Glass, John I.; Covert, Markus W. (July 2012). "एक संपूर्ण-कोशिका कम्प्यूटेशनल मॉडल जीनोटाइप से फेनोटाइप की भविष्यवाणी करता है". Cell. 150 (2): 389–401. doi:10.1016/j.cell.2012.05.044. PMC 3413483. PMID 22817898.
- ↑ Mesarovic, Mihajlo D. (1968). Systems Theory and Biology. Berlin: Springer-Verlag.
- ↑ 18.0 18.1 Rosen, Robert (5 July 1968). "A Means Toward a New Holism". Science. 161 (3836): 34–35. Bibcode:1968Sci...161...34M. doi:10.1126/science.161.3836.34. JSTOR 1724368.
- ↑ Kacser, H; Burns, JA (1973). "The control of flux". Symposia of the Society for Experimental Biology. 27: 65–104. PMID 4148886.
- ↑ Kacser, H; Burns, JA; Fell, DA (1995). "The control of flux". Biochemical Society Transactions. 23 (2): 341–366. doi:10.1042/bst0230341. PMID 7672373.
- ↑ Heinrich, R; Rapoport, TA (1974). "A linear steady-state theory of enzymatic chains: general properties, control and effector strength". European Journal of Biochemistry. 42 (1): 89–95. doi:10.1111/j.1432-1033.1974.tb03318.x. PMID 4830198.
- ↑ Savageau, Michael A. (December 1969). "सैद्धांतिक जीवविज्ञान जर्नल". 25 (3): 365–369. doi:10.1016/S0022-5193(69)80026-3. PMID 5387046.
{{cite journal}}: Cite journal requires|journal=(help) - ↑ Savageau, Michael A. (December 1969). "सैद्धांतिक जीवविज्ञान जर्नल". 25 (3): 370–379. doi:10.1016/S0022-5193(69)80027-5. PMID 5387047.
{{cite journal}}: Cite journal requires|journal=(help) - ↑ Savageau, Michael A. (February 1970). "सैद्धांतिक जीवविज्ञान जर्नल". 26 (2): 215–226. doi:10.1016/S0022-5193(70)80013-3. PMID 5434343.
{{cite journal}}: Cite journal requires|journal=(help) - ↑ "सिस्टम बायोलॉजी - राष्ट्रीय सामान्य चिकित्सा विज्ञान संस्थान". Archived from the original on 19 October 2013. Retrieved 12 December 2012.
- ↑ Hunter, Philip (May 2012). "Back down to Earth: Even if it has not yet lived up to its promises, systems biology has now matured and is about to deliver its first results". EMBO Reports. 13 (5): 408–411. doi:10.1038/embor.2012.49. PMC 3343359. PMID 22491028.
- ↑ 27.0 27.1 Cascante, Marta; Marin, Silvia (2008-09-30). "मेटाबोलॉमिक्स और फ्लक्सोमिक्स दृष्टिकोण". Essays in Biochemistry (in English). 45: 67–82. doi:10.1042/bse0450067. ISSN 0071-1365. PMID 18793124.
- ↑ Cusick, Michael E.; Klitgord, Niels; Vidal, Marc; Hill, David E. (2005-10-15). "Interactome: gateway into systems biology". Human Molecular Genetics (in English). 14 (suppl_2): R171–R181. doi:10.1093/hmg/ddi335. ISSN 0964-6906. PMID 16162640.
- ↑ Aur, Dorian (2012). "न्यूरोइलेक्ट्रोडायनामिक्स से लेकर थिंकिंग मशीन तक". Cognitive Computation (in English). 4 (1): 4–12. doi:10.1007/s12559-011-9106-3. ISSN 1866-9956. S2CID 12355069.
- ↑ Loor, Khuram Shahzad and Juan J. (2012-07-31). "रोमिनेंट फिजियोलॉजी और मेटाबॉलिज्म में टॉप-डाउन और बॉटम-अप सिस्टम दृष्टिकोण का अनुप्रयोग". Current Genomics (in English). 13 (5): 379–394. doi:10.2174/138920212801619269. PMC 3401895. PMID 23372424.
- ↑ Spill, Fabian; Bakal, Chris; Mak, Michael (2018). "कैंसर की मैकेनिकल और सिस्टम बायोलॉजी". Computational and Structural Biotechnology Journal. 16: 237–245. arXiv:1807.08990. Bibcode:2018arXiv180708990S. doi:10.1016/j.csbj.2018.07.002. PMC 6077126. PMID 30105089.
- ↑ Barillot, Emmanuel; Calzone, Laurence; Hupe, Philippe; Vert, Jean-Philippe; Zinovyev, Andrei (2012). कैंसर की कम्प्यूटेशनल सिस्टम जीवविज्ञान. Chapman & Hall/CRCMathematical & Computational Biology. p. 461. ISBN 978-1439831441.
- ↑ Byrne, Helen M. (2010). "Dissecting cancer through mathematics: from the cell to the animal model". Nature Reviews Cancer. 10 (3): 221–230. doi:10.1038/nrc2808. PMID 20179714. S2CID 24616792.
- ↑ Gardner, Timothy .S; di Bernardo, Diego; Lorenz, David; Collins, James J. (4 July 2003). "Inferring Genetic Networks and Identifying Compound Mode of Action via Expression Profiling". Science. 301 (5629): 102–105. Bibcode:2003Sci...301..102G. doi:10.1126/science.1081900. PMID 12843395. S2CID 8356492.
- ↑ di Bernardo, Diego; Thompson, Michael J.; Gardner, Timothy S.; Chobot, Sarah E.; Eastwood, Erin L.; Wojtovich, Andrew P.; Elliott, Sean J.; Schaus, Scott E.; Collins, James J. (March 2005). "Chemogenomic profiling on a genome-wide scale using reverse-engineered gene networks". Nature Biotechnology. 23 (3): 377–383. doi:10.1038/nbt1075. PMID 15765094. S2CID 16270018.
- ↑ 36.0 36.1 Tavassoly, Iman (2015). कैंसर कोशिकाओं में ऑटोफैगी और एपोप्टोसिस की परस्पर क्रिया द्वारा मध्यस्थ कोशिका भाग्य निर्णय की गतिशीलता. Springer Theses. Springer International Publishing. doi:10.1007/978-3-319-14962-2. ISBN 978-3-319-14961-5. S2CID 89307028.
- ↑ Korkut, A; Wang, W; Demir, E; Aksoy, BA; Jing, X; Molinelli, EJ; Babur, Ö; Bemis, DL; Onur Sumer, S; Solit, DB; Pratilas, CA; Sander, C (18 August 2015). "पर्टर्बेशन बायोलॉजी आरएएफ अवरोधक प्रतिरोधी मेलेनोमा कोशिकाओं में अपस्ट्रीम-डाउनस्ट्रीम दवा संयोजनों को नामांकित करती है।". eLife. 4. doi:10.7554/eLife.04640. PMC 4539601. PMID 26284497.
- ↑ 38.0 38.1 Gupta, Ankur; Rawlings, James B. (April 2014). "Comparison of Parameter Estimation Methods in Stochastic Chemical Kinetic Models: Examples in Systems Biology". AIChE Journal. 60 (4): 1253–1268. doi:10.1002/aic.14409. ISSN 0001-1541. PMC 4946376. PMID 27429455.
- ↑ Ananadou, Sophia; Kell, Douglas; Tsujii, Jun-ichi (December 2006). "टेक्स्ट माइनिंग और सिस्टम बायोलॉजी में इसके संभावित अनुप्रयोग". Trends in Biotechnology. 24 (12): 571–579. doi:10.1016/j.tibtech.2006.10.002. PMID 17045684.
- ↑ Glaab, Enrico; Schneider, Reinhard (2012). "PathVar: analysis of gene and protein expression variance in cellular pathways using microarray data". Bioinformatics. 28 (3): 446–447. doi:10.1093/bioinformatics/btr656. PMC 3268235. PMID 22123829.
- ↑ Bardini, R.; Politano, G.; Benso, A.; Di Carlo, S. (2017-01-01). "सिस्टम बायोलॉजी के लिए बहु-स्तरीय और हाइब्रिड मॉडलिंग दृष्टिकोण". Computational and Structural Biotechnology Journal. 15: 396–402. doi:10.1016/j.csbj.2017.07.005. ISSN 2001-0370. PMC 5565741. PMID 28855977.
- ↑ 42.0 42.1 Transtrum, Mark K.; Qiu, Peng (2016-05-17). "जटिल जैविक प्रणालियों के ब्रिजिंग मैकेनिस्टिक और फेनोमेनोलॉजिकल मॉडल". PLOS Computational Biology. 12 (5): e1004915. arXiv:1509.06278. Bibcode:2016PLSCB..12E4915T. doi:10.1371/journal.pcbi.1004915. ISSN 1553-7358. PMC 4871498. PMID 27187545.
- ↑ Chellaboina, V.; Bhat, S. P.; Haddad, W. M.; Bernstein, D. S. (August 2009). "मास-एक्शन कैनेटीक्स की मॉडलिंग और विश्लेषण". IEEE Control Systems Magazine. 29 (4): 60–78. doi:10.1109/MCS.2009.932926. ISSN 1941-000X. S2CID 12122032.
- ↑ Brown, Kevin S.; Sethna, James P. (2003-08-12). "कई कम ज्ञात मापदंडों वाले मॉडलों के लिए सांख्यिकीय यांत्रिक दृष्टिकोण". Physical Review E. 68 (2): 021904. Bibcode:2003PhRvE..68b1904B. doi:10.1103/physreve.68.021904. ISSN 1063-651X. PMID 14525003.
- ↑ Orth, Jeffrey D; Thiele, Ines; Palsson, Bernhard Ø (March 2010). "What is flux balance analysis?". Nature Biotechnology (in English). 28 (3): 245–248. doi:10.1038/nbt.1614. ISSN 1087-0156. PMC 3108565. PMID 20212490.
अग्रिम पठन
- Klipp, Edda; Liebermeister, Wolfram; Wierling, Christoph; Kowald, Axel (2016). Systems Biology - A Textbook, 2nd edition. Wiley. ISBN 978-3-527-33636-4.
- Asfar S. Azmi, ed. (2012). Systems Biology in Cancer Research and Drug Discovery. ISBN 978-94-007-4819-4.
- Kitano, Hiroaki (15 October 2001). Foundations of Systems Biology. MIT Press. ISBN 978-0-262-11266-6.
- Werner, Eric (29 March 2007). "All systems go". Nature. 446 (7135): 493–494. Bibcode:2007Natur.446..493W. doi:10.1038/446493a. provides a comparative review of three books:
- Alon, Uri (7 July 2006). An Introduction to Systems Biology: Design Principles of Biological Circuits. Chapman & Hall. ISBN 978-1-58488-642-6.
- Kaneko, Kunihiko (15 September 2006). Life: An Introduction to Complex Systems Biology. Springer-Verlag. Bibcode:2006lics.book.....K. ISBN 978-3-540-32666-3.
- Palsson, Bernhard O. (16 January 2006). Systems Biology: Properties of Reconstructed Networks. Cambridge University Press. ISBN 978-0-521-85903-5.
- Werner Dubitzky; Olaf Wolkenhauer; Hiroki Yokota; Kwan-Hyun Cho, eds. (13 August 2013). Encyclopedia of Systems Biology. Springer-Verlag. ISBN 978-1-4419-9864-4.
बाहरी संबंध
 Media related to Systems biology at Wikimedia Commons
Media related to Systems biology at Wikimedia Commons- Biological Systems in bio-physics-wiki

