वर्चुअल स्क्रीनिंग: Difference between revisions
No edit summary |
m (6 revisions imported from alpha:वर्चुअल_स्क्रीनिंग) |
||
| (One intermediate revision by one other user not shown) | |||
| Line 97: | Line 97: | ||
[[Category: Machine Translated Page]] | [[Category: Machine Translated Page]] | ||
[[Category:Created On 01/12/2023]] | [[Category:Created On 01/12/2023]] | ||
[[Category:Vigyan Ready]] | |||
Latest revision as of 14:47, 14 December 2023
वर्चुअल स्क्रीनिंग (वीएस) एक कम्प्यूटेशनल विधि है जिसका उपयोग दवा की खोज में छोटे अणुओं के लाइब्रेरीज की खोज करने के लिए किया जाता है जिससे कि उन संरचनाओं की पहचान की जा सके जो ड्रग टारगेट, सामान्यतः प्रोटीन रिसेप्टर (जैव रसायन) या एंजाइम से जुड़ने की सबसे अधिक संभावना होती है।[2][3]
वर्चुअल स्क्रीनिंग को कंप्यूटर प्रोग्राम का उपयोग करके यौगिकों के बहुत बड़े लाइब्रेरीज के स्वचालित मूल्यांकन के रूप में परिभाषित किया गया है।[4] जैसा कि इस परिभाषा से पता चलता है, वीएस अधिक सीमा तक एक संख्या का खेल है जो इस बात पर ध्यान केंद्रित करता है कि 1060 से अधिक का विशाल रासायनिक स्थान को कैसे एक प्रबंधनीय संख्या में फ़िल्टर किया जा सकता है जिसे संश्लेषित, खरीद और परीक्षण किया जा सकता है। यद्यपि पूरे रासायनिक ब्रह्मांड की खोज एक सैद्धांतिक रूप से रोचक समस्या हो सकती है, अधिक व्यावहारिक वीएस परिदृश्य लक्षित संयोजन लाइब्रेरीज को डिजाइन और अनुकूलित करने और इन-हाउस कंपाउंड रिपॉजिटरी या विक्रेता प्रसाद से उपलब्ध यौगिकों के लाइब्रेरीज को समृद्ध करने पर ध्यान केंद्रित करते हैं। जैसे-जैसे विधि की स्पष्टता बढ़ी है, वर्चुअल स्क्रीनिंग दवा खोज प्रक्रिया का एक अभिन्न अंग बन गई है।[5][1] वर्चुअल स्क्रीनिंग का उपयोग स्क्रीनिंग के लिए घरेलू डेटाबेस यौगिकों का चयन करने, बाहरी रूप से खरीदे जा सकने वाले यौगिकों को चुनने और यह चुनने के लिए किया जा सकता है कि आगे किस यौगिक को संश्लेषित किया जाना चाहिए।
विधियाँ
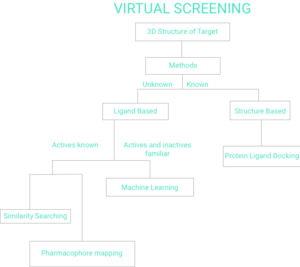
स्क्रीनिंग विधियों की दो व्यापक श्रेणियां हैं: लिगैंड-आधारित और संरचना-आधारित।[6] इस पृष्ठ का शेष भाग वर्चुअल स्क्रीनिंग के चित्र 1 फ़्लो चार्ट को प्रतिबिंबित करेगा।
लिगैंड-आधारित विधियाँ
संरचनात्मक रूप से विविध लिगैंड (जैव रसायन) के एक समूह को देखते हुए, जो एक रिसेप्टर (जैव रसायन) से जुड़ता है, लिगैंड के ऐसे समूह में निहित सामूहिक जानकारी का उपयोग करके रिसेप्टर का एक मॉडल बनाया जा सकता है। विभिन्न कम्प्यूटेशनल विधियाँ अलग-अलग लिगेंड की संरचनात्मक, इलेक्ट्रॉनिक, आणविक आकार और भौतिक रासायनिक समानता का पता लगाती हैं जो एक विशिष्ट आणविक रिसेप्टर या सेल लाइनों के विरुद्ध उनकी कार्रवाई का तरीका बता सकती हैं।[7] एक उम्मीदवार लिगैंड की तुलना फार्माकोफोर मॉडल से की जा सकती है जिससे कि यह निर्धारित किया जा सके कि यह इसके साथ संगत है और इसलिए बाध्य होने की संभावना है।[8] विभिन्न 2डी रासायनिक समानता विश्लेषण विधियां[9] का उपयोग सक्रिय लिगेंड्स को खोजने के लिए डेटाबेस को स्कैन करने के लिए किया गया है। लिगैंड-आधारित वर्चुअल स्क्रीनिंग में उपयोग किया जाने वाला एक अन्य लोकप्रिय दृष्टिकोण ज्ञात सक्रिय पदार्थों के समान आकार वाले अणुओं की खोज करना है, क्योंकि ऐसे अणु लक्ष्य की बाइंडिंग साइट पर फिट होंगे और इसलिए लक्ष्य को बांधने की संभावना होगी। साहित्य में इस वर्ग की विधियों के कई संभावित अनुप्रयोग हैं।[10][11][12] इन 3डी विधियों के फार्माकोफोरिक एक्सटेंशन वेबसर्वर के रूप में भी निःशुल्क उपलब्ध हैं।[13][14] इसके अतिरिक्त आकार आधारित वर्चुअल स्क्रीनिंग ने भी अधिक लोकप्रियता हासिल की है।[15]
संरचना-आधारित विधियाँ
संरचना-आधारित वर्चुअल स्क्रीनिंग दृष्टिकोण में विभिन्न कम्प्यूटेशनल विधियाँ सम्मिलित हैं जो रिसेप्टर की संरचना पर विचार करती हैं जो जांच किए गए सक्रिय लिगैंड का आणविक लक्ष्य है। इनमें से कुछ विधियों में आणविक डॉकिंग (आणविक), संरचना-आधारित फार्माकोफोर पूर्वानुमान और आणविक गतिशीलता सिमुलेशन सम्मिलित हैं।[16][17][7] आणविक डॉकिंग सबसे अधिक उपयोग की जाने वाली संरचना-आधारित विधि है, और यह मैक्रोमोलेक्यूलर रिसेप्टर की बाइंडिंग साइट के विरुद्ध प्रत्येक लिगैंड की फिटनेस का अनुमान लगाने के लिए डॉकिंग के लिए एक अंकन क्रिया प्रयुक्त करती है, जिससे सबसे उच्च संबंध वाले लिगैंड को चुनने में सहायता मिलती है।[18][19][20] वर्तमान में, कुछ वेबसर्वर संभावित वर्चुअल स्क्रीनिंग की ओर उन्मुख हैं।[21][22]
हाइब्रिड विधियाँ
पारंपरिक वीएलएस दृष्टिकोण की सीमाओं को दूर करने के लिए संरचनात्मक और लिगैंड समानता पर भरोसा करने वाली हाइब्रिड विधियां भी विकसित की गईं। यह पद्धति छोटे-अणु बाइंडर्स की पूर्वानुमान करने के लिए विकास-आधारित लिगैंड-बाइंडिंग जानकारी का उपयोग करती है[23][24] और वैश्विक संरचनात्मक समानता और पॉकेट समानता दोनों को नियोजित कर सकता है।[23] एक वैश्विक संरचनात्मक समानता आधारित दृष्टिकोण पीडीबी होलो-टेम्पलेट लाइब्रेरी में प्रोटीन के साथ संरचनात्मक समानता खोजने के लिए एक प्रयोगात्मक संरचना या अनुमानित प्रोटीन मॉडल दोनों को नियोजित करता है। महत्वपूर्ण संरचनात्मक समानता का पता लगाने पर, 2डी फिंगरप्रिंट आधारित टैनिमोटो गुणांक मीट्रिक को छोटे-अणुओं के लिए स्क्रीन पर प्रयुक्त किया जाता है जो चयनित होलो पीडीबी टेम्पलेट्स से निकाले गए लिगैंड के समान होते हैं।[25][26] इस पद्धति से प्राप्त पूर्वानुमानों का प्रयोगात्मक रूप से मूल्यांकन किया गया है और सक्रिय छोटे अणुओं की पहचान करने में अच्छा संवर्धन दिखाया गया है।
उपरोक्त निर्दिष्ट विधि वैश्विक संरचनात्मक समानता पर निर्भर करती है और रुचि के प्रोटीन में किसी विशेष लिगैंड-बाइंडिंग साइट का चयन करने में सक्षम नहीं है। इसके अतिरिक्त, चूंकि विधियां लिगेंड के लिए 2डी समानता मूल्यांकन पर निर्भर करती हैं, इसलिए वे छोटे-अणुओं की स्टीरियोकेमिकल समानता को पहचानने में सक्षम नहीं हैं जो अधिक सीमा तक भिन्न हैं किन्तु ज्यामितीय आकार समानता प्रदर्शित करते हैं। इन चिंताओं को दूर करने के लिए, एक नया पॉकेट केंद्रित दृष्टिकोण, पोली, जो होलो-प्रोटीन टेम्पलेट्स में विशिष्ट बाइंडिंग पॉकेट्स को लक्षित करने में सक्षम है, विकसित किया गया और प्रयोगात्मक रूप से मूल्यांकन किया गया।
कंप्यूटिंग अवसंरचना
परमाणुओं के मध्य युग्मानूसार परस्पर क्रिया की गणना, जो कई आभासी स्क्रीनिंग प्रोग्राम के संचालन के लिए एक नियम है, द्वारा मापी जाती है, N प्रणाली में परमाणुओं की संख्या है। द्विघात स्केलिंग के कारण, कम्प्यूटेशनल व्यय तेजी से बढ़ती है।
लिगैंड-आधारित दृष्टिकोण
लिगैंड-आधारित विधियो को सामान्यतः एकल संरचना तुलना ऑपरेशन के लिए एक सेकंड के एक अंश की आवश्यकता होती है। कभी-कभी एक सीपीयू घंटों के अंदर बड़ी स्क्रीनिंग करने के लिए पर्याप्त होता है। चूँकि, यौगिकों के एक बड़े डेटाबेस के प्रसंस्करण में तेजी लाने के लिए समानांतर में कई तुलनाएँ की जा सकती हैं।
संरचना-आधारित दृष्टिकोण
कार्य के आकार के लिए समानांतर कंप्यूटिंग बुनियादी ढांचे की आवश्यकता होती है, जैसे कि लिनक्स सिस्टम का क्लस्टर, काम को संभालने के लिए बैच क्यू प्रोसेसर चलाना, जैसे सन ग्रिड इंजन या टॉर्क पीबीएस।
बड़े कंपाउंड लाइब्रेरीज से इनपुट को संभालने के साधन की आवश्यकता है। इसके लिए कंपाउंड डेटाबेस के एक रूप की आवश्यकता होती है जिसे समानांतर क्लस्टर द्वारा क्वेरी किया जा सकता है, जो विभिन्न कंप्यूट नोड्स के समानांतर कंपाउंड प्रदान करता है। वाणिज्यिक डेटाबेस इंजन बहुत कठिन हो सकते हैं, और एक उच्च गति अनुक्रमण इंजन, जैसे बर्कले डीबी, एक उत्तम विकल्प हो सकता है। इसके अतिरिक्त, प्रति कार्य एक तुलना चलाना कुशल नहीं हो सकता है, क्योंकि क्लस्टर नोड्स का रैंप अप समय आसानी से उपयोगी कार्य की मात्रा से आगे निकल सकता है। इसके आसपास काम करने के लिए, प्रत्येक क्लस्टर कार्य में यौगिकों के बैचों को संसाधित करना, परिणामों को किसी प्रकार की लॉग फ़ाइल में एकत्रित करना आवश्यक है। लॉग फ़ाइलों को माइन करने और उच्च अंकन अभ्यर्थी को निकालने के लिए एक माध्यमिक प्रक्रिया, पूरे प्रयोग के चलने के बाद चलाई जा सकती है।
स्पष्टता
वर्चुअल स्क्रीनिंग का उद्देश्य नवीन रासायनिक संरचना के अणुओं की पहचान करना है जो मैक्रोमोलेक्युलर जैविक लक्ष्य से जुड़ते हैं। इस प्रकार, वर्चुअल स्क्रीन की सफलता को हिट की कुल संख्या के अतिरिक्त रोचक नए मचान खोजने के संदर्भ में परिभाषित किया गया है। इसलिए, वर्चुअल स्क्रीनिंग स्पष्टता की व्याख्या पर सावधानी से विचार किया जाना चाहिए। रोचक मचानों की कम हिट दर पहले से ही ज्ञात मचानों की उच्च हिट दरों की तुलना में स्पष्ट रूप से उत्तम है।
साहित्य में वर्चुअल स्क्रीनिंग अध्ययन के अधिकांश परीक्षण पूर्वव्यापी हैं। इन अध्ययनों में, वीएस विधि के प्रदर्शन को एक लाइब्रेरी से रुचि के लक्ष्य (सक्रिय अणुओं या सिर्फ सक्रिय) के साथ पहले से ज्ञात अणुओं के एक छोटे समूह को पुनः प्राप्त करने की क्षमता से मापा जाता है, जिसमें अनुमानित निष्क्रिय या बहुत अधिक अनुपात होता है। प्रलोभन। संबंधित सक्रिय अणु के गुणों का मिलान करके डिकॉय का चयन करने के कई अलग-अलग विधियाँ हैं[27] और वर्तमान में डिकॉय को भी गुण-अद्वितीय विधियाँ से चुना गया है।[28] प्रशिक्षण या परीक्षण उद्देश्यों के लिए डिकॉय चयन के वास्तविक प्रभाव पर भी चर्चा की गई है।[28][29]
इसके विपरीत, वर्चुअल स्क्रीनिंग के संभावित अनुप्रयोगों में, परिणामी हिट प्रयोगात्मक पुष्टि के अधीन होते हैं (उदाहरण के लिए, IC50|IC50माप)। इस बात पर आम सहमति है कि पूर्वव्यापी बेंचमार्क संभावित प्रदर्शन के अच्छे भविष्यवक्ता नहीं हैं और परिणामस्वरूप केवल संभावित अध्ययन ही किसी विशेष लक्ष्य के लिए किसी विधि की उपयुक्तता का निर्णायक प्रमाण बनते हैं।[30][31][32][33][34]
दवा खोज के लिए एप्लिकेशन
जब औषधीय रसायन विज्ञान की प्रारंभिक के रूप में हिट अणुओं की पहचान करने की बात आती है तो वर्चुअल स्क्रीनिंग एक बहुत ही उपयोगी एप्लिकेशन है। जैसे-जैसे वर्चुअल स्क्रीनिंग दृष्टिकोण औषधीय रसायन उद्योग के अंदर एक अधिक महत्वपूर्ण और पर्याप्त विधि बनने लगा है, इस दृष्टिकोण में तेजी से वृद्धि हुई है।[35]
लिगैंड-आधारित विधियाँ
संरचना को न जानते हुए भी यह अनुमान लगाने की प्रयास किया जा रहा है कि लिगेंड रिसेप्टर से कैसे बंधेंगे। फार्माकोफोर सुविधाओं के उपयोग से प्रत्येक लिगैंड ने दाता और स्वीकर्ता की पहचान की। समान विशेषताएँ व्याप्त हैं, चूँकि यह देखते हुए कि एक भी सही समाधान होने की संभावना नहीं है।[1]
फार्माकोफोर मॉडल
इस विधि का उपयोग विपरीत संदर्भ यौगिकों, समान वर्णनकर्ताओं और गुणांक, किन्तु विभिन्न सक्रिय यौगिकों का उपयोग करके खोजों के परिणामों को मर्ज करते समय किया जाता है। यह विधि लाभदायक है क्योंकि जब विविध गतिविधियों की बात आती है तो यह सबसे स्पष्ट प्रदर्शन के साथ-साथ एकल संदर्भ संरचना का उपयोग करने की तुलना में अधिक कुशल है।[1]
फार्माकोफोर स्टेरिक और इलेक्ट्रॉनिक विशेषताओं का एक समूह है, जिसकी जैविक प्रतिक्रिया को तेज करने के लिए जैविक लक्ष्य संरचना के साथ इष्टतम सुपरमॉलेक्यूलर इंटरैक्शन या इंटरैक्शन की आवश्यकता होती है। एक्टिविटीज़ के समूह के रूप में एक प्रतिनिधि चुनें, अधिकांश विधियाँ समान बाइंडिंग की तलाश करेंगी।[36] कई कठोर अणुओं को प्राथमिकता दी जाती है और लिगेंड को विविधतापूर्ण होना चाहिए, दूसरे शब्दों में यह सुनिश्चित करना चाहिए कि अलग-अलग विशेषताएं हों जो बंधन चरण के दौरान न हों।[1]
आकार-आधारित वर्चुअल स्क्रीनिंग
आकार-आधारित आणविक समानता दृष्टिकोण को महत्वपूर्ण और लोकप्रिय आभासी स्क्रीनिंग विधियों के रूप में स्थापित किया गया है। वर्तमान में, अत्यधिक अनुकूलित स्क्रीनिंग प्लेटफॉर्म आरओसीएस (रैपिड ओवरले ऑफ केमिकल स्ट्रक्चर्स) को आकार-आधारित, लिगैंड-केंद्रित वर्चुअल स्क्रीनिंग के लिए वास्तविक उद्योग मानक माना जाता है।[37][38][39] यह छोटे कार्बनिक अणुओं के आणविक आयतन को परिभाषित करने के लिए गाऊसी क्रिया का उपयोग करता है। क्वेरी संरचना का चयन कम महत्वपूर्ण है, जो लिगैंड-आधारित मॉडलिंग के लिए आकार-आधारित स्क्रीनिंग को आदर्श बनाता है: चूंकि क्वेरी के लिए बायोएक्टिव संरचना की उपलब्धता स्क्रीनिंग के लिए सीमित कारक नहीं है - यह क्वेरी कंपाउंड का चयन अधिक महत्वपूर्ण है ) जो स्क्रीनिंग प्रदर्शन के लिए निर्णायक है।[15]
क्षेत्र-आधारित वर्चुअल स्क्रीनिंग
आकार-आधारित समानता विधियों में सुधार के रूप में, क्षेत्र-आधारित विधियाँ उन सभी क्षेत्रों को ध्यान में रखने का प्रयास करती हैं जो एक क्वेरी के रूप में उपयोग की जाने वाली रासायनिक संरचना के बारे में अज्ञेयवादी होते हुए लिगैंड-रिसेप्टर इंटरैक्शन को प्रभावित करते हैं। इन विधियों में उपयोग किए जाने वाले अन्य क्षेत्रों के उदाहरण इलेक्ट्रोस्टैटिक या हिड्रोफोबिक क्षेत्र हैं।
मात्रात्मक-संरचना गतिविधि संबंध
मात्रात्मक-संरचना गतिविधि संबंध (क्यूएसएआर) मॉडल में ज्ञात सक्रिय और ज्ञात निष्क्रिय यौगिकों के एक समूह से निकाली गई जानकारी के आधार पर पूर्वानुमानित मॉडल सम्मिलित होते हैं।[40] एसएआर (स्ट्रक्चर एक्टिविटी रिलेशनशिप) जहां डेटा को गुणात्मक रूप से व्यवहार किया जाता है और इसका उपयोग संरचनात्मक वर्गों और एक से अधिक बाइंडिंग मोड के साथ किया जा सकता है। मॉडल लेड की खोज के लिए यौगिकों को प्राथमिकता देते हैं।[1]
मशीन लर्निंग एल्गोरिदम
वर्चुअल स्क्रीनिंग दृष्टिकोण में मशीन लर्निंग एल्गोरिदम का व्यापक रूप से उपयोग किया गया है। पर्यवेक्षित शिक्षण विधियाँ ज्ञात सक्रिय और ज्ञात निष्क्रिय यौगिकों से बने प्रशिक्षण और परीक्षण डेटासमूह का उपयोग करती हैं। विभिन्न एमएल एल्गोरिदम को वर्चुअल स्क्रीनिंग कार्यनीति में सफलता के साथ प्रयुक्त किया गया है, जैसे पुनरावर्ती विभाजन, सपोर्ट वेक्टर मशीन, के-नियेरेस्ट नेबर और न्यूरल नेटवर्क।[41][42][43] ये मॉडल किसी यौगिक के सक्रिय होने की संभावना का पता लगाते हैं और फिर प्रत्येक यौगिक को उसकी संभावना के आधार पर रैंकिंग देते हैं।[1]
मशीन लर्निंग में सबस्ट्रक्चरल विश्लेषण
इस प्रकार यह बड़े डेटासमूह पर उपयोग किया जाने वाला पहला मशीन लर्निंग मॉडल सबस्ट्रक्चर विश्लेषण है जो 1973 में बनाया गया था। प्रत्येक टुकड़ा सबस्ट्रक्चर विशिष्ट प्रकार की गतिविधि में निरंतर योगदान देता है।[1] जब दवा डिजाइन में संरचनाओं का विश्लेषण करने की बात आती है तो सबस्ट्रक्चर एक ऐसी विधि है जो बड़े माप पर आयामीता की कठिनाई को दूर करती है। एक कुशल उपसंरचना विश्लेषण का उपयोग उन संरचनाओं के लिए किया जाता है जिनमें बहु-स्तरीय इमारत या टावर की समानता होती है। ज्यामिति का उपयोग प्रारंभिक में और उत्तम की ओर दी गई संरचना के लिए सीमा जोड़ों को क्रमांकित करने के लिए किया जाता है। जब विशेष स्थैतिक संक्षेपण और प्रतिस्थापन दिनचर्या की विधि विकसित की जाती है तो यह विधि पिछले उपसंरचना विश्लेषण मॉडल की तुलना में अधिक उत्पादक सिद्ध होती है।[44]
पुनरावर्ती विभाजन
पुनरावर्ती विभाजन वह विधि है जो गुणात्मक डेटा का उपयोग करके निर्णय तालिका बनाती है। यह समझना कि किस प्रकार नियम प्रत्येक चरण को दोहराते हुए गलत वर्गीकरण की कम त्रुटि के साथ कक्षाओं को तोड़ते हैं जब तक कि कोई तर्कसंगत विभाजन न मिल जाए। चूँकि, पुनरावर्ती विभाजन में व्यर्थ पूर्वानुमान क्षमता हो सकती है जो संभावित रूप से उसी दर पर अच्छे मॉडल बना सकती है।[1]
संरचना-आधारित विधियाँ ज्ञात प्रोटीन लिगैंड डॉकिंग
प्राथमिकता क्रम निर्दिष्ट करते समय किसी व्यक्तिगत लिगैंड के सबसे संभावित कारण की पहचान करने के लिए डॉकिंग सर्च एल्गोरिदम और अंकन क्रिया का उपयोग करके लिगैंड को प्रोटीन के अंदर एक सक्रिय साइट में बांधा जा सकता है।[1][45]
यह भी देखें
- ग्रिड कंप्यूटिंग
- उच्च परिणाम स्क्रीनिंग
- डॉकिंग (आणविक)
- रेट्रो स्क्रीनिंग
- डॉकिंग के लिए अंकन कार्य
- जिंक डेटाबेस
संदर्भ
- ↑ 1.0 1.1 1.2 1.3 1.4 1.5 1.6 1.7 1.8 1.9 Gillet V (2013). "लिगैंड-आधारित और संरचना-आधारित वर्चुअल स्क्रीनिंग" (PDF). The University of Sheffield.
- ↑ Rester U (July 2008). "From virtuality to reality - Virtual screening in lead discovery and lead optimization: a medicinal chemistry perspective". Current Opinion in Drug Discovery & Development. 11 (4): 559–68. PMID 18600572.
- ↑ Rollinger JM, Stuppner H, Langer T (2008). "Virtual screening for the discovery of bioactive natural products". औषधि के रूप में प्राकृतिक यौगिक खंड I. pp. 211, 213–49. doi:10.1007/978-3-7643-8117-2_6. ISBN 978-3-7643-8098-4. PMC 7124045. PMID 18084917.
{{cite book}}:|journal=ignored (help) - ↑ Walters WP, Stahl MT, Murcko MA (1998). "वर्चुअल स्क्रीनिंग – एक सिंहावलोकन". Drug Discov. Today. 3 (4): 160–178. doi:10.1016/S1359-6446(97)01163-X.
- ↑ McGregor MJ, Luo Z, Jiang X (June 11, 2007). "Chapter 3: Virtual screening in drug discovery". In Huang Z (ed.). ड्रग डिस्कवरी रिसर्च. जीनोमिक युग के बाद के नए मोर्चे. Wiley-VCH: Weinheim, Germany. pp. 63–88. ISBN 978-0-471-67200-5.
- ↑ McInnes C (October 2007). "दवा खोज में वर्चुअल स्क्रीनिंग रणनीतियाँ". Current Opinion in Chemical Biology. 11 (5): 494–502. doi:10.1016/j.cbpa.2007.08.033. PMID 17936059.
- ↑ 7.0 7.1 Santana K, do Nascimento LD, Lima e Lima A, Damasceno V, Nahum C, Braga RC, Lameira J (2021-04-29). "बायोप्रोस्पेक्टिंग में वर्चुअल स्क्रीनिंग के अनुप्रयोग: प्राकृतिक उत्पादों की कीमो-संरचनात्मक विविधता का पता लगाने के लिए तथ्य, बदलाव और परिप्रेक्ष्य". Frontiers in Chemistry. 9: 662688. Bibcode:2021FrCh....9..155S. doi:10.3389/fchem.2021.662688. ISSN 2296-2646. PMC 8117418. PMID 33996755.
- ↑ Sun H (2008). "फार्माकोफोर-आधारित वर्चुअल स्क्रीनिंग". Current Medicinal Chemistry. 15 (10): 1018–24. doi:10.2174/092986708784049630. PMID 18393859.
- ↑ Willet P, Barnard JM, Downs GM (1998). "रासायनिक समानता खोज". Journal of Chemical Information and Computer Sciences. 38 (6): 983–996. CiteSeerX 10.1.1.453.1788. doi:10.1021/ci9800211.
- ↑ Rush TS, Grant JA, Mosyak L, Nicholls A (March 2005). "एक आकार-आधारित 3-डी स्कैफोल्ड हॉपिंग विधि और बैक्टीरिया प्रोटीन-प्रोटीन इंटरैक्शन के लिए इसका अनुप्रयोग". Journal of Medicinal Chemistry. 48 (5): 1489–95. CiteSeerX 10.1.1.455.4728. doi:10.1021/jm040163o. PMID 15743191.
- ↑ Ballester PJ, Westwood I, Laurieri N, Sim E, Richards WG (February 2010). "अल्ट्राफास्ट शेप रिकॉग्निशन के साथ संभावित वर्चुअल स्क्रीनिंग: एरिलैमाइन एन-एसिटाइलट्रांसफेरेज़ के नए अवरोधकों की पहचान". Journal of the Royal Society, Interface. 7 (43): 335–42. doi:10.1098/rsif.2009.0170. PMC 2842611. PMID 19586957.
- ↑ Kumar A, Zhang KY (2018). "आकार समानता विधियों के विकास और औषधि खोज में उनके अनुप्रयोग में प्रगति". Frontiers in Chemistry (in English). 6: 315. Bibcode:2018FrCh....6..315K. doi:10.3389/fchem.2018.00315. PMC 6068280. PMID 30090808.
- ↑ Li H, Leung KS, Wong MH, Ballester PJ (July 2016). "USR-VS: a web server for large-scale prospective virtual screening using ultrafast shape recognition techniques". Nucleic Acids Research (in English). 44 (W1): W436–41. doi:10.1093/nar/gkw320. PMC 4987897. PMID 27106057.
- ↑ Sperandio O, Petitjean M, Tuffery P (July 2009). "wwLigCSRre: a 3D ligand-based server for hit identification and optimization". Nucleic Acids Research. 37 (Web Server issue): W504–9. doi:10.1093/nar/gkp324. PMC 2703967. PMID 19429687.
- ↑ 15.0 15.1 Kirchmair J, Distinto S, Markt P, Schuster D, Spitzer GM, Liedl KR, Wolber G (2009). "How To Optimize Shape-Based Virtual Screening: Choosing the Right Query and Including Chemical Information". Journal of Chemical Information and Modeling. 49 (3): 678–692. doi:10.1021/ci8004226. PMID 19434901.
- ↑ Toledo Warshaviak D, Golan G, Borrelli KW, Zhu K, Kalid O (July 2014). "सहसंयोजक रूप से बंधे लिगेंड्स की खोज के लिए संरचना-आधारित आभासी स्क्रीनिंग दृष्टिकोण". Journal of Chemical Information and Modeling. 54 (7): 1941–50. doi:10.1021/ci500175r. PMID 24932913.
- ↑ Maia EH, Assis LC, de Oliveira TA, da Silva AM, Taranto AG (2020-04-28). "Structure-Based Virtual Screening: From Classical to Artificial Intelligence". Frontiers in Chemistry. 8: 343. Bibcode:2020FrCh....8..343M. doi:10.3389/fchem.2020.00343. PMC 7200080. PMID 32411671.
- ↑ Kroemer RT (August 2007). "Structure-based drug design: docking and scoring". Current Protein & Peptide Science. 8 (4): 312–28. CiteSeerX 10.1.1.225.959. doi:10.2174/138920307781369382. PMID 17696866.
- ↑ Cavasotto CN, Orry AJ (2007). "दवा खोज में लिगैंड डॉकिंग और संरचना-आधारित वर्चुअल स्क्रीनिंग". Current Topics in Medicinal Chemistry. 7 (10): 1006–14. doi:10.2174/156802607780906753. PMID 17508934.
- ↑ Kooistra AJ, Vischer HF, McNaught-Flores D, Leurs R, de Esch IJ, de Graaf C (June 2016). "संयुक्त स्कोरिंग विधि का उपयोग करके जीपीसीआर लिगेंड के लिए फ़ंक्शन-विशिष्ट वर्चुअल स्क्रीनिंग". Scientific Reports. 6: 28288. Bibcode:2016NatSR...628288K. doi:10.1038/srep28288. PMC 4919634. PMID 27339552.
- ↑ Irwin JJ, Shoichet BK, Mysinger MM, Huang N, Colizzi F, Wassam P, Cao Y (September 2009). "Automated docking screens: a feasibility study". Journal of Medicinal Chemistry. 52 (18): 5712–20. doi:10.1021/jm9006966. PMC 2745826. PMID 19719084.
- ↑ Li H, Leung KS, Ballester PJ, Wong MH (2014-01-24). "istar: a web platform for large-scale protein-ligand docking". PLOS ONE. 9 (1): e85678. Bibcode:2014PLoSO...985678L. doi:10.1371/journal.pone.0085678. PMC 3901662. PMID 24475049.
- ↑ 23.0 23.1 Zhou H, Skolnick J (January 2013). "FINDSITE(comb): a threading/structure-based, proteomic-scale virtual ligand screening approach". Journal of Chemical Information and Modeling (in English). 53 (1): 230–40. doi:10.1021/ci300510n. PMC 3557555. PMID 23240691.
- ↑ Roy A, Skolnick J (February 2015). "LIGSIFT: an open-source tool for ligand structural alignment and virtual screening". Bioinformatics. 31 (4): 539–44. doi:10.1093/bioinformatics/btu692. PMC 4325547. PMID 25336501.
- ↑ Gaulton A, Bellis LJ, Bento AP, Chambers J, Davies M, Hersey A, Light Y, McGlinchey S, Michalovich D, Al-Lazikani B, Overington JP (January 2012). "ChEMBL: a large-scale bioactivity database for drug discovery". Nucleic Acids Research. 40 (Database issue): D1100–7. doi:10.1093/nar/gkr777. PMC 3245175. PMID 21948594.
- ↑ Wishart DS, Knox C, Guo AC, Shrivastava S, Hassanali M, Stothard P, Chang Z, Woolsey J (January 2006). "DrugBank: a comprehensive resource for in silico drug discovery and exploration". Nucleic Acids Research. 34 (Database issue): D668–72. doi:10.1093/nar/gkj067. PMC 1347430. PMID 16381955.
- ↑ Réau M, Langenfeld F, Zagury JF, Lagarde N, Montes M (2018). "Decoys Selection in Benchmarking Datasets: Overview and Perspectives". Frontiers in Pharmacology. 9: 11. doi:10.3389/fphar.2018.00011. PMC 5787549. PMID 29416509.
- ↑ 28.0 28.1 Ballester PJ (December 2019). "संरचना-आधारित वर्चुअल स्क्रीनिंग के लिए मशीन-लर्निंग स्कोरिंग फ़ंक्शन का चयन करना". Drug Discovery Today: Technologies. 32–33: 81–87. doi:10.1016/j.ddtec.2020.09.001. PMID 33386098. S2CID 224968364.
- ↑ Li H, Sze KH, Lu G, Ballester PJ (2021). "संरचना-आधारित वर्चुअल स्क्रीनिंग के लिए मशीन-लर्निंग स्कोरिंग फ़ंक्शन". WIREs Computational Molecular Science (in English). 11 (1): e1478. doi:10.1002/wcms.1478. ISSN 1759-0884. S2CID 219089637.
- ↑ Wallach I, Heifets A (2018). "अधिकांश लिगैंड-आधारित वर्गीकरण बेंचमार्क सामान्यीकरण के बजाय याद रखने को पुरस्कृत करते हैं". Journal of Chemical Information and Modeling. 58 (5): 916–932. arXiv:1706.06619. doi:10.1021/acs.jcim.7b00403. PMID 29698607. S2CID 195345933.
- ↑ Irwin JJ (2008). "वर्चुअल स्क्रीनिंग के लिए सामुदायिक बेंचमार्क". Journal of Computer-Aided Molecular Design. 22 (3–4): 193–9. Bibcode:2008JCAMD..22..193I. doi:10.1007/s10822-008-9189-4. PMID 18273555. S2CID 26260725.
- ↑ Good AC, Oprea TI (2008). "सीएएमडी तकनीकों का अनुकूलन 3. वर्चुअल स्क्रीनिंग संवर्धन अध्ययन: उपकरण चयन में मदद या बाधा?". Journal of Computer-Aided Molecular Design. 22 (3–4): 169–78. Bibcode:2008JCAMD..22..169G. doi:10.1007/s10822-007-9167-2. PMID 18188508. S2CID 7738182.
- ↑ Schneider G (April 2010). "वर्चुअल स्क्रीनिंग: एक अंतहीन सीढ़ी?". Nature Reviews. Drug Discovery. 9 (4): 273–6. doi:10.1038/nrd3139. PMID 20357802. S2CID 205477076.
- ↑ Ballester PJ (January 2011). "अल्ट्राफास्ट आकार पहचान: विधि और अनुप्रयोग". Future Medicinal Chemistry. 3 (1): 65–78. doi:10.4155/fmc.10.280. PMID 21428826.
- ↑ Lavecchia A, Di Giovanni C (2013). "Virtual screening strategies in drug discovery: a critical review". Current Medicinal Chemistry. 20 (23): 2839–60. doi:10.2174/09298673113209990001. PMID 23651302.
- ↑ Spitzer GM, Heiss M, Mangold M, Markt P, Kirchmair J, Wolber G, Liedl KR (2010). "One concept, three implementations of 3D pharmacophore-based virtual screening: distinct coverage of chemical search space". Journal of Chemical Information and Modeling. 50 (7): 1241–1247. doi:10.1021/ci100136b. PMID 20583761.
- ↑ Grant JA, Gallard MA, Pickup BT (1996). "A fast method of molecular shape comparison: a simple application of a Gaussian description of molecular shape". Journal of Computational Chemistry. 17 (14): 1653–1666. doi:10.1002/(SICI)1096-987X(19961115)17:14<1653::AID-JCC7>3.0.CO;2-K.
- ↑ Nicholls A, Grant JA (2005). "प्रासंगिक रासायनिक जानकारी के एन्कोडिंग में आणविक आकार और इलेक्ट्रोस्टैटिक्स". Journal of Computer-Aided Molecular Design. 19 (9–10): 661–686. doi:10.1007/s10822-005-9019-x. PMID 16328855.
- ↑ Rush TS, Grant JA, Mosyak L, Nicholls A (2005). "A shape-based 3-D scaffold hopping method and its application to a bacterial protein-protein interaction". Journal of Medicinal Chemistry. 48 (5): 1489–1495. doi:10.1021/jm040163o. PMID 15743191.
- ↑ Neves BJ, Braga RC, Melo-Filho CC, Moreira-Filho JT, Muratov EN, Andrade CH (2018-11-13). "QSAR-Based Virtual Screening: Advances and Applications in Drug Discovery". Frontiers in Pharmacology. 9: 1275. doi:10.3389/fphar.2018.01275. PMC 6262347. PMID 30524275.
- ↑ Alsenan S, Al-Turaiki I, Hafez A (December 2020). "रक्त-मस्तिष्क बाधा पारगम्यता की भविष्यवाणी करने के लिए एक आवर्ती तंत्रिका नेटवर्क मॉडल". Computational Biology and Chemistry. 89: 107377. doi:10.1016/j.compbiolchem.2020.107377. PMID 33010784.
- ↑ Dimitri GM, Lió P (June 2017). "DrugClust: A machine learning approach for drugs side effects prediction". Computational Biology and Chemistry. 68: 204–210. doi:10.1016/j.compbiolchem.2017.03.008. PMID 28391063.
- ↑ Shoombuatong W, Schaduangrat N, Pratiwi R, Nantasenamat C (June 2019). "THPep: A machine learning-based approach for predicting tumor homing peptides". Computational Biology and Chemistry. 80: 441–451. doi:10.1016/j.compbiolchem.2019.05.008. PMID 31151025.
- ↑ Gurujee CS, Deshpande VL (February 1978). "उपसंरचना विश्लेषण की एक बेहतर विधि". Computers & Structures. 8 (1): 147–152. doi:10.1016/0045-7949(78)90171-2.
- ↑ Pradeepkiran JA, Reddy PH (March 2019). "अल्जाइमर रोग में फॉस्फोराइलेटेड ताऊ अवरोधकों के लिए संरचना आधारित डिजाइन और आणविक डॉकिंग अध्ययन". Cells. 8 (3): 260. doi:10.3390/cells8030260. PMC 6468864. PMID 30893872.
अग्रिम पठन
- Melagraki G, Afantitis A, Sarimveis H, Koutentis PA, Markopoulos J, Igglessi-Markopoulou O (May 2007). "Optimization of biaryl piperidine and 4-amino-2-biarylurea MCH1 receptor antagonists using QSAR modeling, classification techniques and virtual screening". Journal of Computer-Aided Molecular Design. 21 (5): 251–67. Bibcode:2007JCAMD..21..251M. doi:10.1007/s10822-007-9112-4. PMID 17377847. S2CID 19563229.
- Afantitis A, Melagraki G, Sarimveis H, Koutentis PA, Markopoulos J, Igglessi-Markopoulou O (February 2006). "Investigation of substituent effect of 1-(3,3-diphenylpropyl)-piperidinyl phenylacetamides on CCR5 binding affinity using QSAR and virtual screening techniques". Journal of Computer-Aided Molecular Design. 20 (2): 83–95. Bibcode:2006JCAMD..20...83A. CiteSeerX 10.1.1.716.8148. doi:10.1007/s10822-006-9038-2. PMID 16783600. S2CID 21523436.
- Eckert H, Bajorath J (March 2007). "Molecular similarity analysis in virtual screening: foundations, limitations and novel approaches". Drug Discovery Today. 12 (5–6): 225–33. doi:10.1016/j.drudis.2007.01.011. PMID 17331887.
- Willett P (December 2006). "Similarity-based virtual screening using 2D fingerprints" (PDF). Drug Discovery Today (Submitted manuscript). 11 (23–24): 1046–53. doi:10.1016/j.drudis.2006.10.005. PMID 17129822.
- Fara DC, Oprea TI, Prossnitz ER, Bologa CG, Edwards BS, Sklar LA (2006). "Integration of virtual and physical screening". Drug Discovery Today: Technologies. 3 (4): 377–385. doi:10.1016/j.ddtec.2006.11.003. PMC 7105924.
- Muegge I, Oloffa S (2006). "Advances in virtual screening". Drug Discovery Today: Technologies. 3 (4): 405–411. doi:10.1016/j.ddtec.2006.12.002. PMC 7105922.
- Schneider G (April 2010). "Virtual screening: an endless staircase?". Nature Reviews. Drug Discovery. 9 (4): 273–6. doi:10.1038/nrd3139. PMID 20357802. S2CID 205477076.
बाहरी संबंध
- VLS3D – list of over 2000 databases, online and standalone in silico tools