विलोपन (आनुवांशिकी): Difference between revisions
No edit summary |
No edit summary |
||
| Line 39: | Line 39: | ||
== परीक्षण == | == परीक्षण == | ||
शास्त्रीय साइटोजेनेटिक विधियों के संयोजन में आणविक प्रौद्योगिकी की प्रारम्भ ने हाल के वर्षों में क्रोमोसोमल असामान्यताओं के लिए नैदानिक क्षमता में अधिक सुधार किया है। विशेष रूप से, बीएसी क्लोन के उपयोग के आधार पर माइक्रोएरे | शास्त्रीय साइटोजेनेटिक विधियों के संयोजन में आणविक प्रौद्योगिकी की प्रारम्भ ने हाल के वर्षों में क्रोमोसोमल असामान्यताओं के लिए नैदानिक क्षमता में अधिक सुधार किया है। विशेष रूप से, बीएसी क्लोन के उपयोग के आधार पर माइक्रोएरे तुलनात्मक जीनोमिक संकरण (सीजीएच) जीनोम वाइड स्तर पर डीएनए कॉपी नंबर परिवर्तनों का पता लगाने के लिए संवेदनशील रणनीति का वादा करता है। पता लगाने का रिज़ॉल्यूशन> 30,000 बैंड जितना अधिक हो सकता है एवं क्रोमोसोमल विलोपन का आकार 5-20 kb जितना छोटा हो सकता है।<ref>{{cite journal|last=Ren|first=H|title=BAC-based PCR fragment microarray: high-resolution detection of chromosomal deletion and duplication breakpoints|journal=Human Mutation|date=May 2005|volume=25|issue=5|pages=476–482|pmid=15832308|doi=10.1002/humu.20164|s2cid=28030180|doi-access=free}}</ref> डीएनए अनुक्रमण विलोपन त्रुटियों जैसे अंत अनुक्रम प्रोफाइलिंग की शोध के लिए अन्य अभिकलन विधियों का चयन किया गया था।<ref>{{cite journal |url=http://www.eng.tau.ac.il/~bengal/VOM_EST.pdf |title=ईएसटी अनुक्रमों में संभावित कोडिंग क्षेत्रों के पुनर्निर्माण के लिए वीओएम मॉडल का उपयोग करना|last1=Shmilovici |first1=A. |last2=Ben-Gal |first2=I. |journal=Journal of Computational Statistics |volume=22 |issue=1 |pages=49–69 |year=2007 |doi=10.1007/s00180-007-0021-8|s2cid=2737235 }}</ref><ref>{{cite journal|last1=Volik|first1=S.|last2=Zhao|first2=S.|last3=Chin|first3=K.|last4=Brebner|first4=J. H.|last5=Herndon|first5=D. R.|last6=Tao|first6=Q.|last7=Kowbel|first7=D.|last8=Huang|first8=G.|last9=Lapuk|first9=A.|last10=Kuo|first10=W.-L.|last11=Magrane|first11=G.|last12=de Jong|first12=P.|last13=Gray|first13=J. W.|last14=Collins|first14=C.|title=End-sequence profiling: Sequence-based analysis of aberrant genomes|journal=Proceedings of the National Academy of Sciences|date=4 June 2003|volume=100|issue=13|pages=7696–7701|doi=10.1073/pnas.1232418100|pmid=12788976|pmc=164650|bibcode=2003PNAS..100.7696V|doi-access=free}}</ref> | ||
Revision as of 21:02, 26 June 2023
आनुवंशिकी में, विलोपन (जिसे जीन विलोपन, कमी या विलोपन उत्परिवर्तन भी कहा जाता है) (संकेत: डेल्टा (अक्षर) | Δ) उत्परिवर्तन (आनुवंशिक विपथन) है जिसमें गुणसूत्र का भाग या डीएनए का क्रम छूट जाता है डीएनए प्रतिकृति के समय। किसी भी संख्या में न्यूक्लियोटाइड्स को एकल न्यूक्लियोबेस से क्रोमोसाम के पूरे टुकड़े तक निकाला जा सकता है।[1] कुछ गुणसूत्रों में नाजुक धब्बे होते हैं जहां टूटना होता है जिसके परिणामस्वरूप गुणसूत्र का भाग नष्ट हो जाता है। ब्रेक गर्मी, वायरस, विकिरण, रसायनों से प्रेरित हो सकते हैं। जब गुणसूत्र टूट जाता है, तो इसका भाग नष्ट हो जाता है या विलुप्त जाता है, गुणसूत्र के लापता भागों को विलोपन या कमी के रूप में संदर्भित किया जाता है।[2]
निष्कर्ष के लिए क्रोमोसोम के मध्य बड़ी अंतरालीय कमी एवं सामान्य पूर्ण होमोलॉग के मध्य होने के लिए, सामान्य होमोलॉग के अयुग्मित क्षेत्र को रैखिक संरचना से विलोपन या क्षतिपूर्ति पाश में लूप करना चाहिए।
डीएनए पोलीमरेज़ सक्रिय साइट के अन्दर टेम्पलेट डीएनए स्ट्रैंड स्लिपेज के पश्चात टेम्प्लेट डीएनए में एकल आधार फ़्लिपिंग द्वारा सबसे छोटा एकल आधार विलोपन म्यूटेशन होता है।[3][4][5]अर्धसूत्रीविभाजन के समय क्रोमोसोमल क्रॉसओवर में त्रुटियों के कारण विलोपन हो सकता है, जो कई गंभीर आनुवंशिक रोगों का कारण बनता है। विलोपन जो तीन आधारों के गुणकों में नहीं होते हैं, आनुवंशिक अनुक्रम के 3-न्यूक्लियोटाइड प्रोटीन रीडिंग फ्रेम को परिवर्तित होकेफ्रेम शिफ्ट मुतसिओन का कारण बन सकते हैं। विलोपन यूकेरियोटिक जीवों के प्रतिनिधि हैं, जिनमें मानव भी सम्मिलित हैं एवं बैक्टीरिया जैसे प्रोकार्योटिक जीवों में नहीं।
कारण
कारणों में निम्नलिखित सम्मिलित हैं:
- क्रोमोसोमल ट्रांसलोकेशन से हानि
- क्रोमोसोमल क्रॉसओवर क्रोमोसोमल व्युत्क्रम के अन्दर
- असमान क्रॉसिंग ओवर
- पुनः जुड़े बिना तोड़ना
प्रकार
विलोपन के प्रकारों में निम्न सम्मिलित हैं:
- टर्मिनल विलोपन - विलोपन जो गुणसूत्र के अंत की ओर होता है।
- अंतरालीय/अंतरालीय विलोपन - विलोपन जो गुणसूत्र के आंतरिक भाग से होता है।
- माइक्रोडिलीशन - विलोपन की अपेक्षाकृत कम मात्रा (5Mb तक जिसमें दर्जन जीन सम्मिलित हो सकते हैं)।
सूक्ष्म विलोपन सामान्यतः शारीरिक असामान्यताओं वाले बच्चों में पाया जाता है। बड़ी मात्रा में विलोपन का परिणाम तत्काल गर्भपात (गर्भपात) होगा।
नामकरण

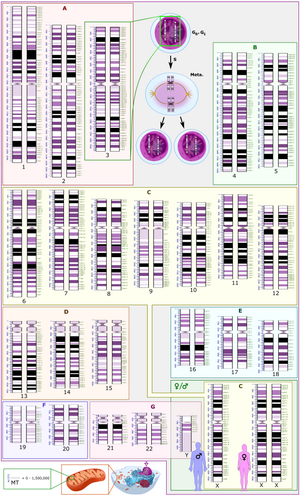
मानव साइटोजेनोमिक नामकरण (आईएससीएन) के लिए अंतर्राष्ट्रीय प्रणाली मानव गुणसूत्र नामकरण के लिए अंतरराष्ट्रीय मानक है, जिसमें मानव गुणसूत्र एवं गुणसूत्र असामान्यताओं के विवरण में उपयोग किए जाने वाले बैंड नाम, प्रतीक एवं संक्षिप्त शब्द सम्मिलित हैं। संक्षिप्ताक्षरों में गुणसूत्र विलोपन के लिए ऋण चिह्न (-) एवं गुणसूत्र के कुछ भागों को हटाने के लिए डेल सम्मिलित हैं।[7]
प्रभाव
छोटे विलोपन के घातक होने की संभावना कम होती है; बड़े विलोपन सामान्यतः घातक होते हैं - हमेशा भिन्नताएं होती हैं जिसके आधार पर जीन खो जाते हैं। कुछ मध्यम आकार के विलोपन पहचानने योग्य मानव विकारों को जन्म देते हैं, उदा। विलियम्स सिंड्रोम।
कई जोड़ों का विलोपन जो तीन से समान रूप से विभाज्य नहीं है, फ्रेमशिफ्ट म्यूटेशन को जन्म देगा, जिसके कारण विलोपन के पश्चात होने वाले सभी कोडन अनुवाद (आनुवांशिकी) के समय गलत तरीके से पढ़े जा सकते हैं, गंभीर रूप से परिवर्तित एवं संभावित रूप से अन्य-कार्यात्मक प्रोटीन का उत्पादन करते हैं। इसके विपरीत, विलोपन जो समान रूप से तीन से विभाज्य है, इन-फ्रेम विलोपन कहलाता है।[8] विलोपन आनुवंशिक विकारों की श्रृंखला के लिए उत्तरदायी हैं, जिनमें पुरुष बांझपन के कुछ विषय, Duchenne पेशी dystrophy के दो तिहाई विषय सम्मिलित हैं,[1]एवं पुटीय तंतुशोथ के दो तिहाई विषय (जो ΔF508 के कारण होते हैं)।[9] क्रोमोसोम 5 की छोटी भुजा के भागों को हटाने से बिल्ली का रोना सिंड्रोम होता है।[1]उत्तरजीविता मोटर न्यूरॉन-एन्कोडिंग जीन में विलोपन के कारण रीढ़ की हड्डी में पेशी शोष होता है, जो शिशु मृत्यु का सबसे आम आनुवंशिक कारण है।
माइक्रोडिलीशन कई अलग-अलग स्थितियों से जुड़े हैं, जिनमें एंजेलमैन सिंड्रोम, प्रेडर-विली सिंड्रोम एवं डिजॉर्ज सिंड्रोम सम्मिलित हैं।[10] एंजेलमैन सिंड्रोम एवं प्रेडर-विली सिंड्रोम सहित कुछ सिंड्रोम, माइक्रोडिलीशन एवं जीनोमिक इंप्रिनटिंग दोनों से जुड़े हुए हैं, जिसका अर्थ है कि ही माइक्रोडिलीशन दो अलग-अलग सिंड्रोम का कारण बन सकता है, जिसके आधार पर विलोपन माता-पिता से होता है।[11] हाल के काम से पता चलता है कि अत्यधिक संरक्षित अनुक्रमों (CONDELs) के कुछ विलोपन निकट संबंधी प्रजातियों के मध्य उपस्थित विकासवादी अंतरों के लिए उत्तरदायी हो सकते हैं। मनुष्यों में इस प्रकार के विलोपन, जिसे hCONDELs कहा जाता है, मनुष्यों, आम चिंपैंजी एवं अन्य प्रकार के स्तनधारियों जैसे बंदर या बंदर|बंदरों के मध्य शारीरिक एवं व्यवहारिक अंतर के लिए उत्तरदायी हो सकते हैं।[12] द कैंसर जीनोम एटलस कॉहोर्ट्स में हाल ही में व्यापक रोगी-स्तरीय वर्गीकरण एवं चालक घटनाओं की मात्रा का पता चला है कि प्रति ट्यूमर औसतन 12 चालक घटनाएं होती हैं, जिनमें से 2.1 ट्यूमर दमन करने वाला जीन के विलोपन हैं।[13]
परीक्षण
शास्त्रीय साइटोजेनेटिक विधियों के संयोजन में आणविक प्रौद्योगिकी की प्रारम्भ ने हाल के वर्षों में क्रोमोसोमल असामान्यताओं के लिए नैदानिक क्षमता में अधिक सुधार किया है। विशेष रूप से, बीएसी क्लोन के उपयोग के आधार पर माइक्रोएरे तुलनात्मक जीनोमिक संकरण (सीजीएच) जीनोम वाइड स्तर पर डीएनए कॉपी नंबर परिवर्तनों का पता लगाने के लिए संवेदनशील रणनीति का वादा करता है। पता लगाने का रिज़ॉल्यूशन> 30,000 बैंड जितना अधिक हो सकता है एवं क्रोमोसोमल विलोपन का आकार 5-20 kb जितना छोटा हो सकता है।[14] डीएनए अनुक्रमण विलोपन त्रुटियों जैसे अंत अनुक्रम प्रोफाइलिंग की शोध के लिए अन्य अभिकलन विधियों का चयन किया गया था।[15][16]
माइटोकॉन्ड्रियल डीएनए विलोपन
यीस्ट सैक्रोमाइसेस सेरेविसिया में, परमाणु जीन Rad51p, Rad52p एवं Rad59p उन प्रोटीनों को एनकोड करते हैं जो पुनर्संयोजन के सुधार के लिए आवश्यक हैं एवं माइटोकॉन्ड्रियल डीएनए में डीएनए क्षति (स्वाभाविक रूप से होने वाली) के सुधार में कार्यरत हैं।[17] इन प्रोटीनों के नष्ट होने से माइटोकॉन्ड्रिया में सहज डीएनए विलोपन की घटनाओं की दर कम हो जाती है।[17] इस शोध का तात्पर्य है कि सजातीय पुनर्संयोजन द्वारा डीएनए डबल-स्ट्रैंड के टूटने का सुधार माइटोकॉन्ड्रियल डीएनए विलोपन के निर्माण में कदम है।
यह भी देखें
- इंडेल
- गुणसूत्र असामान्यताएं
- अशक्त एलील
- आनुवंशिक विकारों की सूची
- चिकित्सा आनुवंशिकी
- माइक्रोडिलीशन सिंड्रोम
- क्रोमोसोमल विलोपन सिंड्रोम
- सम्मिलन (आनुवांशिकी)
संदर्भ
- ↑ 1.0 1.1 1.2 Lewis, R. (2004). Human Genetics: Concepts and Applications (6th ed.). McGraw Hill. ISBN 978-0072951745.
- ↑ Klug, William S. (2015). आनुवंशिकी की अवधारणाएँ. Michael R. Cummings, Charlotte A. Spencer, Michael Angelo Palladino (Eleventh ed.). Boston. ISBN 978-0-321-94891-5. OCLC 880404074.
{{cite book}}: CS1 maint: location missing publisher (link) - ↑ Banavali, Nilesh K. (2013). "डीएनए डुप्लेक्स टर्मिनी के पास स्ट्रैंड स्लिपेज के लिए आंशिक आधार फ़्लिपिंग पर्याप्त है". Journal of the American Chemical Society. 135 (22): 8274–8282. doi:10.1021/ja401573j. PMID 23692220.
- ↑ Banavali, Nilesh K. (2013). "डीएनए डुप्लेक्स टर्मिनी के पास सिंगल बेस फ्लिपिंग और स्ट्रैंड स्लिपेज के बीच संबंध का विश्लेषण". The Journal of Physical Chemistry B. 117 (46): 14320–14328. doi:10.1021/jp408957c. PMID 24206351.
- ↑ Manjari, Swati R.; Pata, Janice D.; Banavali, Nilesh K. (2014). "Cytosine Unstacking and Strand Slippage at an Insertion–Deletion Mutation Sequence in an Overhang-Containing DNA Duplex". Biochemistry. 53 (23): 3807–3816. doi:10.1021/bi500189g. PMC 4063443. PMID 24854722.
- ↑ Warrender JD, Moorman AV, Lord P (2019). "कैरियोटाइप के लिए पूरी तरह से कम्प्यूटेशनल और उचित प्रतिनिधित्व।". Bioinformatics. 35 (24): 5264–5270. doi:10.1093/bioinformatics/btz440. PMC 6954653. PMID 31228194.
{{cite journal}}: CS1 maint: multiple names: authors list (link)
- "This is an Open Access article distributed under the terms of the Creative Commons Attribution License (http://creativecommons.org/licenses/by/4.0/)" - ↑ "आईएससीएन प्रतीक और संक्षिप्त शर्तें". Coriell Institute for Medical Research. Retrieved 2022-10-27.
- ↑ LSDB — Controlled vocabulary terms Archived 2011-10-06 at the Wayback Machine at The GEN2PHEN Knowledge Centre. Posted Fri, 08/01/2010.
- ↑ Mitchell, Richard Sheppard; Kumar, Vinay; Robbins, Stanley L.; Abbas, Abul K.; Fausto, Nelson (2007). रॉबिन्स बेसिक पैथोलॉजी. Saunders/Elsevier. ISBN 978-1-4160-2973-1.
- ↑ Srour, Myriam; Shevell, Michael (2015-01-01), Rosenberg, Roger N.; Pascual, Juan M. (eds.), "Chapter 14 - Global Developmental Delay and Intellectual Disability", Rosenberg's Molecular and Genetic Basis of Neurological and Psychiatric Disease (Fifth Edition) (in English), Boston: Academic Press, pp. 151–161, ISBN 978-0-12-410529-4, retrieved 2022-01-07
- ↑ Kalsner, Louisa; Chamberlain, Stormy J. (April 22, 2015). "Prader-Willi, Angelman, and 15q11-q13 duplication syndromes". Pediatric Clinics of North America. 62 (3): 587–606. doi:10.1016/j.pcl.2015.03.004. ISSN 0031-3955. PMC 4449422. PMID 26022164.
- ↑ McLean CY, Reno PL, Pollen AA, Bassan AI, Capellini TD, Guenther C, Indjeian VB, Lim X, Menke DB, Schaar BT, Wenger AM, Bejerano G, Kingsley DM (March 2011). "नियामक डीएनए की मानव-विशिष्ट हानि और मानव-विशिष्ट लक्षणों का विकास". Nature. 471 (7337): 216–9. Bibcode:2011Natur.471..216M. doi:10.1038/nature09774. PMC 3071156. PMID 21390129.
- ↑ Vyatkin, Alexey D.; Otnyukov, Danila V.; Leonov, Sergey V.; Belikov, Aleksey V. (14 January 2022). "टीसीजीए पैनकैनएटलस कॉहोर्ट्स में व्यापक रोगी-स्तरीय वर्गीकरण और चालक घटनाओं की मात्रा का ठहराव". PLOS Genetics. 18 (1): e1009996. doi:10.1371/journal.pgen.1009996. PMC 8759692. PMID 35030162.
- ↑ Ren, H (May 2005). "BAC-based PCR fragment microarray: high-resolution detection of chromosomal deletion and duplication breakpoints". Human Mutation. 25 (5): 476–482. doi:10.1002/humu.20164. PMID 15832308. S2CID 28030180.
- ↑ Shmilovici, A.; Ben-Gal, I. (2007). "ईएसटी अनुक्रमों में संभावित कोडिंग क्षेत्रों के पुनर्निर्माण के लिए वीओएम मॉडल का उपयोग करना" (PDF). Journal of Computational Statistics. 22 (1): 49–69. doi:10.1007/s00180-007-0021-8. S2CID 2737235.
- ↑ Volik, S.; Zhao, S.; Chin, K.; Brebner, J. H.; Herndon, D. R.; Tao, Q.; Kowbel, D.; Huang, G.; Lapuk, A.; Kuo, W.-L.; Magrane, G.; de Jong, P.; Gray, J. W.; Collins, C. (4 June 2003). "End-sequence profiling: Sequence-based analysis of aberrant genomes". Proceedings of the National Academy of Sciences. 100 (13): 7696–7701. Bibcode:2003PNAS..100.7696V. doi:10.1073/pnas.1232418100. PMC 164650. PMID 12788976.
- ↑ 17.0 17.1 Stein A, Kalifa L, Sia EA. Members of the RAD52 Epistasis Group Contribute to Mitochondrial Homologous Recombination and Double-Strand Break Repair in Saccharomyces cerevisiae. PLoS Genet. 2015 Nov 5;11(11):e1005664. doi:10.1371/journal.pgen.1005664. PMID: 26540
